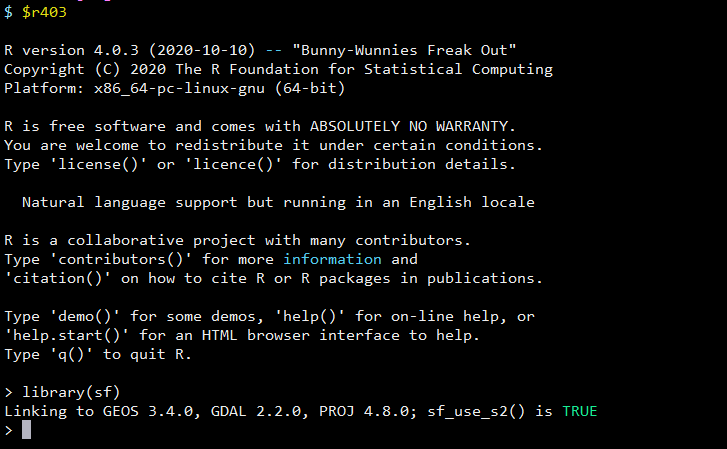
折腾了一天,终于把这个 R 包装上了
事情起因于在推特看到关于ggVennDiagram这个 R 包教程的一条推文,想着去复现一下,于是开始去安装,不料安装过程中出现了sf这个依赖包始终安装不成功的一堆错误,于是有了这一篇文章,特此记录一下,也希望给遇到类似问题的小伙伴一个参考。
第一个错误
首先说明一下,我用的 R 版本是 4.3.0,Linux 系统是 Red Hat 6.5。
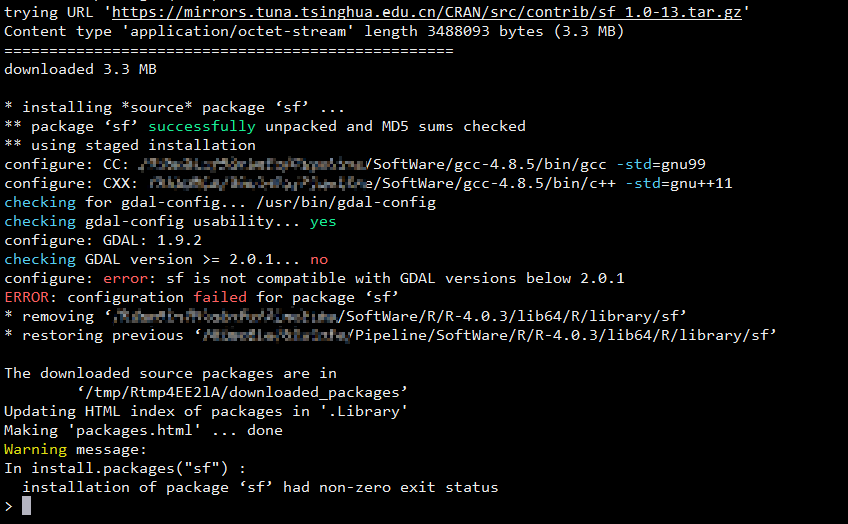
最开始安装sf这个 R 包遇到的第一个 error 是 GDAL/GEOS/Proj.4 版本不符合要求的提示。
于是,开始手动去安装 GDAL/GEOS/Proj.4,之所以选择基于普通用户手动去源码编译安装,主要一个原因是系统版本太老,第二出于安全考虑避免 root 带来的一系列麻烦。
一番折腾,把 gdal-2.2.3+geos-3.4.0+proj-4.9.1 都装好了,设置完 PATH+LD_LIBRARY_PATH 后,却悲催的出现类似这个的报错:https://github.com/r-spatial/sf/issues/678 —— 这是sf包的一个错误!
于是,问题变成了如何成功去安装sf包,或者说如何解决sf的 proj_conf_test.c:4:28: error: expected ')' before 'const' 安装错误。
sf 包依赖与解决
在sf包的 https://github.com/r-spatial/sf#linux 中明确提到了这个包需要依赖 GDAL/GEOS/Proj.4,具体版本要求如下。
📢 For Unix-alikes, GDAL (>= 2.0.1), GEOS (>= 3.4.0) and Proj.4 (>= 4.8.0) are required.
后来经过一番的折腾尝试,才发现:
- gdal-2.2.3+geos-3.4.0+proj-4.9.1 出现类似这个的报错 —— proj_conf_test.c:4:28: error: expected ')' before 'const'!
- gdal-2.2.0+geos-3.4.0+proj-4.8.0 的组合可以解决以上遇到的问题!
- 手动源码安装的话,Proj.4 要先于 GDAL 安装,因为 GDAL 安装的时候需要指定 Proj.4 进行编译。
GEOS
- 要求 GEOS version >= 3.4.0;
export PATH=/Bioinfo/Pipeline/SoftWare/gcc-4.8.5/bin:$PATH
wget https://download.osgeo.org/geos/geos-3.4.0.tar.bz2
tar xvjf geos-3.4.0.tar.bz2 -C ../build/
cd ../build/geos-3.4.0
./configure --prefix=/Bioinfo/Pipeline/SoftWare/geos-3.4.0
make -j4 && make install
Proj.4
- 要求 Proj.4 (>= 4.8.0) ;
- Proj.4 要先于 GDAL 安装!!!
export PATH=/Bioinfo/Pipeline/SoftWare/gcc-4.8.5/bin:$PATH
wget https://download.osgeo.org/proj/proj-4.8.0.tar.gz
tar zvxf proj-4.8.0.tar.gz -C ../build/
cd ../build/proj-4.8.0/
./configure --prefix=/Bioinfo/Pipeline/SoftWare/proj-4.8.0
make -j4 && make install
GDAL
- 要求 GDAL version >= 2.0.1;记住一定要加 --with-static-proj4 进行编译,否则 https://github.com/r-spatial/sf/issues/678 这个问题没法解决!!
- 尝试了一下 gdal-2.2.3+proj-4.9.1 组合,好像有问题(configure: error: GDALAllRegister not found in libgdal);
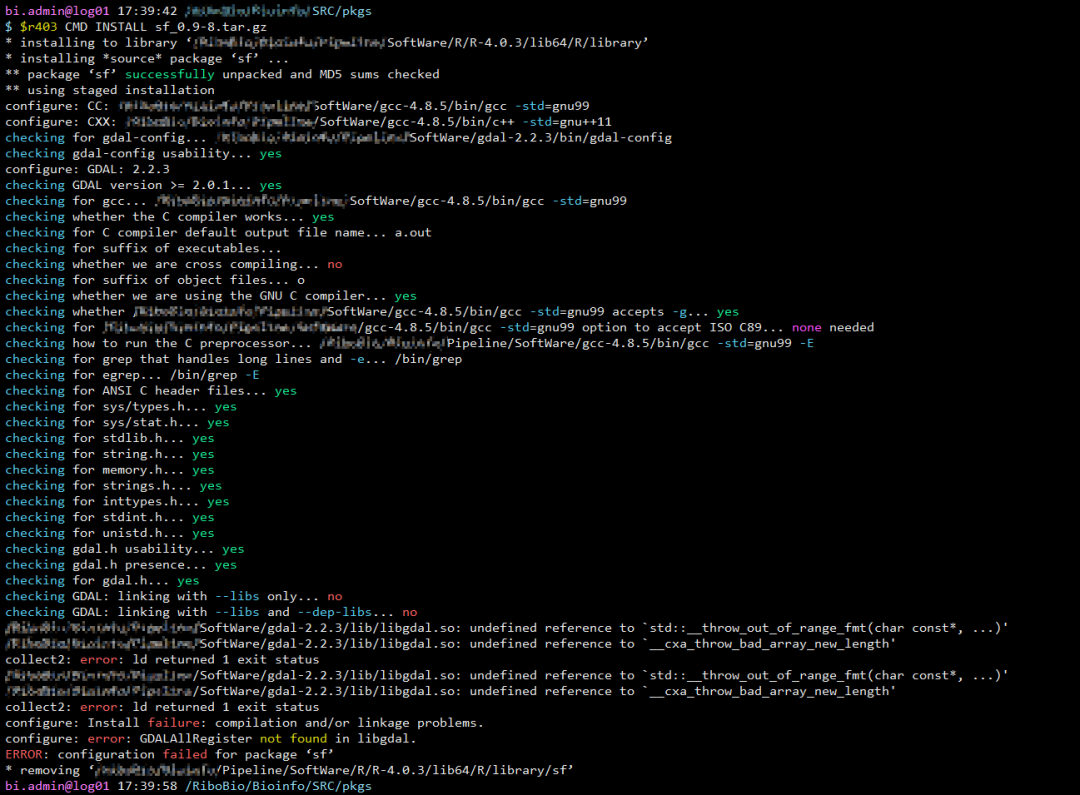
- 但是 GDAL-2.2.0+proj-4.8.0 是可以的!
$enabledevtoolset4
wget http://download.osgeo.org/gdal/2.2.0/gdal-2.2.0.tar.gz
tar zvxf gdal-2.2.0.tar.gz -C ../build/
cd ../build/gdal-2.2.0
./configure --prefix=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0 --with-static-proj4=/Bioinfo/Pipeline/SoftWare/proj-4.8.0/
make -j4 && make install
最终安装命令
export PATH=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0/bin:/Bioinfo/Pipeline/SoftWare/geos-3.4.0/bin:/Bioinfo/Pipeline/SoftWare/proj-4.8.0/bin:$PATH
export LD_LIBRARY_PATH=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0/lib:/Bioinfo/Pipeline/SoftWare/geos-3.4.0/lib:/Bioinfo/Pipeline/SoftWare/proj-4.8.0/lib:$LD_LIBRARY_PATH
出现 configure: error: libproj not found in standard or given locations. 异常,参考:https://github.com/r-spatial/sf/issues/1471 得到解决:
# configure: error: libproj not found in standard or given locations.
options("repos"=c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
install.packages('sf', configure.args='--with-gdal-config=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0/bin/gdal-config --with-geos-config=/Bioinfo/Pipeline/SoftWare/geos-3.4.0/bin/geos-config --with-proj-include=/Bioinfo/Pipeline/SoftWare/proj-4.8.0/include --with-proj-lib=/Bioinfo/Pipeline/SoftWare/proj-4.8.0/lib', configure.vars='GDAL_DATA=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0/share/gdal')
使用说明
安装完 ggVennDiagram 后,这个包可以直接library(ggVennDiagram)使用;但如果要使用sf包,则需要:
export LD_LIBRARY_PATH=/Bioinfo/Pipeline/SoftWare/gdal-2.2.0/lib:/Bioinfo/Pipeline/SoftWare/geos-3.4.0/lib:/Bioinfo/Pipeline/SoftWare/proj-4.8.0/lib:$LD_LIBRARY_PATH