🤩 Monocle 3 | 太牛了!单细胞必学R包!~(四)(差异分析之建模与评估)
🤩 Monocle 3 | 太牛了!单细胞必学R包!~(四)(差异分析之建模与评估)
1写在前面
今天发工资了,简直想死啊,门诊绩效4块多,不到5块。😬
我简直不敢相信自己的眼睛。🫠
现在真的是按劳分配吗!?🤒
天冷了,不方便骑车了,不知道有没有爱骑行的小伙伴,冬天你们封车吗?🤒
又是混过一天,不知所云。🫠
今天我们来讲一下如何使用monocle3进行差异分析。😘
2用到的包
library(tidyverse)
library(monocle3)
3示例数据
cds

4回归分析
Monocle的工作原理是对每个基因拟合一个回归模型。🧐
比如,在示例数据中,细胞是在不同的时间点收集的,我们可以通过首先对每个基因拟合一个广义的线性模型来检验上述任何一个基因的表达是否随时间变化。🤩
ciliated_genes <- c("che-1",
"hlh-17",
"nhr-6",
"dmd-6",
"ceh-36",
"ham-1")
cds_subset <- cds[rowData(cds)$gene_short_name %in% ciliated_genes,]
gene_fits <- fit_models(cds_subset, model_formula_str = "~embryo.time")
我们选择看下基因随时间的变化。😏
首先,我们使用coefficient_table()函数从每个模型中提取一个系数。😍
fit_coefs <- coefficient_table(gene_fits)
DT::datatable(fit_coefs)
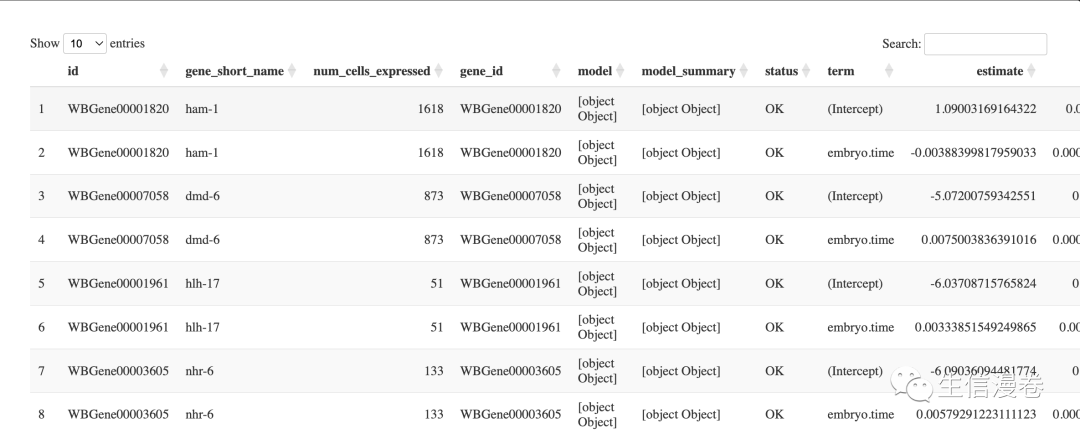
我们提取一下时间项。🥳
emb_time_terms <- fit_coefs %>% filter(term == "embryo.time")
过滤一下有意义的基因。🥰
emb_time_terms %>% filter (q_value < 0.05) %>%
select(gene_short_name, term, q_value, estimate)
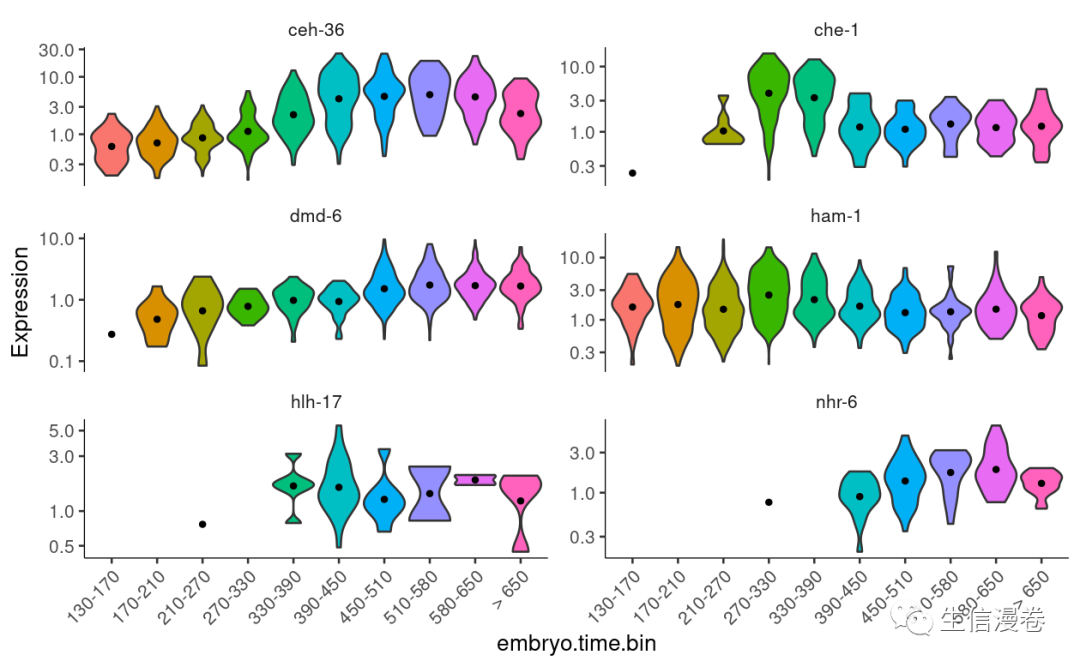
可视化一下吧,violin plot。😏
plot_genes_violin(cds_subset, group_cells_by="embryo.time.bin", ncol=2) +
theme(axis.text.x=element_text(angle=45, hjust=1))
5控制批次效应和混杂因素
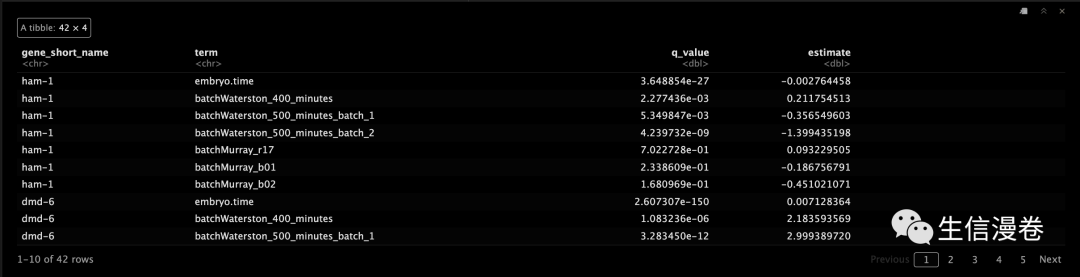
gene_fits <- fit_models(cds_subset, model_formula_str = "~embryo.time + batch")
fit_coefs <- coefficient_table(gene_fits)
fit_coefs %>% filter(term != "(Intercept)") %>%
select(gene_short_name, term, q_value, estimate)
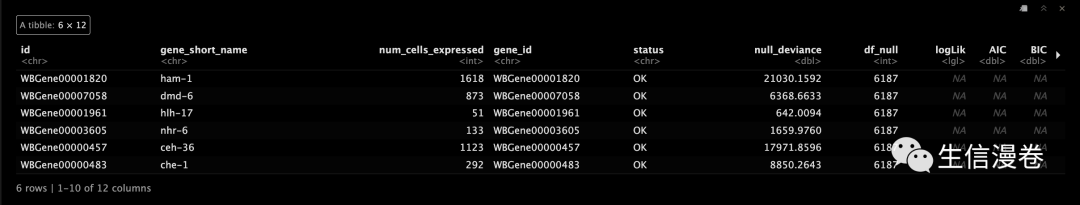
6评估模型
evaluate_fits(gene_fits)
有时候我们会想知道,建模时是否需要包括批次效应,这里也有个使用的功能,即compare_models()。🤓
这里我们建2个模型,一个有batch,一个没有batch。😘
这里,几乎所有的q_value都是有意义的,表明数据中存在大量的批次效应。🤒
因此,我们需要在模型中加入批处理项。😋
time_batch_models <- fit_models(cds_subset,
model_formula_str = "~embryo.time + batch",
expression_family="negbinomial")
time_models <- fit_models(cds_subset,
model_formula_str = "~embryo.time",
expression_family="negbinomial")
compare_models(time_batch_models, time_models) %>% select(gene_short_name, q_value)
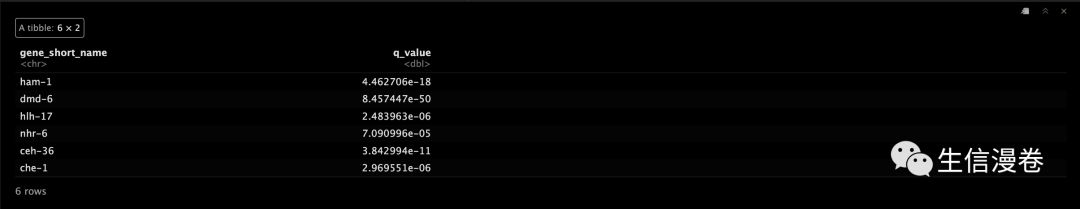
这里expression_family有多个多个选项,包括,quasipoisson, negbinomial, poisson, binomial。😬
比较推荐的就是negbinomial了。🥳

最后祝大家早日不卷!~
点个在看吧各位~ ✐.ɴɪᴄᴇ ᴅᴀʏ 〰 📍 往期精彩
📍 🤩 LASSO | 不来看看怎么美化你的LASSO结果吗!? 📍 🤣 chatPDF | 别再自己读文献了!让chatGPT来帮你读吧!~ 📍 🤩 WGCNA | 值得你深入学习的生信分析方法!~ 📍 🤩 ComplexHeatmap | 颜狗写的高颜值热图代码! 📍 🤥 ComplexHeatmap | 你的热图注释还挤在一起看不清吗!? 📍 🤨 Google | 谷歌翻译崩了我们怎么办!?(附完美解决方案) 📍 🤩 scRNA-seq | 吐血整理的单细胞入门教程 📍 🤣 NetworkD3 | 让我们一起画个动态的桑基图吧~ 📍 🤩 RColorBrewer | 再多的配色也能轻松搞定!~ 📍 🧐 rms | 批量完成你的线性回归 📍 🤩 CMplot | 完美复刻Nature上的曼哈顿图 📍 🤠 Network | 高颜值动态网络可视化工具 📍 🤗 boxjitter | 完美复刻Nature上的高颜值统计图 📍 🤫 linkET | 完美解决ggcor安装失败方案(附教程) 📍 ......