榕树集--RoseTTAFold All-Atom 的简介,安装以及使用
榕树集--RoseTTAFold All-Atom 的简介,安装以及使用


前言
之前追过一篇AF的最新文章,官方称之为AlphaFold-latest,他可以预测蛋白质和小分子,蛋白质和核酸的复合物结构,对于传统复合物预测方法来说是降维式打击。唯一的缺点是:小道消息得知,Deepmind在那是没有将其开源的打算。额,好吧,这是我的缺点。关于文章的具体细节,放在下面。
Well, Rosetta同样也给出了全原子的解决方案,文章发表于24年3月7日,还热乎。RoseTTAFold All-Atom(RFAA)可以预测蛋白质,核酸,小分子,金属,共价修饰的组装体,并且将其进行了开源。
我现在倒是比较好奇,我们是处在一个AI可以给出最终解决方案的时代,还是处于一个大家一起搭架子,建设改进的时代。
简介
深度学习方法已经彻底改变了蛋白质结构预测和设计领域。本文介绍了两种模型:1, RoseTTAFold All-Atom(RFAA),其可以对包含蛋白质,小分子,核算,金属和共价修饰的组装体进行建模;2,RFdiffusionAA,在降噪任务上进行微调之后获得了RFdiffusionAA,他可以围绕小分子来构建蛋白结构。
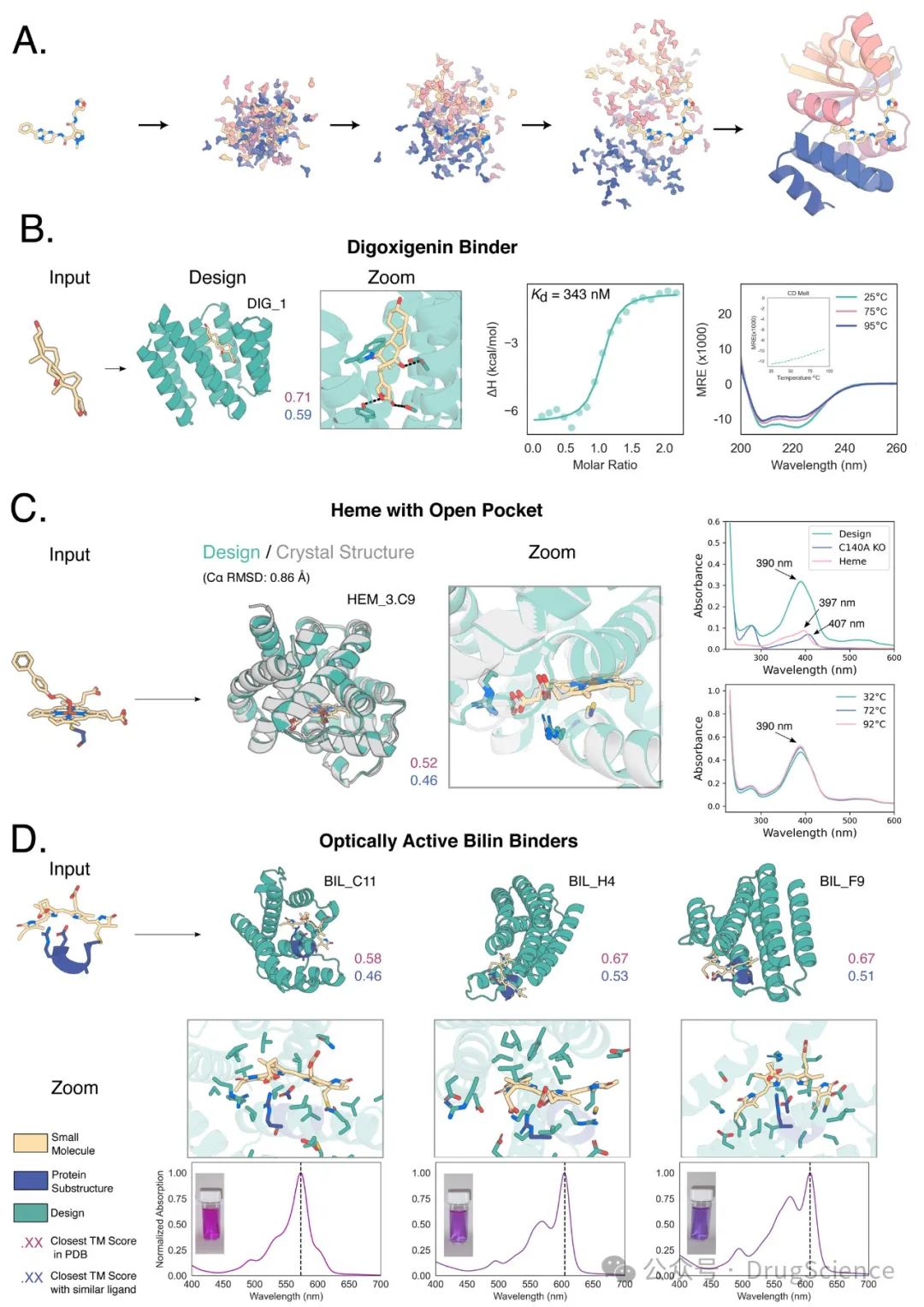
作者同样进行了实验验证,其设计了可以结合digoxigenin的蛋白,可以结合heme的蛋白,可以结合光捕获分子胆绿素的蛋白。
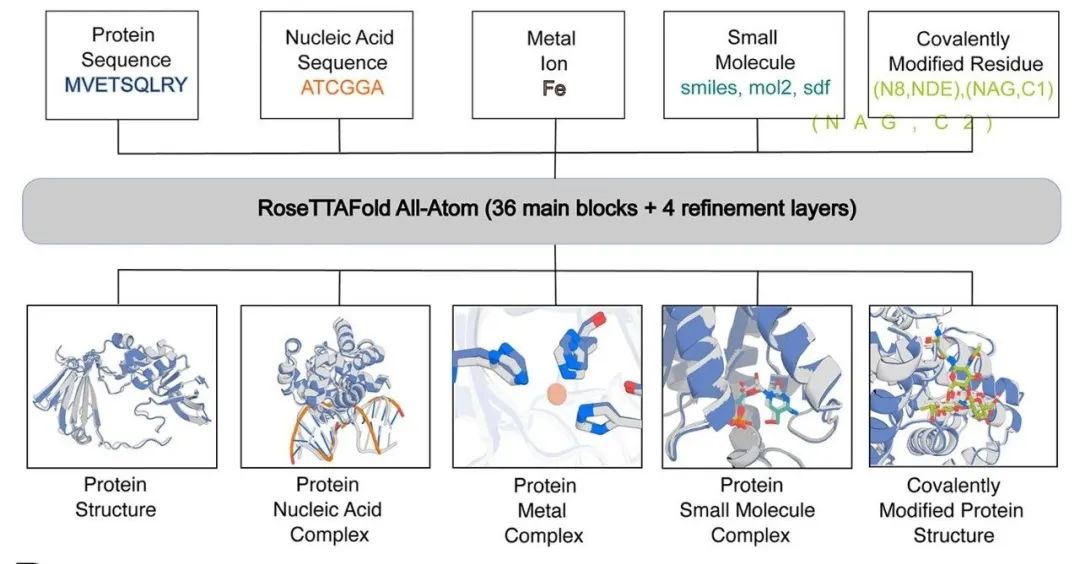
构建和训练RFAA
作者借鉴了RoseTTAFold2(RF2)的预测网络来进行设计。保留了RF2中的蛋白质和核酸的表示方式,并将小分子,共价修饰,非天然氨基酸表示为图(原子-键)。在1D track上,作者输入每个非聚合物原子的化学元素类型;2D track,输入原子之间的化学键;3D track,输入手性信息(R/S)。
作者从PDB数据库中整理出了一个蛋白质--生物分子复合物数据集,包含蛋白质--小分子,蛋白质--金属,和共价修饰的蛋白质复合物,常见的溶剂和添加剂被过滤掉。为了避免偏好,作者进行了聚类(30%的序列一致性)。同时为了帮助神经网络可以学习小分子通用属性,而不是PDB数据中的特定特征,作者将剑桥结构数据库(Cambridge Structural Database)中的小分子晶体结构数据作为补充训练集。
结果
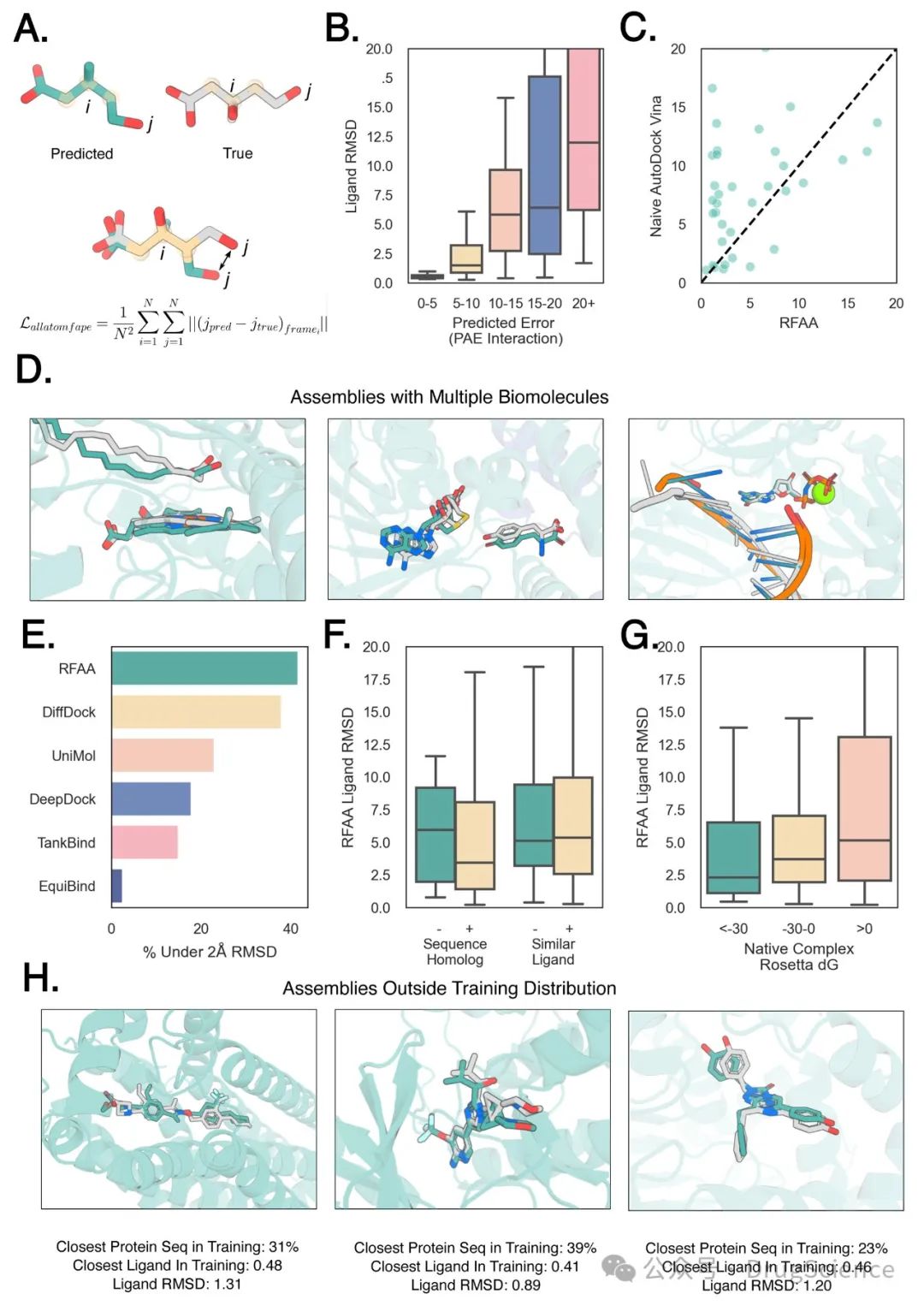
预测蛋白质小分子复合物
在CAMEO对接评估上构建了一个RFAA服务器,该服务器每周对提交给PDB的所有结构进行预测。在CAMEO的结构中,有43%的结构,RFAA预测结果呈现高置信度(PAE Interaction < 10),其中77%的高置信度结构相当准确,配体RMSD小于2Å,见上图(B)。同时在于Vina的比较中,RFAA预测成功率为32%,Vina仅为8%。RFAA最常见的失误是:分子位于正确的口袋,但是其分子朝向不正确。同时也应该注意到,如果专家操作,或许Vina的对接成功率会更高。
作者也比较了DiffDock和RFAA的预测结果,RFAA成功率为42%,而DiffDock成功率为38%,但是值得注意的是RFAA同时也预测了蛋白质的侧链和骨架,而DiffDock是直接获取了蛋白质结构进行的对接,RFAA的操作难度更大。
在结合蛋白质结构被提供的情况下,Vina会比RFAA表现好(52% vs 42%),因为RFAA的任务更为艰巨,它需要从序列中预测蛋白质的主链和侧链细节.
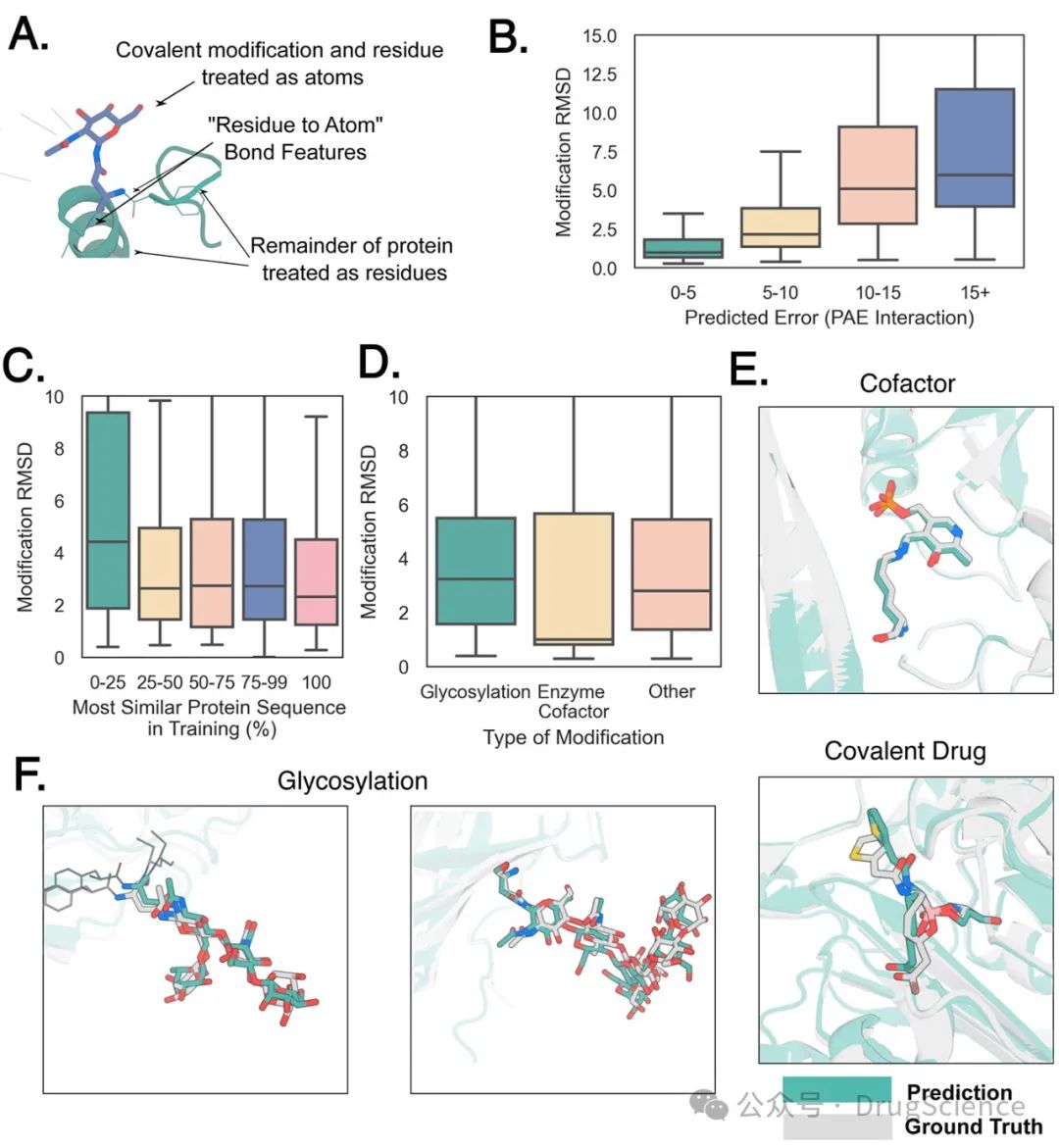
预测蛋白质共价键修饰
作者对PDB数据中的931个共价修饰的case进行了预测,成功率为46%(RMSD < 2.5Å, 此处RMSD指当其余部分对齐时,修饰残基的RMSD )。同时RFAA可以有效的预测聚糖结构,对引入糖基化的碳水化合物基团进行建模,测试集上的中位数RMSD为3.2 Å。RFAA并不是简单地学习结构模拟聚糖,因为预测结果与实验密度图相匹配。该网络即使在序列与训练集中的序列相差甚远的情况下,也能对聚糖相互作用进行准确的预测,而且能对多达七个单糖的聚糖进行预测。
从头设计小分子binder
之前的关于小分子的结合蛋白设计的研究工作中,经常使用的方法是将分子对接到天然的或者专家选择的蛋白质scaffold结构库中。基于扩散的方法可以生成与目标蛋白结合的蛋白,这些蛋白具有相当高的亲和力和特异性。通过扩散处理,作者可以生成与小分子紧密结合的蛋白质。测试集上,生成的蛋白质结构和真实的蛋白质结构高度相似。
讨论
RFAA表明,单个神经网络可以被训练来准确模拟包含多种非蛋白质组分的生物分子组装体。RFAA已经学习了蛋白质-小分子复合物的详细特征。首先,该网络能够对与训练数据集中的蛋白质和配体差异较大的复合物进行高精度预测,并且使用Rosetta物理模型计算的相互作用能量有助于复合物预测精度提高。其次,通过RFdiffusionAA也有助于设计小分子结合的新蛋白。
安装
总体评价,安装较为简单。
你可以直接依照此链接上的教程进行安装,https://github.com/baker-laboratory/RoseTTAFold-All-Atom。
我们这里给予一些,简介和注意事项。
1. 安装Mamba
wget "https://github.com/conda-forge/miniforge/releases/latest/download/Mambaforge-$(uname)-$(uname -m).sh"
bash Mambaforge-$(uname)-$(uname -m).sh # 接受所有项目,安装到默认位置
rm Mambaforge-$(uname)-$(uname -m).sh # (可选) 安装之后删除
source ~/.bashrc这里值得注意的是,当你原始环境中存在conda时,原有的conda环境路径,会被替换为mamba的conda路径。
2. 将这个package克隆下来
git clone https://github.com/baker-laboratory/RoseTTAFold-All-Atom
cd RoseTTAFold-All-Atom 3. 使用mamba创建一个RFAA环境
mamba env create -f environment.yamlconda activate RFAA # NOTE: 退出自然就是 conda deactivate RFAA cd rf2aa/SE3Transformer/pip3 install --no-cache-dir -r requirements.txtpython3 setup.py installcd ../../和conda操作基本一致,也没有什么其余的注意事项。
4. 下载signalp6以及对其进行配置
你可以从以下网址进行安装,注册一个学术版本的帐号:https://services.healthtech.dtu.dk/services/SignalP-6.0/
邮箱会收到邮件,使用wget下载 signalp-6.0h.fast.tar.gz
signalp6-register signalp-6.0h.fast.tar.gz # NOTE: 注册
mv $CONDA_PREFIX/lib/python3.10/site-packages/signalp/model_weights/distilled_model_signalp6.pt $CONDA_PREFIX/lib/python3.10/site-packages/signalp/model_weights/ensemble_model_signalp6.pt # NOTE:重命名 distilled 模型权重 5. 安装依赖项
bash install_dependencies.sh 6. 下载权重文件
wget http://files.ipd.uw.edu/pub/RF-All-Atom/weights/RFAA_paper_weights.pt 7. 下载MSA使用以及模板产生的序列库
# uniref30 [46G]
wget http://wwwuser.gwdg.de/~compbiol/uniclust/2020_06/UniRef30_2020_06_hhsuite.tar.gz
mkdir -p UniRef30_2020_06
tar xfz UniRef30_2020_06_hhsuite.tar.gz -C ./UniRef30_2020_06
# BFD [272G]
wget https://bfd.mmseqs.com/bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gz
mkdir -p bfd
tar xfz bfd_metaclust_clu_complete_id30_c90_final_seq.sorted_opt.tar.gz -C ./bfd
# structure templates (including *_a3m.ffdata, *_a3m.ffindex)
wget https://files.ipd.uw.edu/pub/RoseTTAFold/pdb100_2021Mar03.tar.gz
tar xfz pdb100_2021Mar03.tar.gz如果你之前安装过AF2,
- 那么BFD数据库可以不需要重复下载。
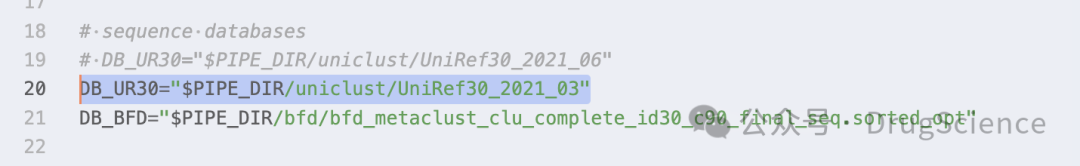
- uniref30数据库的请注意版本,这是2020年06,当然你也可以修改运行脚本,会放在后面。
- pdb100_2021Mar03.tar.gz,下载需要一场<年会不能停>的电影时长。解压也许要20-30mins左右。
8. 可选:
- 将数据库,创建软链接在本地
如果你是在
RoseTTAFold-All-Atom文件夹下进行下载解压的活,此步可以跳过。因为我的数据库文件存储在固定的位置,所以设置了很多的软链接。 - 给予make.sh权限 chmod 777 input_prep/make.sh
运行
上述完成之后,就可以直接运行了。
# 单体蛋白预测
python -m rf2aa.run_inference --config-name protein
# 蛋白质核酸复合物预测
python -m rf2aa.run_inference --config-name nucleic_acid
# 蛋白质小分子复合物预测
python -m rf2aa.run_inference --config-name protein_sm运行过程大概,10-30mins左右
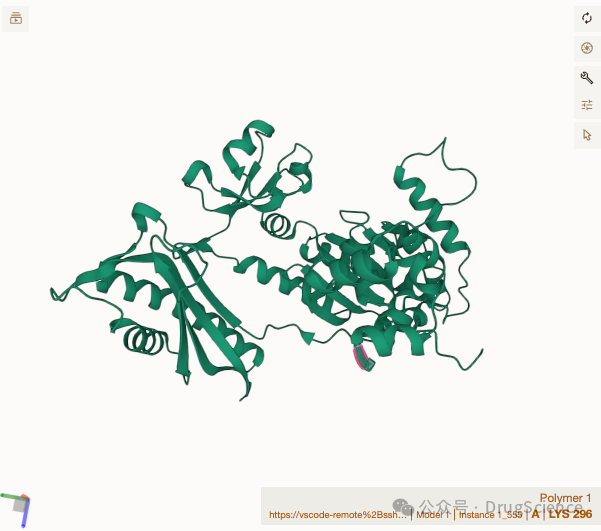
- 蛋白质结构预测
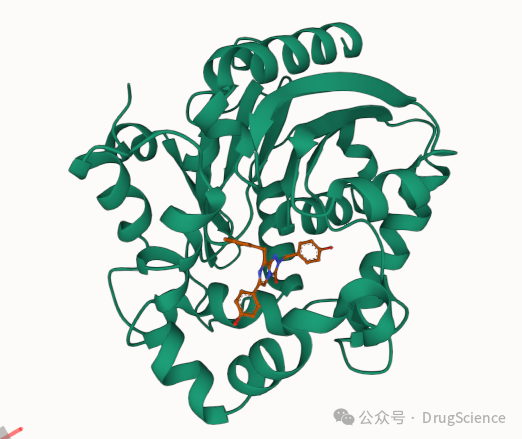
- 蛋白质小分子复合物预测
你可以在rf2aa/config/inference中找到配置文件,并进行自定义的配置。
如下,并没有很多需要解释的地方。
参考
- Rohith Krishna et al.,Generalized biomolecular modeling and design with RoseTTAFold All-Atom.Science0,eadl2528DOI:10.1126/science.adl2528