Python 离群点检测算法 -- PCA
Python 离群点检测算法 -- PCA

PCA 如何工作?
高维数据集是指包含大量变量的数据集,也称为 "维度诅咒",通常给计算带来挑战。尽管大功率计算在某种程度上可以处理高维数据,但在许多应用中,仍有必要降低原始数据的维度。PCA 能够降低由大量相关变量组成的数据集的维度,并尽可能地保留方差。它找到新的变量,而原始变量只是它们的线性组合,这些被称为主成分(PC)。主成分是正交的,即彼此垂直。
线性变换如图(A)所示,通过旋转原始的 X 轴和 Y 轴来更好地拟合数据体(红色部分)。主成分分析中的第一个主成分(PC1)捕捉到数据中最大的方差,而第二个主成分则捕捉到了PC1未能捕捉到的数据中的最大差异。接下来的主成分将继续捕捉前几个未能捕捉到的方差,直到所有方差都被解释。主成分的数量应当等于原始变量的数量。
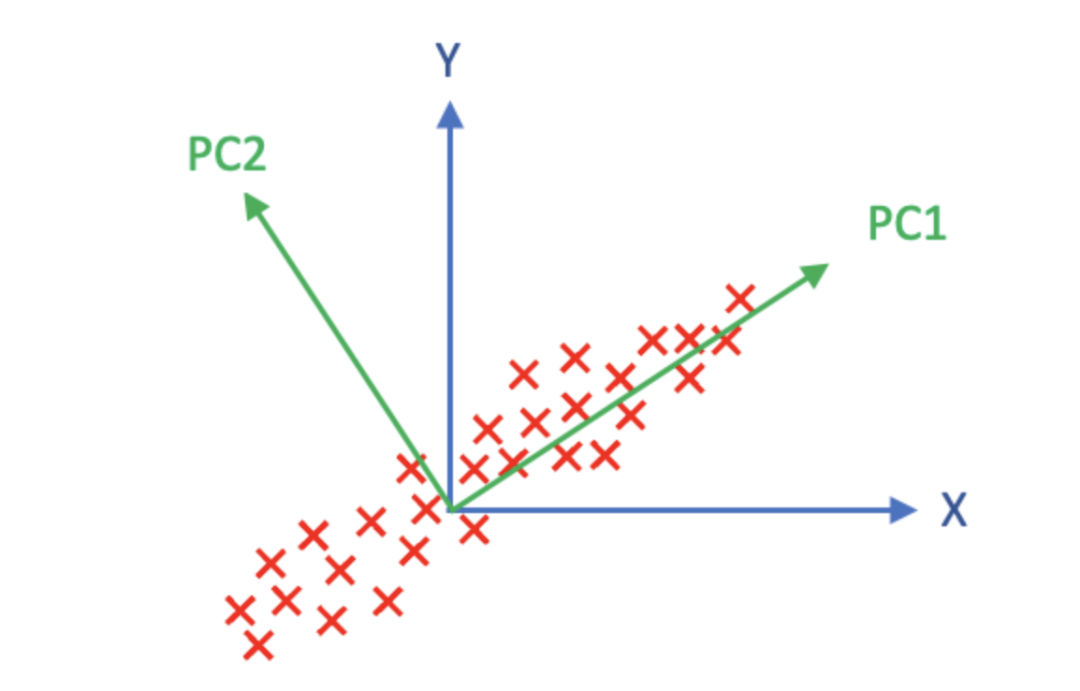
PCA
在线性变换中,协方差矩阵可以被分解成特征值相关的正交向量,即特征向量。特征值是用来缩放特征向量的因子。特征值高的特征向量能够捕捉到数据中的大部分方差。德语单词"Eigen"意思是"自己的"或"典型的"。因此,特征向量也可以称为"特征向量"。
降维可以找出异常值
当维度减少到几个主要维度时,模式就会被识别出来,然后异常值就会显现出来。离群值是指与其他观测值偏差很大的观测值,以至于让人怀疑它是由不同的机制产生的。由于离群值往往遵循不同的工具,它们通常不在前几个主成分中。当它们被投影到低维超平面上时,会落在一些特征值较小的独特特征向量上。可以说,离群点检测是降维的副产品。根据这一特性,PCA 中数据点的离群点得分可用以下公式表示:
离群点得分 = 每个观测点到由所选特征向量构建的超平面之间的加权欧氏距禂之和。
运行 PCA 之前切记对数据进行标准化处理
在进行 PCA 分析之前,数据需要被标准化处理。标准化后,所有变量的标准差和权重都将相同。如果忽略标准化步骤,在计算坐标轴时,标准差较大的变量会得到更高的权重。另一个标准化的考虑是数据集中的不同变量可能具有不同的测量单位,例如美元金额和单位等。因此,有必要对所有变量的数据进行标准化处理。在 PyOD 中的 PCA 类中,内置了对数据进行标准化处理的程序,可以在执行 PCA 之前使用。
建模流程
步骤 1 - 建立模型
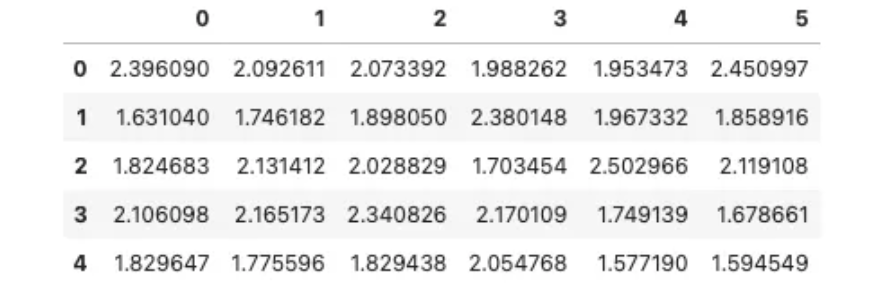
我生成了一个包含 500 个观测值和 6 个变量的模拟数据集。异常值的百分比被设定为 5%。无监督模型只使用 X 变量,而模拟数据集中的目标变量 Y 仅用于验证。
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
from pyod.utils.data import generate_data
contamination = 0.05 # percentage of outliers
n_train = 500 # number of training points
n_test = 500 # number of testing points
n_features = 6 # number of features
X_train, X_test, y_train, y_test = generate_data(
n_train=n_train,
n_test=n_test,
n_features= n_features,
contamination=contamination,
random_state=123)
X_train_pd = pd.DataFrame(X_train)
X_train_pd.head()
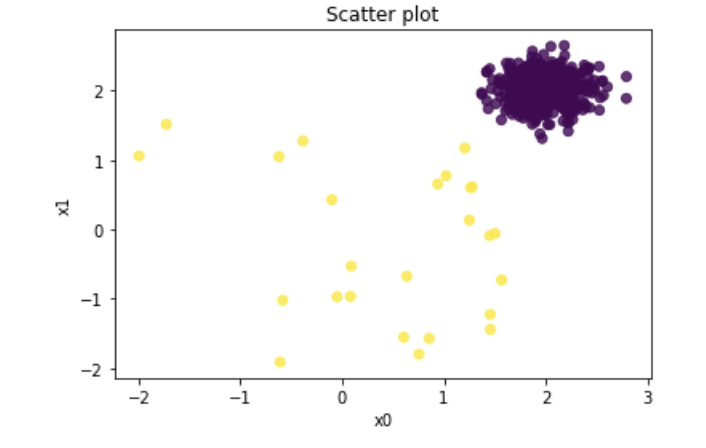
两个变量的散点图
上图中黄点为异常值,紫点为正常数据点。
# Plot
plt.scatter(X_train_pd[0], X_train_pd[1], c=y_train, alpha=0.8)
plt.title('Scatter plot')
plt.xlabel('x0')
plt.ylabel('x1')
plt.show()
下面的代码用于构建模型,并对训练数据和测试数据进行评分。
label_:训练数据的标签向量,当使用.predict()训练数据时也一样。decision_scores_:训练数据的分数向量,当使用.decision_functions()训练数据时也一样。decision_score():为每个观测值分配离群值分数的评分函数。predict():根据指定的阈值给出1或0的预测函数。contamination:表示离群值的百分比,默认为10%。在大多数情况下,我们无法知道异常值的百分比,因此可以根据任何先验知识进行赋值。该参数不会影响离群值分数的计算。
from pyod.models.pca import PCA
pca = PCA(contamination=0.05)
pca.fit(X_train)
# get the prediction labels and outlier scores of the training data
y_train_pred = pca.labels_ # binary labels (0: inliers, 1: outliers)
y_train_scores = pca.decision_scores_ # .decision_scores_ yields the raw outlier scores for the training data
y_train_scores = pca.decision_function(X_train) # You also can use .decision_function()
y_train_pred = pca.predict(X_train) # You also can use .decision_function()
y_test_scores = pca.decision_function(X_test) # You also can use .decision_function()
y_test_pred = pca.predict(X_test) # You also can use .decision_function()
PCA 的默认参数包括默认为 "true" 的标准化和 5% 的污染率设置。
pca.get_params()
{'contamination': 0.05,
'copy': True,
'iterated_power': 'auto',
'n_components': None,
'n_selected_components': None,
'random_state': None,
'standardization': True,
'svd_solver': 'auto',
'tol': 0.0,
'weighted': True,
'whiten': False}
在PCA中,解释方差比是一个重要指标,表示每个主成分解释的总方差的比例。在PyOD中,可以通过函数.explained_variance_ratio_来获取解释方差。例如,第一个主成分解释了73.4%的方差,第二个主成分解释了7.4%,第三个主成分解释了5.6%,以此类推。总共六个主成分的解释方差之和为100%。
[pca.explained_variance_,
pca.explained_variance_ratio_]
[array([4.13739583, 0.55186189, 0.38712658,
0.34746491, 0.31134247, 0.27683236]),
array([0.68818684, 0.09179303, 0.06439205,
0.057795 , 0.05178663, 0.04604645])]
步骤 2 - 确定合理的阈值
PyOD内置函数"threshold_"可以根据给定的污染率计算训练数据的阈值,默认值为10%。模型中的值设置为5%,所以阈值为5%。
print("The threshold for the defined comtanimation rate:" , pca.threshold_)
The threshold for the defined comtanimation rate:
219.77196222591996
通常情况下,我们不知道异常值的百分比,可以使用异常值得分直方图来选择合理的阈值。如果有先验知识表明异常值占1%,则应选择导致异常值约为1%的阈值。PCA离群点得分直方图显示阈值为200.0,因为直方图中存在一个自然切点。
# get the prediction on the test data
y_test_pred = pca.predict(X_test) # outlier labels (0 or 1)
y_test_scores = pca.decision_function(X_test) # outlier scores
import matplotlib.pyplot as plt
plt.hist(y_train_scores, bins='auto') # arguments are passed to np.histogram
plt.title("Histogram with 'auto' bins")
plt.xlabel('PCA outlier score')
plt.show()
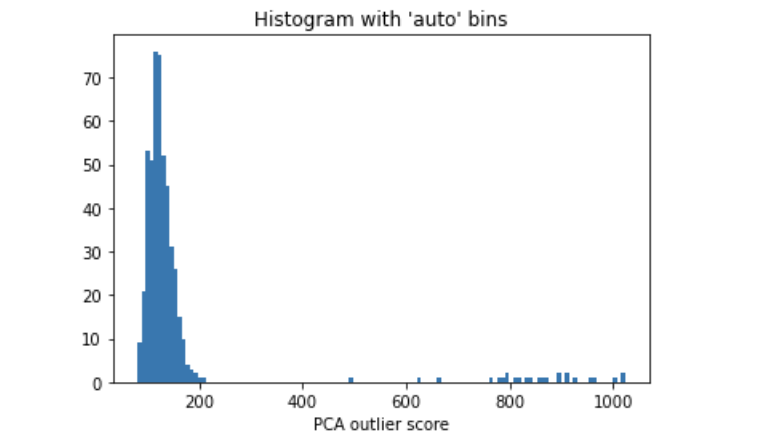
PCA 离群值直方图
第 3 步--展示正常组和异常组的描述性统计
分析正常组和离群组对于验证模型的合理性至关重要。正常组和异常组的特征统计数据应与先前的领域知识一致。如果异常组中某个特征的平均值出现异常,建议您重新检查、修改或放弃该特征。需要反复进行建模过程,确保所有特征与先验知识保持一致。
threshold = pca.threshold_
threshold
def descriptive_stat_threshold(df,pred_score, threshold):
# Let's see how many '0's and '1's.
df = pd.DataFrame(df)
df['Anomaly_Score'] = pred_score
df['Group'] = np.where(df['Anomaly_Score']< threshold, 'Normal', 'Outlier')
# Now let's show the summary statistics:
cnt = df.groupby('Group')['Anomaly_Score'].count().reset_index().rename(columns={'Anomaly_Score':'Count'})
cnt['Count %'] = (cnt['Count'] / cnt['Count'].sum()) * 100 # The count and count %
stat = df.groupby('Group').mean().round(2).reset_index() # The avg.
stat = cnt.merge(stat, left_on='Group',right_on='Group') # Put the count and the avg. together
return (stat)
descriptive_stat_threshold(X_train,y_train_scores, threshold)
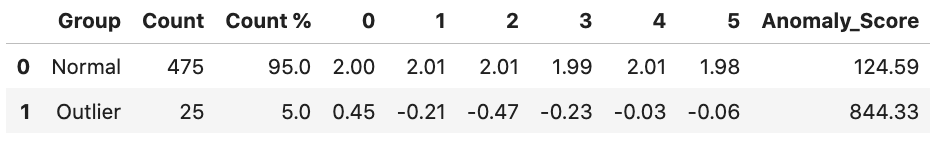
正常组和离群组的描述性统计
正常组和异常组的特征已显示于上表,显示了它们的计数和计数百分比。重要结果包括:
- 离群组的规模一旦确定了阈值就确定了,大小统计可作为参考。
- 每组中的特征统计量,离群组的均值小于正常组的均值。
- 异常点组的平均得分应高于正常组(844.33>124.59)。
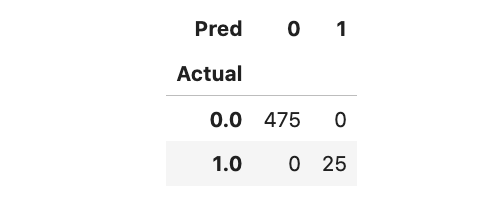
基于生成数据,我们可以利用混淆矩阵来了解模型性能,它成功识别了所有 25 个异常值。
Actual_pred = pd.DataFrame({'Actual': y_test, 'Anomaly_Score': y_test_scores})
Actual_pred['Pred'] = np.where(Actual_pred['Anomaly_Score']< threshold,0,1)
pd.crosstab(Actual_pred['Actual'],Actual_pred['Pred'])
模型之间的比较
现在已经建立了多个模型。这些模型之间存在重叠。多个模型是否都能识别出离群值。
########
# HBOS #
########
from pyod.models.hbos import HBOS
n_bins = 50
hbos = HBOS(n_bins=n_bins,contamination=0.05)
hbos.fit(X_train)
y_train_hbos_scores = hbos.decision_function(X_train)
y_test_hbos_scores = hbos.decision_function(X_test)
########
# ECOD #
########
from pyod.models.ecod import ECOD
clf_name = 'ECOD'
ecod = ECOD(contamination=0.05)
ecod.fit(X_train)
y_test_ecod_scores = ecod.decision_function(X_test)
########
# PCA #
########
from pyod.models.pca import PCA
pca = PCA(contamination=0.05)
pca.fit(X_train)
# get the prediction labels and outlier scores of the training data
y_test_pca_scores = pca.decision_function(X_test) # You also can use .decision_function()
# Thresholds
[ecod.threshold_, hbos.threshold_, pca.threshold_]
[16.320821760780653, 5.563712646460526, 219.77196222591996]
我将实际 Y 值、HBO 分数、ECOD 分数和 PCA 分数放在一个数据框中。
# Put the actual, the HBO score and the ECOD score together
Actual_preds = pd.DataFrame({'Actual': y_test,
'HBO_Score': y_test_hbos_scores,
'ECOD_Score': y_test_ecod_scores,
'PCA_Score': y_test_pca_scores})
Actual_preds['HBOS_pred'] = np.where(Actual_preds['HBO_Score']>hbos.threshold_,1,0)
Actual_preds['ECOD_pred'] = np.where(Actual_preds['ECOD_Score']>ecod.threshold_,1,0)
Actual_preds['PCA_pred'] = np.where(Actual_preds['PCA_Score']>pca.threshold_,1,0)
Actual_preds.head()
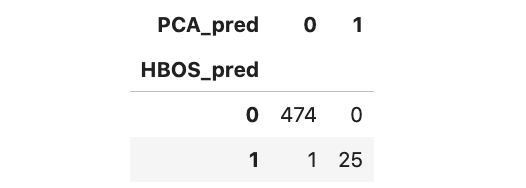
当对HBOS和PCA的预测结果进行交叉分析时,发现两个模型都存在25个异常值。交叉分析其他分数也是很有帮助的,尤其是在没有基本事实的情况下。
pd.crosstab(Actual_preds['HBOS_pred'],Actual_preds['PCA_pred'])

PCA 算法总结
异常值与正常数据点不同,它们在投影到低维超平面时会落在特征值较小的特征向量上。数据点的离群值是该数据点与低维超平面之间的加权欧氏距离之和。