AlphaFold3及其与AlphaFold2相比的改进
AlphaFold3及其与AlphaFold2相比的改进

蛋白质结构预测是生物化学中最重要的挑战之一。高精度的蛋白质结构对于药物发现至关重要。蛋白质结构预测始于20世纪50年代,随着计算方法和对蛋白质结构的认识不断增长。最初主要采用基于物理的方法和理论模型。当时的计算能力有限,这些模型往往难以成功地预测大多数蛋白质的结构。蛋白质结构模型的下一个发展阶段是同源建模,出现在20世纪70年代。这些模型依赖于同源序列具有相似结构的原理。通过将目标序列与已知结构的模板序列进行多序列比对,首次成功地确定了以前未解决的序列的结构。然而,这些模型的分辨率仍然有限。20世纪80年代出现了从头开始的方法,带来了下一个分辨率提升。这些方法应用了基于物理的技术和优化算法。结合计算技术的进步,这导致了蛋白质结构预测的显著改进。为了对所有这些新方法进行基准测试,从90年代初开始了蛋白质结构预测技术评估的关键阶段(CASP)系列活动。近年来,机器学习和深度学习技术已经越来越多地集成到蛋白质结构预测方法中,尤其是自2007年以来使用长短期记忆(LSTM)以来。
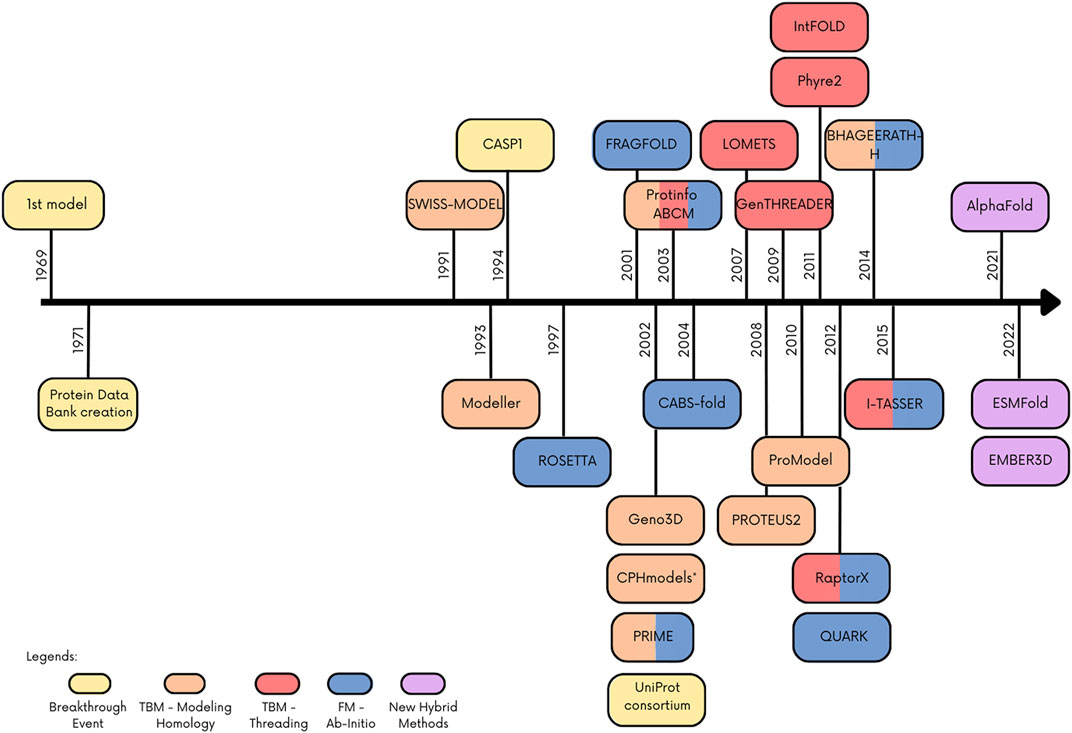
谷歌的DeepMind在2018年举办的第13届CASP比赛中推出了AlphaFold。AlphaFold采用了神经网络方法,直接使用蛋白质序列和同源序列预测给定蛋白质的所有非氢原子的三维坐标。虽然在CASP13中立即成为最佳预测方法,但仍然不足以称之为成功的蛋白质结构预测任务。成功的理论方法应该达到实验精度。AlphaFold2达到了这个精度,在CASP14中推出了一种重新设计的模型结构。它由Evoformer模块和结构模型组成,如下图所示。
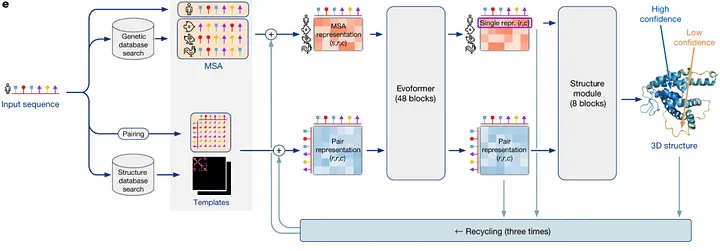
AlphaFold2的网络架构
AlphaFold2发布后不久,出现了具有类似架构的模型,旨在提高其准确性或加快速度。一些例子包括来自David Baker实验室的RosettaFold,MetaAI的OmegaFold或ESMFold。DeepMind与欧洲生物信息学研究所合作创建了AlphaFold-EBI数据库,其中包含了超过2亿个蛋白质的预测结构,覆盖了UniProt中的大多数序列。许多生物技术公司经常使用这些预测来设计新药物。
AlphaFold3 性能
Nature | AlphaFold 3 预测了所有生命分子的结构和相互作用
2024年5月8日,Isomorphic Labs和DeepMind发布了AlphaFold3。尽管AlphaFold3在预测单个蛋白质结构方面比AlphaFold2更精确,但AlphaFold3的主要优势在于其对蛋白质复合物的更精确预测,以及将其应用从蛋白质扩展到其他分子,几乎包括了蛋白质数据银行(PDB)中的所有分子。例如,AlphaFold3在PoseBusters基准测试中的蛋白质-配体界面上表现优于经典对接工具如最新版本的Vina和最近的机器学习工具如RoseTTAFold All-Atom,该基准测试包含了2021年之前发布的428个来自PDB的蛋白质-配体结构。与SOTA RoseTTAFold2NA和CASP15中最佳AI算法Alchemy_RNA相比,AlphaFold3在蛋白质-核酸复合物和RNA结构的预测中也获得了更高的准确性,该数据来自CASP15的示例和PDB蛋白质-核酸数据集。在CASP15基准测试中,最佳的人工专家辅助AIchemy_RNA2略优于AF3。
此外,AlphaFold3还更准确地预测了蛋白质、RNA或DNA上共价修饰的效应,如成键配体、糖基化、修饰的蛋白质残基和核酸碱基。然而,没有与其他工具进行比较。在高质量实验数据集上,通过口袋RMSD(均方根偏差)小于2Å来衡量的良好预测数量,从RNA修饰残基的40%到几乎80%的成键配体之间有所变化。数据集中的样本数量有限,因此这些值存在相对较高的统计误差。AlphaFold3还改进了对蛋白质-蛋白质复合物的预测,在抗体-蛋白质界面方面与AlphaFold-Multimer v2.3相比有显著改善。
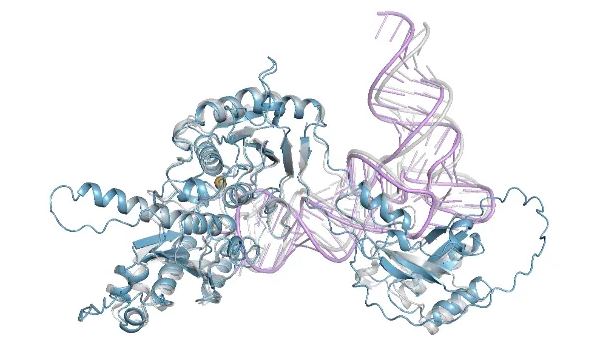
AlphaFold3 prediction of the 8AW3 — RNA modifying protein.
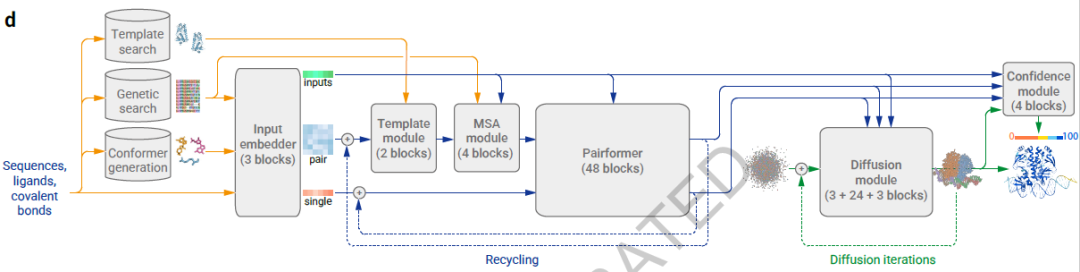
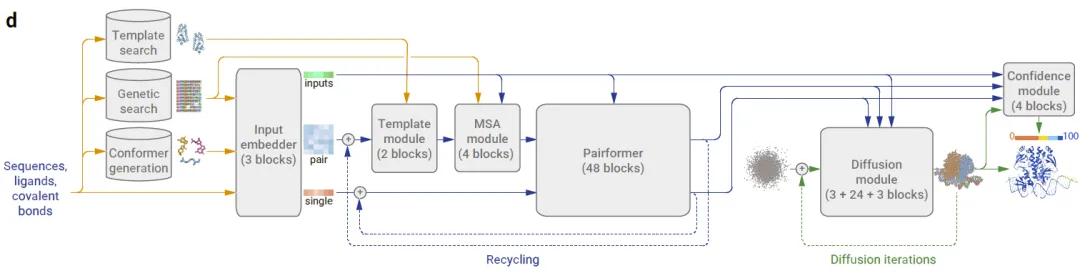
AlphaFold3的总体架构
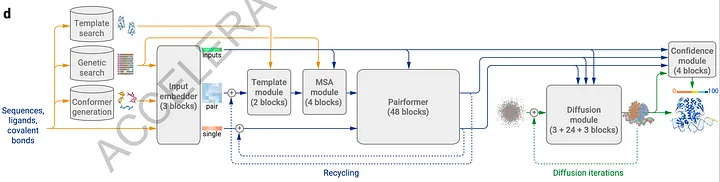
AlphaFold 3的网络架构
模型的架构与AlphaFold2的架构类似,但许多步骤已经得到改进,以更准确地预测蛋白质结构(复合物)。与AlphaFold2类似,进行了模板搜索和遗传搜索。除了执行构象搜索的输出外,这两个模块都被用作模板模块和MSA模块的输入。MSA模块比AlphaFold2中的更。Pairformer模块替换了AlphaFold2中的Evoformer模块。该模块仅处理单个和成对表示,而不处理MSA表示。AlphaFold2中的架构模型被扩散模型所取代。作为一种生成方法,扩散提供了结构的分布,而不是单个带有不确定性的结构,使最终预测变得准确,并避免了参数化。重要的是,在扩散过程中不需要像AlphaFold2中对侧链原子位置进行的基于物理的最小化(由AMBER执行)。为了在扩散过程中防止在未结构化区域生成物理上合理的结构,使用了来自AlphaFold-Multimer v2.3的训练数据进行交叉蒸馏,该数据集中包含了这些区域的环。最后,引入了一个置信度模块来衡量从原子级和成对计算中的错误的置信度。
现在,让我们逐步了解所有这些模块,了解它们的功能以及与AlphaFold2相比如何改进了蛋白质结构的预测。
AlphaFold3算法
与AlphaFold2中的架构类似,AlphaFold3中的架构依赖于Transformers。主要算法如下:

AlphaFold 3的算法
Step 1: 输入 token 和嵌入
首先,需要将输入的分子(蛋白质、RNA、DNA、小分子)转换为数学形式。分子由原子和连接这些原子的键描述。然而,蛋白质、RNA 和 DNA 具有规则化的一般结构(例如,蛋白质由氨基酸组成,DNA 和 RNA 由核酸组成),这使得在原子级别上表示这些实体过于详细。因此,使用不同的标记来表示不同的分子。AlphaFold2是一个纯蛋白质结构预测工具,没有考虑非蛋白质结合伙伴。AlphaFold2中有23个标记:20个标记代表标准氨基酸中的每一个,一个标记代表未知氨基酸,一个代表间隙(gap)的标记,以及一个用于掩码多序列比对(MSA)的标记。在AlphaFold3中,还考虑了RNA、DNA 和一般分子。对于DNA 和 RNA,标记对应于整个核苷酸。对于所有其他一般分子,一个标记由一个单个重原子表示。
这些标记具有必须进行嵌入的特征。在AlphaFold3中使用以下特征:
蛋白质特征包括残基编号(residue_index)、标记编号(token_index,从输入的起始标记单调递增)、链编号(asym_id)、序列编号(entity_id)、共享序列的链的 ID(sym_id)、残基类型(restype,20(+1 未知)种氨基酸,4(+1 未知)种 RNA 核苷酸,4(+1 未知)种 DNA 核苷酸,间隙残基)以及指定分子类型的标记(is_protein / rna / dna / ligand)。
参考构象特征包括在应用随机旋转和平移后的原子位置(ref_pos,以 Å 为单位给出的 3 个值)、用于构象的掩码标记(ref_mask,指示构象中使用的原子)、原子的元素的原子序号(ref_element)、原子的电荷(ref_charge)、唯一原子名称(ref_atom_name_chars)以及每个链 ID 和残基索引的唯一 ID(ref_space_uid)。
MSA 特征包括已处理的 MSA 的编码(msa)、一个二进制特征 has_deletion,指示左侧是否存在缺失,左侧的标准化删除数(deletion_value)、主 MSA 在残基类型上的分布情况,以及主 MSA 中的平均删除数(deletion_mean)。
模板特征包括模板序列(template_restype)、用于模板伪 β 掩码(template_pseudo_beta_mask)和模板骨架框架掩码(template_backbone_frame_mask)的掩码标记,分别指示该残基是否存在 CB 原子,以及该残基的所有骨架原子是否存在坐标,所有 CB 原子间距的成对编码(template_distogram)以及具有所有残基 CA 原子位移的向量(template_unit_vector)。
最后,有一个键特征(token_bonds),一个二维矩阵,指定了所有聚合物-配体和配体-配体键之间是否存在键。如果它们在2.4 Å范围内,则存在键。
这些特征必须被嵌入。这需要上下文嵌入(通过输入嵌入器实现)和位置编码。

这里,f 是上面提到的特征。在AlphaFold3中,执行了两种类型的嵌入。
用户通过 token_bonds 特征指定的键特征由线性嵌入处理。它们通过包含权重矩阵 W 和偏置向量 b 的线性变换的线性层。
残基类型、参考构象、MSA 分布和 deletion_mean 特征使用下面显示的算法进行嵌入。这意味着所有转换的每个原子特征都使用 AtomAttentionEncoder,并且编码器的每个原子特征输出然后与 restype、MSA 分布和 deletion_mean 特征连接。最后两个特征可以在 MSA 处理之前计算,因为它们仅应用于主 MSA。


所有原子特征首先被连接成一个大矩阵,然后通过一个没有偏置的线性层,这意味着它们被一个权重矩阵相乘。这样就为配体分子中的所有 Nₐₜₒₘₛ 原子创建了输出向量 cₗ。这些向量的长度为 cₐₜₒₘ = 128,例如,每个原子来自分子输入特征的 128 个任意特征(实值)。

相对距离是在参考构象中的所有两个原子的组合之间计算的。这些距离与一个权重矩阵相乘。如果原子 l 和 m 来自相同的链 ID 和残基索引,则将结果加到 pₗₘ 上,这意味着仅计算内部残基距离偏移量。

首先计算了配对原子之间的倒数平方距离,并与一个权重矩阵相乘。然后对于相同的残基,将结果进行嵌入。此外,在乘以一个权重矩阵之后,也会对掩码进行嵌入(如果距离属于相同的残基,则为1,否则为0)。

保存单原子表示以供进一步操作。

对于位置 l 处的标记索引的主干嵌入(见下文)进行了层归一化(实际上是减去所有值的均值并除以标准差)。然后将它们乘以一个权重矩阵。将结果添加到原子单个表示中。类似地,对于属于标记 l 和 m 的标记索引的所有成对嵌入,经过层归一化并乘以一个权重矩阵后,将它们添加到成对嵌入 pₗₘ 中。在乘以噪声权重矩阵后,将噪声 rₗ 添加到其中。请注意,所有步骤只有在给定噪声 rₗ 时才执行,而对于上面的 1D 嵌入,这并不是这种情况。

单原子l和m的表示也影响它们的相互作用。应用 ReLU 激活函数并将其与特定权重矩阵相乘后,将总和添加到成对嵌入中。
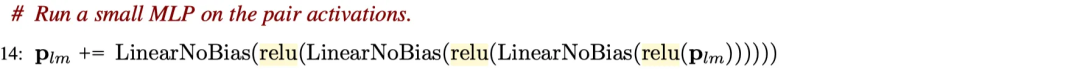
成对嵌入通过由三层组成的多层感知器,具有 ReLU 激活函数和权重矩阵,无偏差。
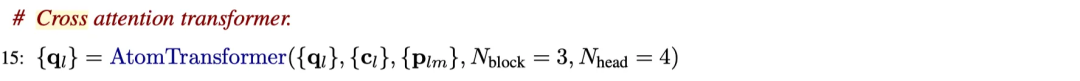
前面的步骤已经改变了单个原子的嵌入 cₗ,特别是通过包含主干嵌入(步骤 9)。为了不丢失原始嵌入的信息,并包括来自所有其他原子 m 的成对嵌入(主要是来自同一个残基的),应用了 AtomTransformer。AtomTransformer 使用三个块和每个块四个头的多头交叉注意力应用于由未修改的单个原子嵌入 qₗ、修改后的单个原子嵌入 cₗ 和成对嵌入 pₗₘ 表示的输入。AtomTransformer 执行实际的条件扩散。

最终的单个原子表示通过另一个没有偏置的线性层,并使用ReLU激活函数。嵌入将通过对属于标记 i 的所有原子取均值来转换为分数 aᵢ。最后,标记 i 的嵌入以及单个原子和成对嵌入由 AtomAttentionEncoder 返回。在上述输入嵌入的情况下,仅使用标记嵌入,并将其与 restype、MSA 分布和 deletion_mean 特征连接,以给出标记 i 的输入嵌入 sᵢ。
Steps 2 and 3: 线性层和成对嵌入
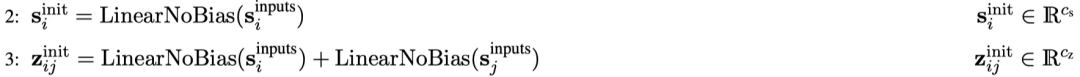
第一步计算得到的输入嵌入通过一个权重矩阵相乘,生成标记 i 的初始单个标记嵌入 sᵢ。将单个标记嵌入 i 和 j 乘以特定的权重矩阵后,生成初始成对标记嵌入 zᵢⱼ。需要注意的是,单个标记嵌入的长度为 384,而成对嵌入的长度较短,为 128。
Step 4: 相对位置编码
上面计算得到的成对嵌入包含了基于其特征的标记信息。然而,它们并不包含关于标记在输入序列中的顺序或位置的信息。这些信息被包含在位置编码中。AlphaFold2 和 AlphaFold3 使用两个标记之间的相对位置编码。相对位置编码已经在 AlphaFold2 中使用,并且具有一个优点,即(AlphaFold)模型的质量不会随着模型训练时序列长度的增加而下降。如果没有相对编码,不同位置的输入序列中相同的残基或链可能具有相同的嵌入。

相对位置编码在 AlphaFold3 中使用以下算法计算。

步骤1、2和3分别确定了来自相同链的标记对 i 和 j,具有相同的残基索引,甚至具有相同的实体。
步骤4和5计算了在相同链中(步骤1)标记 i 和 j 在输入序列中的相对位置的 one-hot 编码(根据它们的残基编号 residue_index)。这个相对位置由 rₘₐₓ = 32 进行剪裁,意味着在同一链中相距超过 32 个残基的残基不会根据其位置被区分。这种剪裁已经在 AlphaFold2 中使用。它减少了主要序列距离的影响;换句话说,它强调了标记之间其他输入特征差异的影响,正如上面计算的那样。
步骤6和7使用相同的剪裁截断 rₘₐₓ = 32,计算了相同链(步骤1)和残基(步骤2)中标记 i 和 j 之间的标记索引差的 one-hot 编码。步骤9和10在标记 i 和 j 属于不同链(步骤1)时,计算了链索引差的相同 one-hot 编码。在这种情况下,使用了一个截断值 sₘₐₓ = 2,因为 AlphaFold 试图预测包含两条不同链的复合物的结构。如果存在更多的链,则不会对标记之间的链索引差进行编码。
最后,相对于标记 i 和 j 的相对残基编号差异、相对标记索引差异和相对链索引差异的 one-hot 编码以及一个指示这些标记是否具有相同标识的掩码被连接并乘以一个权重矩阵,以生成标记 i 和 j 的相对位置编码。这些编码与上面计算的成对标记嵌入相加,从而创建新的成对输入嵌入。
Step 5: 线性层
到目前为止,除了用户定义的标记之间的键外,所有特征都已嵌入。如上所述,AlphaFold3 的用户可以通过 token_bonds 特征指定标记 i 和 j 之间的键(例如,非蛋白质/RNA/DNA 分子中的重原子)。通过将这个特征乘以一个权重矩阵,将它添加到成对嵌入中。

此步骤生成初始成对嵌入。结合步骤 2 中的单个令牌嵌入,我们可以继续讨论 AlphaFold3 的第一个模块。
Step 6:初始化
成对令牌和单个令牌嵌入最初初始化为0向量,并将在下一个周期的每一轮中更新。
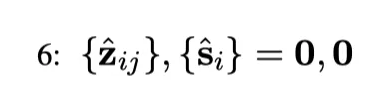
Step 7:迭代
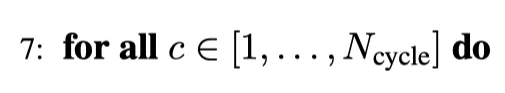
这不是一个实际的步骤,但它告诉我们以下步骤 8(线性层和层标准化)、9(TemplateEmbedder)、10(MsaModule)、11(另一个线性层和层标准化)、12(Pairformer Stack)和13(值更新)针对N _cycle = 4 次回收迭代执行。
Step 8: 对成对表示进行线性层处理

上一个循环中更新的成对标记嵌入(在第一个循环中为 0 向量)在每个新的迭代循环开始时通过层归一化和权重矩阵的乘法处理。输出结果与步骤 5 中的初始嵌入相加,以生成该循环的成对嵌入。
Step 9: 模板嵌入器
模板嵌入器使用前一步骤 8 中生成的成对嵌入和来自模板搜索的特征作为输入。模板嵌入器的主要任务是根据当前的成对嵌入值对模板中的区域进行加权注意力。更新这些成对嵌入会将焦点转移到模板结构中的“更重要”的区域

特征是从输入序列的 UniRef90 MSA 的单个蛋白质链的模板搜索中提取的。非常长的序列被裁剪为前 300 个残基。使用 hmmbuild 从 MSA 中生成隐藏马尔可夫模型(HMM),然后使用 hmmsearch 从结果中生成模板。长度小于十个残基的短模板和与查询的序列覆盖率小于 10% 或大于 95% 的模板被删除。从剩余的模板中,在推理期间使用四个,在 AlphaFold3 训练期间根据 e 值最多使用四个。序列的结构数据从 PDB70 中提取,或者如果与 PDB 数据库的相应 mmCIF 的序列不完全匹配,则在使用 KAlign 对齐后提取。总之,与 AlphaFold2 相比,AlphaFold3 中的模板搜索保持不变,除了一些细节,如模板的截止日期。
从模板的结构中提取特征,并与前一循环的成对嵌入一起输入到模板嵌入器中。

AlphaFold3 论文中提取的模板嵌入器算法。
在步骤 1-5 中,将 template_backbone_frame_mask、template_distogram、template_restype、template_pseudo_beta_mask 和 template_unit_vector 特征进行了连接。请检查上面主算法中步骤 1 中的定义。对于所有 t 个模板和包含的标记 i 和 j,相应的嵌入包含有关这些特征的所有信息 a_tij。将这些特征嵌入乘以一个权重矩阵,并在将其归一化并乘以一个权重矩阵后添加到上一步的成对嵌入中。连接的嵌入通过下面描述的 PairformerStack 的两个块,这取代了 AlphaFold2 中的 Evoformer 模块。结果通过残差连接添加到未修改的连接嵌入中,以保持在修改之前使用 Pairformer 模型之前的特征,然后进行归一化。在为所有模板结构执行此操作后,输出嵌入通过模板结构的数量进行归一化,并通过具有 ReLU 激活函数的线性层处理。
总之,模板嵌入器使用每个周期的当前成对嵌入来关注所有模板中当前最重要的区域。通过将此应用于所有模板,将结构焦点移到具有成对嵌入中较高权重的蛋白质区域的结构变化。AlphaFold2 中的模板对堆栈遵循类似的概念,即根据模板结构关注成对特征,但构建在不同的层次结构上。
Step 10:MSA模块

MSA 模块的任务是在每次循环迭代中生成一个新的 MSA 子集。MSA 模块的结构如下图所示。
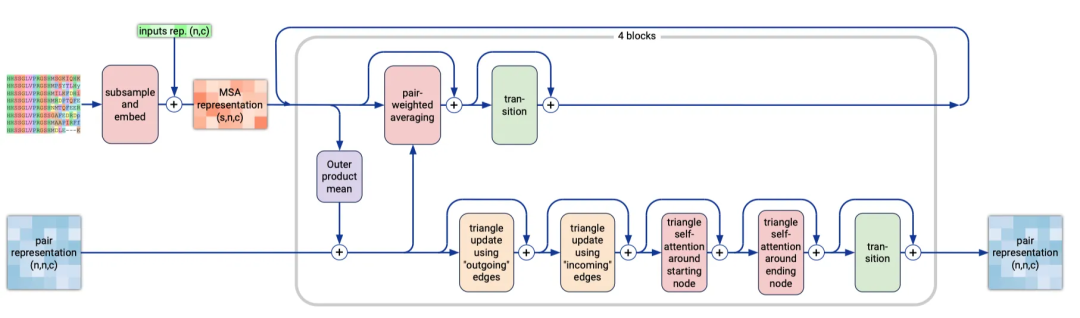
MSA 模块使用从 MSA 子集中提取的特征、模板模块输出的成对表示以及单个标记表示作为输入来计算新的成对表示。

MSA 模块包含四个块,比 AlphaFold2 的 48 个块少得多,与 AlphaFold2 中的 pair stack 块类似。然而,这并不是 MSA 模块中唯一的变化。最重要的技术差异在于 AlphaFold3 的 MSA 模块不使用逐行门控自注意力。为了理解这一点,让我们看看 MSA 是如何生成的。
AlphaFold3 中的 MSA 包含 16,384 行。第一行是查询(输入)序列。接下来的 8191 行(如果找到的对齐较少,则为更少行)是通过将 MSA 复制 n 次以用于同源复合物(其中 n 为链重复的次数)或者通过将每个链的 MSA 从左到右堆叠在一起来构建的,然后配对异源复合物的序列。剩余未配对的 MSA 序列将添加在下方,但在最多 8191 行后截断。MSA 的另一半行用原始 MSA 填充。对于遗传搜索,使用 Jackhmmer 和 HHBlits 在 UniRef90、UniProt、Uniclust30 + BFD、Reduced BFD 和 MGnify 蛋白质数据库中搜索,使用 mmseqs 在 Rfam、RNACentral 和 Nucleotide collection RNA 链数据库中搜索。
最终,MSA 矩阵的行中包含不同的 MSA 序列,而对齐的残基位于列中。在 AlphaFold2 中应用逐行门控自注意力会生成残基对的注意力权重。成对嵌入作为额外的偏置被包含在内。在 AlphaFold3 中,注意力是针对每一行独立进行的。这也意味着注意力权重是从成对嵌入生成的。换句话说,这种变化更加关注成对表示,而不是残基对之间的 MSA。然而,这些成对表示包含来自 MSA 行中的残基对的一些信息,这些信息来自输入嵌入。
这种变化的影响是什么呢?嗯,MSA 行注意力关注同一序列的不同残基对。这由导致这些标记之间相互作用的特征表示。这些特征应该被编码到这些残基的成对表示中,因此让完整的信息通过成对表示是有意义的。

首先,所有 MSA 特征与 has_deletion 和 deletion_value 特征进行连接,这些特征告诉我们在 MSA 中是否存在删除以及删除数量(请参见主算法中的步骤 1 的解释)。然后,随机选择一定数量的 MSA,并将其所有标记的对应表示与权重矩阵相乘。通过残差连接,将输入的单个标记表示与另一个权重矩阵相乘后的表示相加。这些表示进入 MSA 块。对这些表示应用一个由层归一化、与权重矩阵相乘、平均化、展平和线性层组成的 OuterProductMean 层。
结果进入 MSA 堆栈和成对堆栈。MSA 堆栈对这些嵌入进行了成对加权平均,然后在 MSA 行上进行了 0.15 的 dropout。这确保了在每次新的执行中都会包含新的 MSA 子集在嵌入中。最后,应用过渡层进行归一化、线性化和 SwiGLU 激活,以生成用于下一个 MSA 堆栈或与成对表示的外积的输入。
成对堆栈包含两个三角形乘法、两个三角形自注意力和一个过渡层。这些三角形更新是在 AlphaFold2 中引入的。它们确保了标记之间(在 AlphaFold2 中仅限于氨基酸)的成对表示可以表示为带有距离三角不等式等约束的 3D 结构。简而言之,更新操作以三角形边的形式排列,每个边有三个节点。缺失的节点通过轴向注意力添加,在三角形乘法更新步骤和最终的注意力步骤中进行更新。由于这个成对堆栈步骤与 AlphaFold2 中的相同,我建议参考 AlphaFold2 论文以了解有关三角形层的更多细节。
经过 MSA 模块的四个块后,其最后成对堆栈的成对表示输出通过 Pairformer 堆栈。
Step 11:单一表示上的线性层


模板和MSA模块提取了关于标记之间关系的信息,由成对表示表示。因此,这些模块只修改了成对表示。这些模块没有更新单个标记的表示。单个和成对表示在Pairformer中使用,并在不同的迭代中进行重复利用。这需要使用上一个迭代的输出来更新单个表示。通过层归一化和与权重矩阵相乘,在上一个迭代的表示或第一个迭代周期中应用于0向量后,将其输出添加到来自输入嵌入器(步骤2)的初始单个标记嵌入中,以生成直接用于下一个Pairformer的新输入,而无需进一步修改。这一步与步骤8中的对成对表示的操作相呼应。
Step 12: 配对
AlphaFold3中的Pairformer模块类似于AlphaFold2中的Evoformer模块。它使用来自上一次迭代的单个表示,加上来自输入嵌入器的单个表示以及来自MSA模块的成对表示作为输入。与AlphaFold2中的Evoformer模块相比,它不使用特定的MSA表示输入。MSA的信息已经包含在成对表示中(参见步骤10)。

在AlphaFold2中,Evoformer使用MSA的子集作为表示,并应用列注意力。但在Pairformer模块中这是不需要的。因为只使用单个表示,所以只需对单个序列应用基于行的注意力,使用来自单个表示的嵌入。下面是Pairformer堆栈的算法:

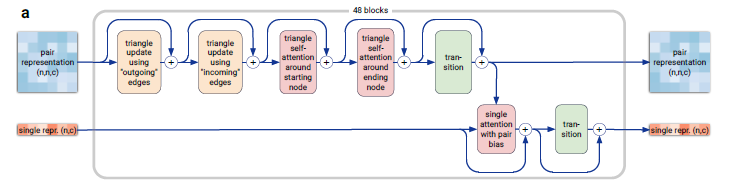
可以直接看出,这些步骤与MSA模块中执行的步骤相似。与MSA模块一样,成对表示经过两个三角形更新、两个三角形自注意力和一个带有SwiGLU激活函数的过渡层。在所有层中,都应用了残差连接。单个表示不影响成对表示。直观地说,这意味着表示一对标记之间交互的特征(例如一对氨基酸或核苷酸)受成对特征的影响,但不受单个标记特征的影响。然而,表示与其他标记的交互的特征会影响单个标记的特征。这由单个注意力层定义,该层通过该块的成对表示传递单个表示。该层具有来自单个表示和该块的成对表示输出的输入。它使用16个头在单个表示上应用多头自注意力,使用成对表示作为偏置。输出通过另一个过渡层,两个层都应用了残差连接。成对和单个堆栈的过渡层的输出作为下一个块的输入。与AlphaFold2类似,应用了48个块,使Pairformer成为AlphaFold3架构中的主要处理模块。
Step 13:值更新和回收
Pairformer模块的单个表示和成对表示输出要么是下一个循环的输入,要么是下一个Diffusion和ConfidenceHead模块的输入。
Step 14:迭代结束
在AlphaFold3的流程中,Pairformer模块标志着一次迭代的结束。输出被用作下一次迭代的输入。通过不断更新单个和成对表示,并使用MSA的新子集,进行四个这样的循环。在所有循环结束时,学习到了单个标记和标记对的表示,可以在随后的扩散模块中用于结构生成。
Step 15:扩散模块
扩散模型生成结构的分布,取代了AlphaFold2中的结构模块。Sample Diffusion从Pairformer模块中获取单个和成对表示,从InputEmbedder获取特征和单个输入嵌入,并生成原子和/或标记的坐标。

由于这个模块是AlphaFold3中的新模块,让我们详细看一下SampleDiffusion算法,以了解它的具体实现。

首先,从原点周围的高斯分布中生成初始位置。然后,应用不同的噪声。对于所有这些噪声,原子的位置被居中和旋转,然后加上嘈杂的位移。这些嘈杂的位置进入扩散模块。从该模块估计得到的去噪位置被用来更新原子位置。这个过程对T种不同的噪声进行重复,并对原子的最终位置进行抽样。
下面是扩散模块,它用于去噪原子位置。
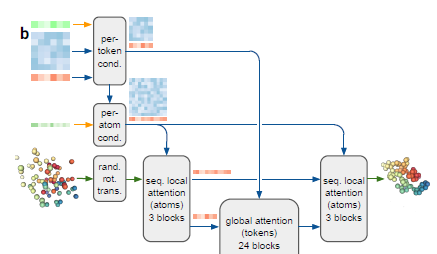
Diffusion module.
让我们逐步查看扩散模型的算法。

首先,使用先前步骤中计算的输入条件化扩散。将成对嵌入与输入特征的相对位置编码连接起来,进行归一化处理,乘以权重矩阵,并通过两个具有SwiGLU激活函数和残差连接的转换层。来自Pairformer输出和输入嵌入器的单个表示也被连接起来,进行归一化处理,乘以权重矩阵。但接着使用了傅立叶嵌入和余弦函数。在我之前关于变换器的文本中,我解释了为什么要使用三角函数来进行位置嵌入。嵌入再次归一化,乘以权重矩阵,并通过两个具有残差连接的转换层。

第二步,将输入位置进行缩放,生成无量纲向量,其方差接近单位。

第三步,重新缩放后的位置、特征、单体嵌入和成对嵌入经过了主算法中描述的原子注意力编码器。需要注意的是,与主算法中的第一步不同,这里的输入是噪声位置。这意味着编码器的单体和成对嵌入以及噪声位置会通过线性层和残差连接进行更新(参见上述描述)。此外,编码器使用多头自注意力生成新的位置 qₗ。

Atom Transformer使用沿着成对矩阵对角线的矩形块进行序列中的局部原子注意力。这确保只有附近的原子被关注。使用这些注意力掩码,调用了一个由24个块组成的Diffusion Transformer,它使用自适应层归一化进行单体调整,并使用对数偏置进行成对调整。我会写一篇关于扩散Transformer的单独文章,并在这里链接这篇文章,但是这个Diffusion Transformer在这一步中对原子级别的结构进行了去噪处理,这就是其神奇之处。
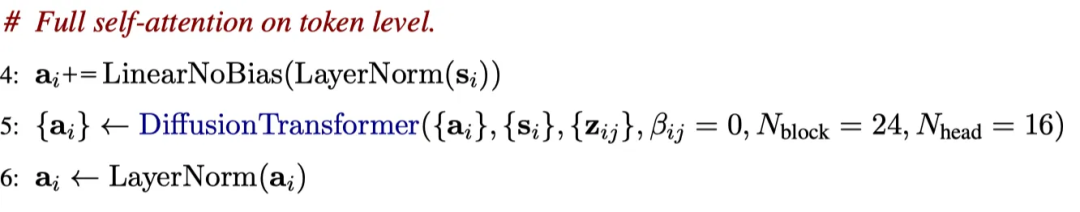
在对原子进行调整后,使用类似结构的扩散Transformer在令牌级别上进行自注意力操作。最终的令牌表示被归一化。


接下来,令牌嵌入被用于原子注意力解码器。该解码器首先将令牌激活转换为原子表示,然后使用上述的原子Transformer生成新的位置。

AlphaFold3 中,通过将更新后的位置与原始的噪声原子位置相加,生成了新的去噪原子位置。随后,这些更新后的位置被重新缩放为几乎具有单位方差的无量纲向量。
总之,AlphaFold3 中的扩散模型取代了 AlphaFold2 中的结构模型,从噪声的原子坐标中预测真实的原子坐标,采用了标准的扩散模型而没有旋转框架和等变性。不同的噪声级别用于在局部水平(低噪声)和较大长度尺度(高噪声)上学习蛋白质结构。扩散模型生成了一系列结构的分布,从而使得能够预测准确的侧链几何结构。然而,这样的分布可能会在非结构化区域生成物理上合理的结构。为了确保 AlphaFold3 对预测的去噪结构有信心,扩散模型的输出进入了置信度模块的输入。
Step 16:置信度头模块
置信度模块使用来自输入嵌入器的单体输入、来自 Pairformer 模块最后一次迭代的单体和对表示以及扩散模块预测的坐标,来估计结构预测的置信水平。这一点很重要,例如用于过滤在非结构化区域中的合理结构。


置信度头模块开始通过将两个标记 i 和 j 的单体嵌入器乘以权重矩阵来预测标记位置的置信度。在添加两个输出之后,将结果加到两个标记的对嵌入中。基于它们在一定距离阈值内的事实,两个标记的代表原子之间的距离进行了 one-hot 编码。在与另一个权重矩阵相乘后,这些关于距离的信息也包含在对嵌入中。换句话说,在第 3 步后的对嵌入中包含了代表原子是否在一定距离阈值内以及来自输入嵌入器的单体标记嵌入信息。这些表示通过 4 个 Pairformer 模块块进行处理,具有残差连接,并相应地进行了更新。更新后的单体和对嵌入与权重矩阵相乘。然后,使用 softmax 函数根据四个度量指标来预测标记或原子是否接近地面实况:单个原子上的预测局部距离差异测试(pLDDT),成对原子-原子对齐误差(PAE),成对原子-原子距离上的预测距离误差(PDE)以及与实验确定的实况相匹配的分辨率。
Step 17:Distogram Head
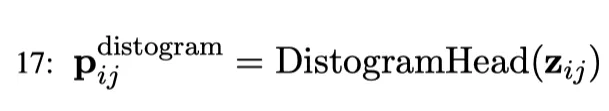
除了前面步骤中的置信度度量之外,AlphaFold3 还计算了预测的所有标记对之间的分段距离直方图。为了计算包含多个原子的标记的直方图,选择一个代表性原子。对于所有氨基酸残基,选择 CB 原子,对于甘氨酸,选择 CA 原子,对于嘧啶类选择 C4 原子,对于嘌呤类选择 C2 原子。
直方图的预测首先对对嵌入进行对称处理,例如,计算 z_ij + z_ji。然后,将结果线性投射到等宽的范围从 2 Å 到 22 Å 的箱中。接下来使用 softmax 函数获得概率 p_ij。
Step 18:预测模块
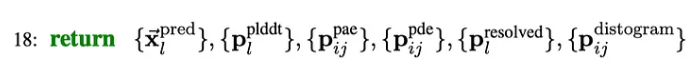
AlphaFold3 返回生成结构的坐标预测、直方图概率以及基于 pLDDT 分数、PAE 分数、PDE 分数和与实验确定的结构匹配的分数的置信度值。
总结
类似于之前的AlphaFold版本,AlphaFold3是蛋白质结构预测的最新SOTA方法。它在单链预测方面略优于AlphaFold2,但AlphaFold3的主要优势在于准确预测复合物。特别值得注意的是,与AlphaFold-multimer v2.3相比,AlphaFold3在抗体-抗原界面预测方面有了显著改进。
这些进步是基于AlphaFold3中的新架构。虽然AlphaFold3是在AlphaFold2的基础上构建的,但与AlphaFold2和AlphaFold1之间的架构差异相比,AlphaFold3与AlphaFold2之间的差异要小得多,但仍然有一些重大修改。它们包括:
1. AlphaFold2中的结构模块被AlphaFold3中的扩散模块所取代。值得注意的是,在不引入不变或等变约束的情况下改进了预测效果。
2. AlphaFold3将词汇表从代表蛋白质的氨基酸扩展到代表RNA和DNA的核苷酸以及代表所有化学分子(包括配体)的重原子。
3. AlphaFold3中的MSA模块比AlphaFold2小得多(仅有四个块),并且已从新的Pairformer模块中删除。
4. AlphaFold3中的Pairformer模块取代了AlphaFold2中的Evoformer模块。虽然两个模块的结构相似,除了删除了MSA模块(见上一点),但Pairformer模块还有一些内部的变化。例如,信息从对表示到单表示的流动,但反之则不然。
5. 大多数但不是所有的ReLU激活函数在AlphaFold2中被SwiGLU激活函数所替换,以提高性能。
其中一些修改是为了准确预测蛋白质复合物(例如,对对表示的关注),而其他修改则是对机器学习(ML)方法的更新,这些方法自AlphaFold2发布以来已经出现并得到普及(例如,扩散模型或SwiGLU激活函数)。有趣的是看到类似OpenFold这样的开源模型如何利用这些模型来提高其准确性。我们可以预期很快会出现几种新的复合物预测工具,其中大多数将使用在AlphaFold3中实施的方法。
编译 | 王建民
参考资料
- Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature (2024). https://doi.org/10.1038/s41586-024-07487-w
- https://medium.com/@falk_hoffmann/alphafold3-and-its-improvements-in-comparison-to-alphafold2-96815ffbb044
- Bertoline, Letícia MF, Angélica N. Lima, Jose E. Krieger, and Samantha K. Teixeira. "Before and after AlphaFold2: An overview of protein structure prediction." Frontiers in Bioinformatics 3 (2023): 1120370.