序列比对:双序列比对与BLAST
序列比对
当研究一条DNA或蛋白质序列时,主要关注的是其包含的遗传信息;当研究两条或多条DNA或蛋白质序列时,则主要关注不同序列之间的差别与联系。在生物信息学中,对生物大分子的序列比对是非常基本的工作。
前两篇文章DNA与蛋白质的序列比对原理和替换计分矩阵介绍了序列相似性和距离的定量分析的基础,即序列对齐与匹配/非匹配字符不同权重的打分。具体说来,序列比对就是寻找使两个或多个序列产生最大相似性得分或最小编辑距离的序列对齐方式的过程,或者理解为找出两个序列最长公共子序列(longest common subsequence,LCS)的过程。今天首先为大家介绍双序列比对,也即两条序列(或者多条序列两两之间)进行的比对,常用于同源分析、蛋白质结构推断、相似片段搜寻与数据库比对检索、基因注释等。
双序列比对算法
⑴基本算法(LCS算法)
序列比对实质上是一个路径寻找问题,若有序列v=ATGTTAT和w=ATCGTAC两个短序列,其比对过程可以用下图表示:

从(0,0)到(7,7),每穿过一个顶点相当于成功匹配一个碱基,向右、向下穿过边则相当于在w、v中插入一个空格。寻找LCS也即寻找尽可能多的穿过定点的路径(最长路径),最简单的办法是遍历从从(0,0)到(7,7)所有的路径并计算最长的一条。显然,当序列较长的时候,计算量将变得十分庞大。
⑵动态规划算法
动态规划(dynamic programming)算法是目前最基本的序列比对算法。动态规划中的规划(programming)一词来自于数学规划(mathematical programming),Saul Needleman和Christian Wunsch两人首先将其用于两条序列的全局比对,其算法后被称为NeedlemanWunsch算法,其主要思想是把多阶段过程转化为一系列单阶段问题,利用各阶段之间的关系,逐个求解。例如对于如下假定的序列:
I. a, b是使用某一字符集∑的序列(DNA或蛋白质序列),其中m为a的长度,n为b的长度;
II. S(i, j)是按照某替换计分矩阵得到的a序列的前i个字符a[1...i]与b序列的前j个字符b[1...j]的最大相似性得分;
III. w(c, d)是某位置的字符c和d按照替换计分矩阵计算的得分。
那么对于a与b的子序列a[1...i]与b[1...j]其得分有如下规律:
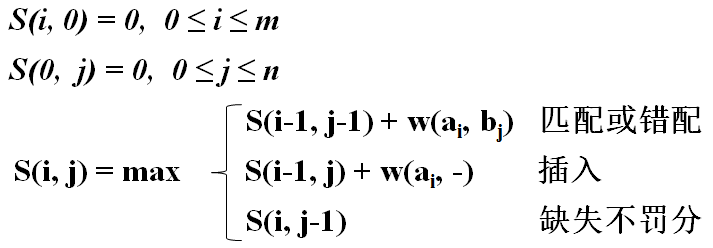
例如,对于序列a=ACACACTA,序列b=AGCACACA,计分规则为:w(匹配)=+2;w(ai, -)=w(-, bj)=w(失配)=-1,也即匹配得分+2,缺失、插入、失配得分为-1,那么根据该规则可以获得替换计分矩阵,并根据上面的规则进一步得到关于S(i, j)的得分矩阵:

为了得到最佳比对,仅需从最大得分处开始回溯得分矩阵(如箭头所示),直到起点(0, 0),回溯过程中可能遇到多个路径,选择最大得分作为最优路径,即是最优解。双序列比对所需要的计算时间和内存空间与这两个序列的长度有关,或者说正比于这两个序列长度的乘积,用O(mn)表示。
双序列比对工具
常用的双序列比对工具有BLAST、FASTA、diamond等。其中最常用的BLAST(Basic Local Alignment Search Tool)是一套在蛋白质数据库或DNA数据库中进行相似性比较搜索的分析工具。BLAST采用一种局部的算法获得两个序列中具有相似性的序列,能迅速与公开数据库进行相似性序列比较,BLAST结果中的得分是对一种对相似性的统计说明。
BLAST是免费软件,除了在线比对检索服务,也可以从NCBI文件服务器上下载获得本地版本。本地运行须有BLAST格式的数据库,常用的有NCBI的DNA数据库(nt)和蛋白质数据库(nr),均可从NCBI官网下载。此外,也可以使用任意数据库序列文件通过BLAST提供的格式转换工具由其他格式序列文件转换而得到,如下所示:
软件下载地址:ftp://ftp.ncbi.nlm.nih.gov/blast/executables/LATEST/
makeblastdb -in db.fasta -input_type fasta -dbtype prot -parse_seqids -out dbname
参数说明:
-in:待格式化的序列文件
-input_type:输入序列文件的格式,默认为fasta
-title:输出数据库的title,默认使用-in参数文件名的前缀
-dbtype:数据库类型,蛋白质prot或核酸nucl
-out:输出数据库的文件名前缀,默认使用-in参数文件名的前缀
-parse_seqids:解析输入序列的id,一般不用设置
-max_file_sz:文件最大字节数,默认1000000000B
-taxid_map:指定gi到物种ID的映射文件BLAST实际上是综合的一组程序,不仅用于对核酸序列数据库和蛋白质序列数据库进行搜索,而且可以将查询序列翻译为蛋白质后再进行搜索,进行序列比对时,需要根据要比对的序列类型选择软件工具以及数据库,如下所示:

Blast算法基于动态规划算法开发。具体原理是,首先去掉输入序列(要查询的序列,query sequence)中的低复杂度或重复区域,将输入序列划分成K-letter words(类似于k-mers),一般来说蛋白质word size为3,核酸为11,K越小灵敏度越高,计算速度越慢。然后在数据库中搜寻能比对到的序列,称为种子序列(seeding),在数据库中定位这些种子序列,K-letter words比对上一次称为一个hit,利用打分矩阵左右延伸寻找到hit cluster,直到打分低于某个阈值,最终得到高分片段(high-scoring segment pair,HSP)。
延伸条件为:两个hits没有overlap,两个hit在输入序列与输出序列相对位置相符,两个hit在同一个窗口(window)内。最终对比对结果也即score足够高的HSPs进行显著性分析,将输入序列与一系列长度相等的随机序列进行比对,其分值符合Gumbel极值分布,在这种随机情况下,获得比当前比对得分高的随机序列条数的期望称为expectation value(e-value),e-value越小说明比对结果越显著。该工具使用方法如下所示:
blastp -query test.faa -out nr_blast.out -db nr -outfmt 6 -evalue 1e-5 -num_threads 20
参数说明(可以使用blastp -help查看):
-query:输入文件的文件名,即基因组预测的基因蛋白序列
-db:Blast数据库的名字及其路径
-out:输出文件的文件名
-evalue:设置输出结果的e-value值,大于此值的比对被舍弃,默认为10
-word_size:K-letter words,应大于2,默认为3
-matrix:计分矩阵名字,默认为BLOSUM62
-threshold:最小的K-letter words比对得分,应大于0
-outfmt:输出文件格式,总共有19种格式,默认为0为pairwise,6是tabular格式对应BLAST的m8格式,如下所示:
0 = Pairwise,
1 = Query-anchored showing identities,
2 = Query-anchored no identities,
3 = Flat query-anchored showing identities,
4 = Flat query-anchored no identities,
5 = BLAST XML,
6 = Tabular,
7 = Tabular with comment lines,
8 = Seqalign (Text ASN.1),
9 = Seqalign (Binary ASN.1),
10 = Comma-separated values,
11 = BLAST archive (ASN.1),
12 = Seqalign (JSON),
13 = Multiple-file BLAST JSON,
14 = Multiple-file BLAST XML2,
15 = Single-file BLAST JSON,
16 = Single-file BLAST XML2,
18 = Organism Report
-num_descriptions:对于每个输入序列,在结果中显示的高分比对结果的描述数目,不适合outfmt大于4的情况,默认为500
-num_alignments:对于每个输入序列,在结果中显示的高分比对结果的详细比对情况数目,默认为250
-line_length:结果中详细比对情况的行的长度,不适合outfmt大于4的情况默认为60
-max_target_seqs:输出的最大比对上的subject序列数目
-html:是否生成HTML格式的结果
-seg:是否使用SEG过滤输入序列,可选'yes'、'window locut hicut'、'no',默认为'no'
-soft_masking:标记过滤掉的序列为soft masks,默认'false'
-lcase_masking:使用小写字母标记过滤掉的序列
-qcov_hsp_perc:小数值,每个HSP在查询序列的覆盖比例阈值
-max_hsps:对于每个查询序列,每条对象序列subject sequence最大HSP数
-window_size:延伸时的window大小,默认为0也即使用1-hit算法
-ungapped:延伸时不容许有gap
-num_threads:程序运行所使用的核数,默认为1需要注意的是,其中对于选项6、7和10,可以有以下详细选项(不同版本稍有不同):
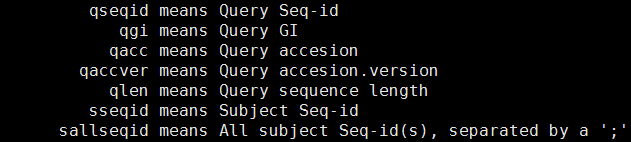
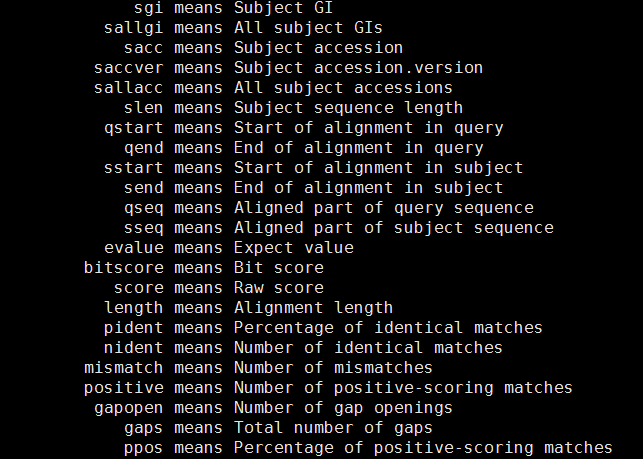
对于这三个选项,如果不设置详细条目,默认包含qacc sacc pident length mismatch gapopen qstart qend sstart send evalue bitscore,也即如下两种设置是等效的:
blastp -outfmt 6
blastp -outfmt '6 qacc sacc pident length mismatch gapopen qstart qend sstart send evalue bitscore'还有一款常用的工具DIAMOND(Double Index Alignment of Next-generation sequencing Data),该工具为基于BLAST的快速序列检索比对工具,但目前仅支持blastp、blastx,也即使用蛋白质或核酸序列在蛋白质数据库中进行比对检索。与BLAST一样,Diamond也需要根据数据库序列制作格式化的序列文件,如下所示:
软件下载地址:https://github.com/bbuchfink/diamond
diamond makedb --in nr.faa -d nr -p 10
该命令会生成以.dmnd为后缀的库文件。
参数说明:
--in:输入的数据库序列文件(FASTA格式)
-p:程序运行使用的核数
-d:输出结果的文件名前缀数据库建成后,即可对目标序列进行比对检索,其使用方法与BLAST类似。。
END
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2022-07-01,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

