跟着Nature Biotechnology学作图:R语言pca分析并使用ggplot2可视化结果
跟着Nature Biotechnology学作图:R语言pca分析并使用ggplot2可视化结果
用户7010445
发布于 2023-01-06 19:42:34
发布于 2023-01-06 19:42:34
论文
Removing unwanted variation from large-scale RNA sequencing data with PRPS
https://www.nature.com/articles/s41587-022-01440-w#data-availability
数据链接
https://zenodo.org/record/6459560#.Y2D2NHZBzid
https://zenodo.org/record/6392171#.Y2D2SXZBzid
代码链接
https://github.com/RMolania/TCGA_PanCancer_UnwantedVariation
今天推文重复的图没有出现在论文中,是论文中提供的代码里的一个图
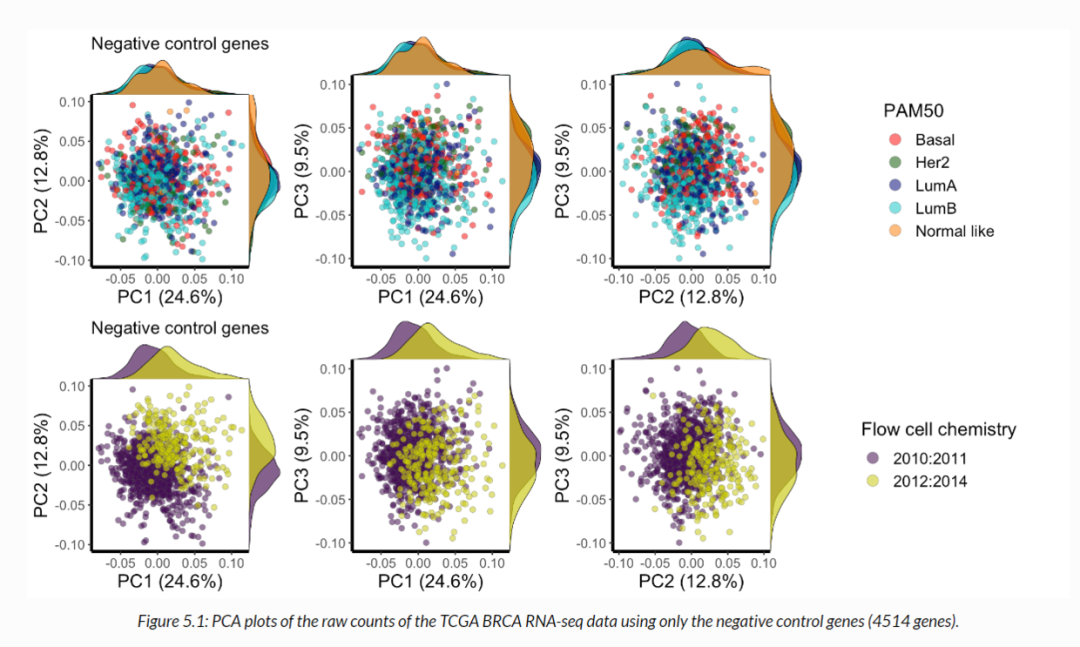
image.png
但是没有能够重复出来论文中用到的作图数据,所以这里用R语言自带的鸢尾花数据集来演示
首先是论文中提供的两个自定义函数,一个是用来做主成分分析的pca,
.pca <- function(data, is.log) {
if (is.log)
data <- data
else
data <- log2(data + 1)
svd <- base::svd(scale(
x = t(data),
center = TRUE,
scale = FALSE
))
percent <- svd$d ^ 2 / sum(svd$d ^ 2) * 100
percent <-
sapply(seq_along(percent),
function(i) {
round(percent[i], 1)
})
return(list(
sing.val = svd,
variation = percent))
}
一个是用来作图展示结果的 用到了ggplot2 ggpubr 和 cowplot 包
.scatter.density.pc <- function(
pcs,
pc.var,
group.name,
group,
color,
strokeSize,
pointSize,
strokeColor,
alpha,
title
){
pair.pcs <- utils::combn(ncol(pcs), 2)
pList <- list()
for(i in 1:ncol(pair.pcs)){
if(i == 1){
x <- pair.pcs[1,i]
y <- pair.pcs[2,i]
p <- ggplot(mapping = aes(
x = pcs[,x],
y = pcs[,y],
fill = group)) +
xlab(paste0('PC', x, ' (', pc.var[x], '%)')) +
ylab(paste0('PC', y, ' (', pc.var[y], '%)')) +
geom_point(
aes(fill = group),
pch = 21,
color = strokeColor,
stroke = strokeSize,
size = pointSize,
alpha = alpha) +
scale_x_continuous(breaks = scales::pretty_breaks(n = 5)) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5)) +
ggtitle(title) +
theme(
legend.position = "right",
panel.background = element_blank(),
axis.line = element_line(colour = "black", size = 1.1),
legend.background = element_blank(),
legend.text = element_text(size = 12),
legend.title = element_text(size = 14),
legend.key = element_blank(),
axis.text.x = element_text(size = 10),
axis.text.y = element_text(size = 10),
axis.title.x = element_text(size = 14),
axis.title.y = element_text(size = 14)) +
guides(fill = guide_legend(override.aes = list(size = 4))) +
scale_fill_manual(name = group.name, values = color)
le <- ggpubr::get_legend(p)
}else{
x <- pair.pcs[1,i]
y <- pair.pcs[2,i]
p <- ggplot(mapping = aes(
x = pcs[,x],
y = pcs[,y],
fill = group)) +
xlab(paste0('PC', x, ' (',pc.var[x], '%)')) +
ylab(paste0('PC', y, ' (',pc.var[y], '%)')) +
geom_point(
aes(fill = group),
pch = 21,
color = strokeColor,
stroke = strokeSize,
size = pointSize,
alpha = alpha) +
scale_x_continuous(breaks = scales::pretty_breaks(n = 5)) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5)) +
theme(
panel.background = element_blank(),
axis.line = element_line(colour = "black", size = 1.1),
legend.position = "none",
axis.text.x = element_text(size = 10),
axis.text.y = element_text(size = 10),
axis.title.x = element_text(size = 14),
axis.title.y = element_text(size = 14)) +
scale_fill_manual(values = color, name = group.name)
}
p <- p + theme(legend.position = "none")
xdens <- cowplot::axis_canvas(p, axis = "x")+
geom_density(
mapping = aes(
x = pcs[,x],
fill = group),
alpha = 0.7,
size = 0.2
) +
theme(legend.position = "none") +
scale_fill_manual(values = color)
ydens <- cowplot::axis_canvas(
p,
axis = "y",
coord_flip = TRUE) +
geom_density(
mapping = aes(
x = pcs[,y],
fill = group),
alpha = 0.7,
size = 0.2) +
theme(legend.position = "none") +
scale_fill_manual(name = group.name, values = color) +
coord_flip()
p1 <- insert_xaxis_grob(
p,
xdens,
grid::unit(.2, "null"),
position = "top"
)
p2 <- insert_yaxis_grob(
p1,
ydens,
grid::unit(.2, "null"),
position = "right"
)
pList[[i]] <- ggdraw(p2)
}
pList[[i+1]] <- le
return(pList)
}
这两个自定义函数在函数名前都加了一个点,暂时不知道加这个点和不加有什么区别,将这两个函数放到一个文件里
source("pca_and_ggplot2.R")
library(ggplot2)
library(ggpubr)
library(cowplot)
pca.ncg<-.pca(data = iris[,1:4],
is.log = FALSE)
.scatter.density.pc(pcs = pca.ncg$sing.val$v[, 1:3],
pc.var = pca.ncg$variation,
strokeColor = 'gray30',
strokeSize = .2,
pointSize = 2,
alpha = .6,
title = "A",
group.name = "B",
group=iris$Species,
color=c("red","blue","green")) -> p
do.call(
gridExtra::grid.arrange,
c(p,ncol=4))

这里自定义的pca结果可视化函数参数还挺多的,这里就不逐个介绍了,争取抽时间录制成视频介绍,敬请期待
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2022-11-09,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

