GEO数据读取-笔记分享
原创这里面所有的内容都来自我整理的小洁老师的讲解和B站视频里的知识
芯片基础知识打卡http://www.biotrainee.com/thread-992-1-1.html
GEO数据库一览
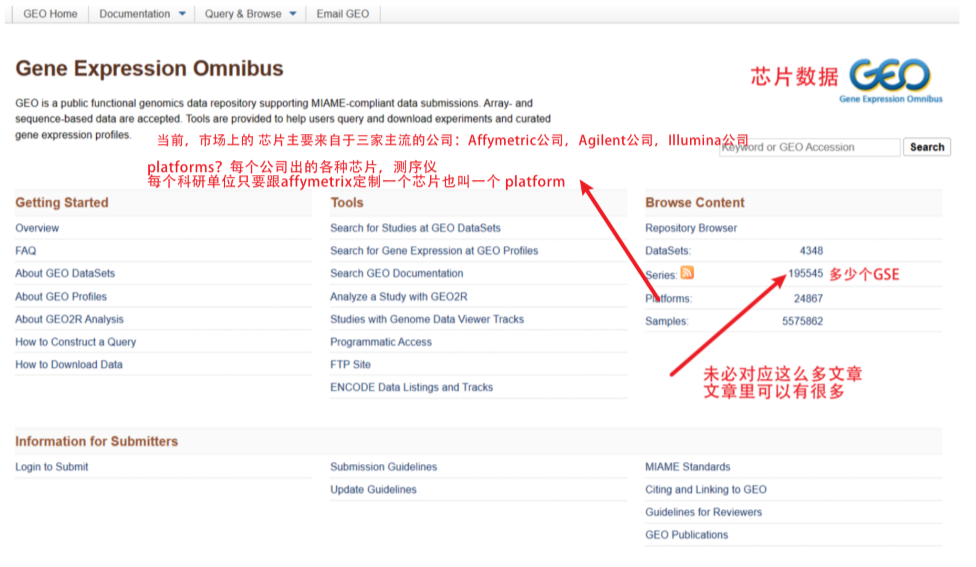
GPL GSM GSE 的区别?
• 1.想从文章里找到作者用的数据集编号,编号开头是? • GSE
• 2.某公司开发的一款芯片产品,他在GEO数据库中的编 • GPL
号开头是?
• 3.表达矩阵的行名是探针名,列名是样本名,所以列名
的编号开头是? •GSM
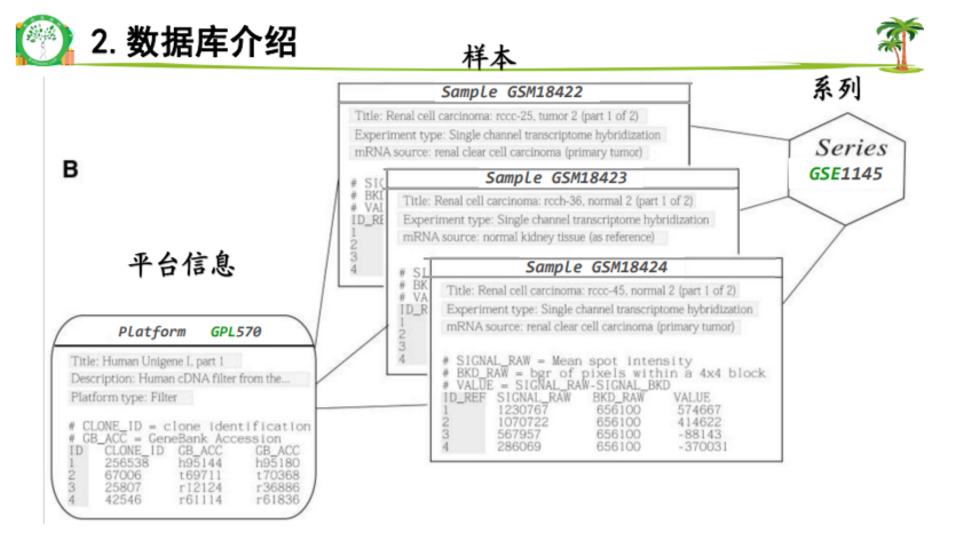
知识点讲解:芯片
高通量、全基因组的DNA芯片已经成为生物领域十分有用的工具。然而,芯片实验产生的数据量日益增长,由于不同的分析方法,会得出不同结论,因而分析起着关键作用。
根据芯片的使用目的,一张芯片可能包含数十、数百甚至数十万的不同序列。被排列成矩阵的DNA片段通常称为探针,而样本RNA则被成为靶标。
基本的芯片实验中,样本mRNA首先被反转录成cDNA(在过程中同时被荧光标记),后与芯片上的核酸探针混合,互补杂交的cDNA就结合到芯片上,而未被杂交的样本被洗脱掉。芯片被一个荧光扫描仪扫描后,芯片上某个位置探针结合上了样本中互补的核酸,就在该位置显出了一个荧光点,此位置提示基因的身份,而荧光强度则提示了原始样本中该mRNA水平的高低。芯片技术不只用于检测基因表达,也可以用于检测单核苷酸多态性等。
在芯片技术中有两种基本方法:单染色技术和双染色技术。
单染色技术是将一个样本经一种荧光标记后单独杂交的一张芯片上,是目前使用最多的方法。将一个样本单独与一张芯片杂交,可以方便简单地在多张芯片之间进行比较。产生的芯片数据为单通道信号数据,这种方法产生的数据变异大,需要通过重复实验来减少误差。
双染色技术是把两个样本用不同荧光标记后一起杂交到同一张芯片上。用于检测两种不同条件下基因表达的差异情况,如疾病组织和正常组织(往往多个正常组织DNA混合在一起,作为”pool“样本);处理组与对照组。两个样本(如处理与对照)被两种不同荧光标记。一个样本的cDNA用Cy5(一种显示为红色染料)标记,另一个样本用Cy3(一种显示为绿色的染料)标记。这两种荧光标记的样本混合后与芯片上的探针竞争杂交。
这样产生的芯片数据为双通道信号数据。这种双通道信号数据便于两样本间的直接比较,有助于减少数据变异性,提高组间差异表达分析的准确性,同时减少了芯片的使用量,节约了成本。但由于使用这种技术已经确定好了实验设计,就无法与其他样本进行比较了。
当前,市场上芯片主要来自三家公司:Affymetric公司、Agilent公司和Illumina公司。
基因芯片分析一般对硬件要求不高,普通的计算机就能运行,但如果处理较多的数据量时,建议提高内存,一般拥有16g内存和i7的处理器基本就能快速运行所有分析了。目前基因芯片的分析工具很多,但各有优缺点。
根据难易程度推荐以下三款软件和工具。
- GeneSpring 优点:互动式的视窗操作界面,傻瓜式操作,功能强大,拥有超过4400篇的高水平参考文献的引用,表达谱数据分析的金标准。缺点:商业软件收费,操作繁琐,功能拓展性差。如同SPSS一样,适用于零基础。
- BRB-Array 优点:基于excel的分析工具,自动调用R包,功能强大,拓展性强,操作简单,免费使用。缺点:专业性强,格式要求高,稍有不符就报错。适用于有一定专业基础。
- R-Bioconductor 优点:R语言,生信必学的分析工具,强大的统计分析和作图工具,集合了几乎所有最新的分析算法和工具包,免费下载使用。缺点:需要有一定计算机编程能力。
一般来说要比较和整合不同实验室和不同实验的数据是比较困难的。因此,科学家成立了一个联盟(MGED学会)来规范化芯片数据的输出和注释,促进数据共享和统一数据库的建立。指定的标准化规则称为MIAME,权威期刊一般只接受遵循MIAME规则的芯片数据论文。NCBI的GEO和EBI的ArrayExpress是目前最大的公开资源数据库,用于存储和发布与MIAME相容的芯片数据。
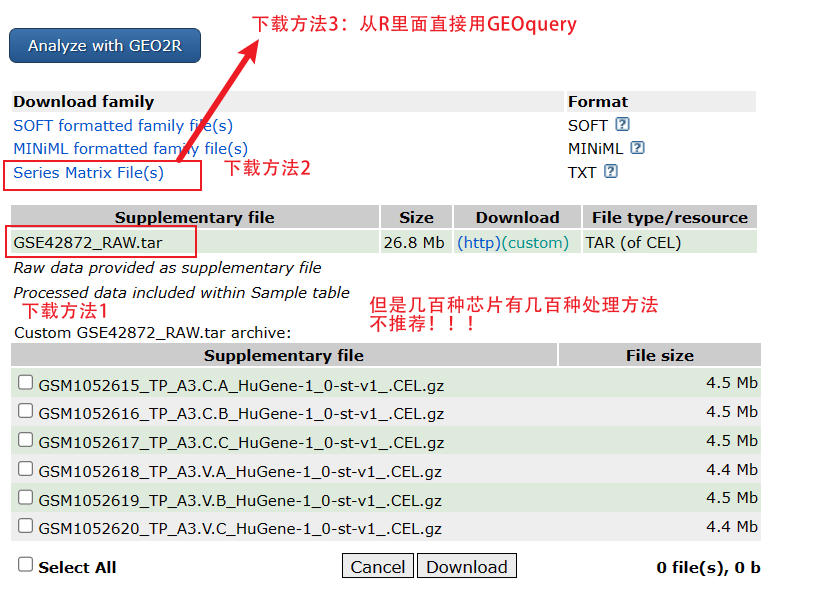
GEO数据下载:
一般采用方法3
什么是正常的数据呢?
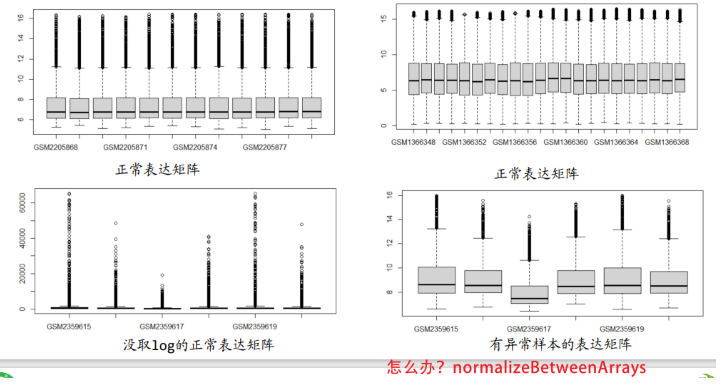
数据处理过程中应该注意:

代码如下:
1.数据下载与整理
if(! require("GEOquery")) BiocManager::install("GEOquery",update=F,ask=F)
library(GEOquery)
library(AnnoProbe)
library(tidyverse)
library(estimate)
library(stringr)
Sys.setenv("VROOM_CONNECTION_SIZE"=131072*2)
##下载并且了解你的数据
gse_number = "GSE58294"
#eSet=getGEO('GSE58294',destdir = '.',getGPL = F)
eSet <- geoChina(gse_number, destdir = '.')#比较新的下载不了,比较快
eSet = eSet[[1]]
exp=exprs(eSet)
dim(exp)
#54675 92
max(exp)
# 7.85433
pd=pData(eSet)
p = identical(rownames(pd),colnames(exp));p
if(!p) exp = exp[,match(rownames(pd),colnames(exp))]
#(4)提取芯片平台编号
gpl_number <- eSet@annotation;gpl_number
#[1] "GPL570"2.group_ids ,探针注释
# 生成Group向量的三种常规方法,三选一,选谁就把第几个逻辑值写成T,另外两个为F。如果三种办法都不适用,可以继续往后写else if
if(F){
# 1.Group
# 第一种方法,有现成的可以用来分组的列
Group = pd$`disease state:ch1`
}else if(F){
# 第二种方法,自己生成
Group = c(rep("control",times=10),
rep("stroke",times=29))
Group = rep(c("RA","control"),times = c(13,9))
}else if(T){
# 第三种方法,使用字符串处理的函数获取分组
Group=ifelse(str_detect(pd$source_name_ch1,"control"),
"control",
"RA")
}
Group=c(rep('CT',23),rep('IS',69))
Group=factor(Group,levels = c('CT','IS'))
#对照组在前,处理组在后 前面的是向量,只要他和表达矩阵是一一对应的就行
#我们只要管levels的顺序
Group = factor(Group,levels = c("control","stroke"))
Group
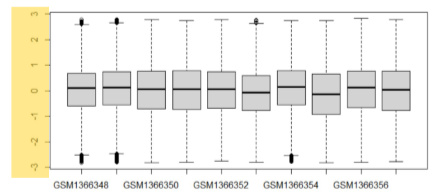
#标准化和log2转化
boxplot(exp,outline=FALSE, notch=T,col=Group, las=2)
library(limma)
exp=normalizeBetweenArrays(exp)
boxplot(exp,outline=FALSE, notch=T,col=Group, las=2)
dim(exp)
#54675 92
max(exp)
#6.63483
#exp=log2(exp+1)#自行判断是否需要log 在0-20之间是正常
exp=as.data.frame(exp)
#探针注释
#捷径
library(tinyarray)
find_anno('GPL570') #打出找注释的代码
ids <- AnnoProbe::idmap('GPL570')
#四种方法,方法1里找不到就从方法2找,以此类推。
#方法1 BioconductorR包(最常用)
gpl_number
#http://www.bio-info-trainee.com/1399.html
if(!require(hgu133plus2.db))BiocManager::install("hgu133plus2.db")
library(hgu133plus2.db)
ls("package:hgu133plus2.db")
ids <- toTable(hgu133plus2SYMBOL)
head(ids)
# 方法2 读取GPL网页的表格文件,按列取子集
##https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL570
if(F){
#注:从GEO页面上下载下来的表格,也有的GPL平台没有提供注释表格
b = read.delim("GPL570-55999.txt",
check.names = F,
comment.char = "#")
colnames(b)
ids = b[,c("ID","Gene Symbol")]
colnames(ids2) = c("probe_id","symbol")
k1 = ids2$symbol!="";table(k1)
k2 = !str_detect(ids2$symbol,"///");table(k2) #只有几百个直接去掉
ids = ids2[ k1 & k2,]
# ids = ids2
}
# 方法3 官网下载注释文件并读取
##http://www.affymetrix.com/support/technical/byproduct.affx?product=hg-u133-plus
# 方法4 自主注释
#https://mp.weixin.qq.com/s/mrtjpN8yDKUdCSvSUuUwcA
ids <- idmap("GPL570")3.差异分析
#差异分析,用limma包来做
#需要表达矩阵和Group,不需要改
library(limma)
design=model.matrix(~Group)
fit=lmFit(exp,design)
fit=eBayes(fit)
deg=topTable(fit,coef=2,number = Inf)4.保留表达量最大的那个探针
#为deg数据框添加几列
#1.加probe_id列,把行名变成一列
library(dplyr)
deg <- mutate(deg,probe_id=rownames(deg))
#2.加上探针注释
ids = ids[!duplicated(ids$symbol),]
ids <- distinct(ids,symbol,.keep_all = T)
#其他去重方式在zz.去重方式.R
deg <- inner_join(deg,ids,by="probe_id")
nrow(deg)
#3.加change列,标记上下调基因
logFC_t=1
p_t = 0.05
k1 = (deg$P.Value < p_t)&(deg$logFC < -logFC_t)
k2 = (deg$P.Value < p_t)&(deg$logFC > logFC_t)
deg <- mutate(deg,change = ifelse(k1,"down",ifelse(k2,"up","stable")))
table(deg$change)
save(deg,exp,ids,pd,file = "out_put.rdata")
load("out_put.rdata")5.画图
{
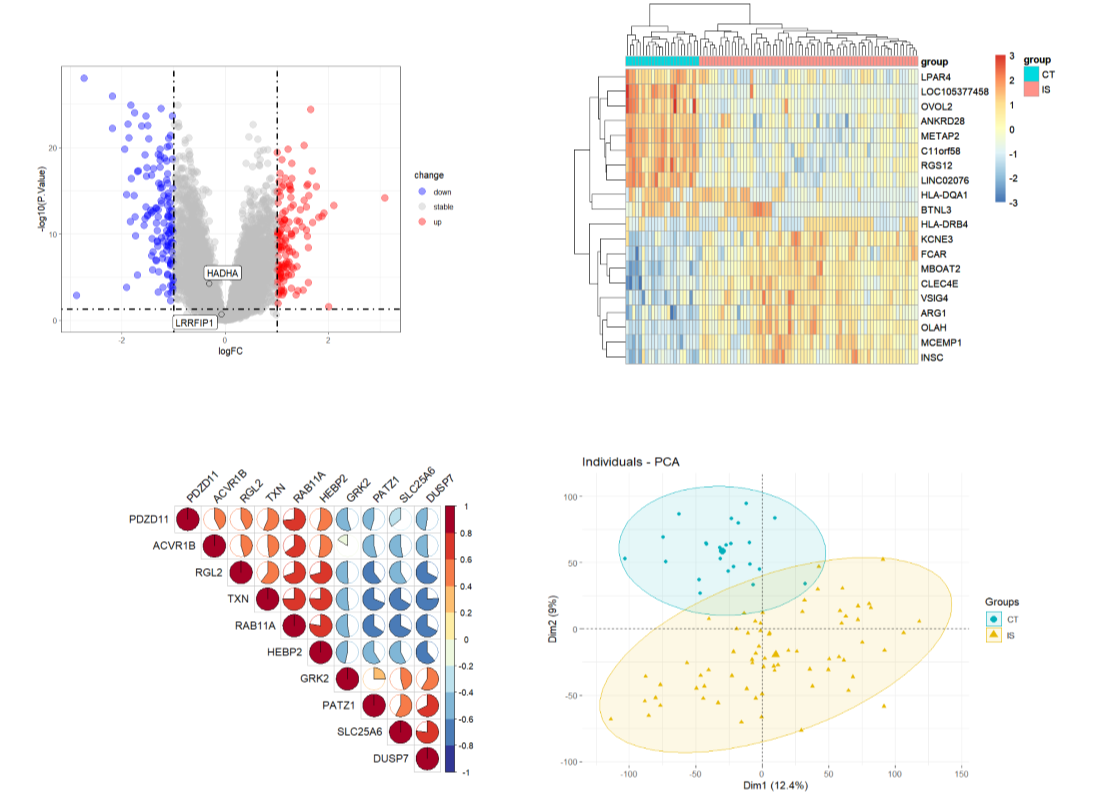
#1.火山图----
library(dplyr)
library(ggplot2)
dat = deg
p <- ggplot(data = dat,
aes(x = logFC,
y = -log10(P.Value))) +
geom_point(alpha=0.4, size=3.5,
aes(color=change)) +
scale_color_manual(values=c("blue", "grey","red"))+
geom_vline(xintercept=c(-logFC_t,logFC_t),lty=4,col="black",linewidth=0.8) +
geom_hline(yintercept = -log10(p_t),lty=4,col="black",linewidth=0.8) +
theme_bw()
p
for_label <- dat%>%
filter(symbol %in% c("HADHA","LRRFIP1"))
volcano_plot <- p +
geom_point(size = 3, shape = 1, data = for_label) +
ggrepel::geom_label_repel(
aes(label = symbol),
data = for_label,
color="black"
)
volcano_plot
#2.差异基因热图----
# 表达矩阵行名替换
exp = exp[dat$probe_id,]
rownames(exp) = dat$symbol
if(T){
#取前10上调和前10下调
library(dplyr)
dat2 = dat %>%
filter(change!="stable") %>%
arrange(logFC)
cg = c(head(dat2$symbol,10),
tail(dat2$symbol,10))
}else{
#全部差异基因
cg = dat$symbol[dat$change !="stable"]
length(cg)
}
n=exp[cg,]
dim(n)
#差异基因热图
library(pheatmap)
annotation_col=data.frame(group=Group)
rownames(annotation_col)=colnames(n)
heatmap_plot <- pheatmap(n,show_colnames =F,
scale = "row",
#cluster_cols = F,
annotation_col=annotation_col,
breaks = seq(-3,3,length.out = 100)
)
heatmap_plot
#拼图
library(patchwork)
volcano_plot +heatmap_plot$gtable
# 3.感兴趣基因的相关性----
library(corrplot)
g = sample(deg$symbol[1:500],10) # 这里是随机取样,注意换成自己感兴趣的基因
g
M = cor(t(exp[g,]))
pheatmap(M)
library(paletteer)
my_color = rev(paletteer_d("RColorBrewer::RdYlBu"))
my_color = colorRampPalette(my_color)(10)
corr_plot <- corrplot(M, type="upper",
method="pie",
order="hclust",
col=my_color,
tl.col="black",
tl.srt=45)
library(cowplot)
cor_plot
#4.PCA----
# 拼图
dat=as.data.frame(t(exp))
library(FactoMineR)
library(factoextra)
dat.pca <- PCA(dat, graph = FALSE)
pca_plot <- fviz_pca_ind(dat.pca,
geom.ind = "point", # show points only (nbut not "text")
col.ind = Group, # color by groups
palette = c("#00AFBB", "#E7B800"),
addEllipses = TRUE, # Concentration ellipses
legend.title = "Groups"
)
pca_plot
save(pca_plot,file = "pca_plot.Rdata")
library(patchwork)
plot_grid(pca_plot,cor_plot,
volcano_plot,heatmap_plot$gtable)
dev.off()
#保存
pdf("deg.pdf")
plot_grid(pca_plot,corr_plot,
volcano_plot,heatmap_plot$gtable)
dev.off()
}图表如下:
大功告成!!!
小洁老师语录:流水不争先,争的是滔滔不绝

原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

