空转 | 结合scRNA完成空转spot注释(Seurat Mapping) & 彩蛋(封面的空转主图代码)
空转 | 结合scRNA完成空转spot注释(Seurat Mapping) & 彩蛋(封面的空转主图代码)

生信补给站
发布于 2023-08-25 10:28:44
发布于 2023-08-25 10:28:44
前面空间转录组|没有单细胞数据如何做空转spot “注释”?文献和代码都给你!介绍了不结合单细胞进行空转spot的注释方式,文献中更多还是结合单细胞转录组数据进行spot注释 。整合scRNA-seq和空间转录组数据主要有(1)映射(Mapping)和(2)去卷积(Deconvolution)两种方法。本文介绍下使用Seurat的实现Mapping结合的方式 ,主要是将基于scRNA的细胞亚型定位到HPRI图谱上,后续会介绍SPOTlight ,CARD 等Deconvolution 的方式。
本文使用2022年Cell Rep Med 期刊 “Distinct phenotypic states and spatial distribution of CD8+ T cell clonotypes in human brain metastases ”中的人 脑转移数据,有对应的单细胞数据。选择文献中提到的 patient16作为示例,也就是GSM5416364_16_features.tsv.gz 和 GSM5420750_spatial.tar.gz。
数据来源:GSE179373 和 GSE179572 ,或者后台回复 “空转数据”即可获得本文使用的patient16数据。
一 scRNA ,ST 标准分析
1 空间转录组
载入相关的R包,读取脑转移的数据,快读进行空转的标准分析空间转录组|Load10X_Spatial函数修改适配多形式数据 + 空转标准流程
library(Matrix)
library(data.table)
library(Seurat)
library(SeuratData)
library(tidyverse)
library(gt)
library(RColorBrewer)
library(gridExtra)
############空间数据部分###################
Brain_ST = Load10X_Spatial(data.dir = "./Brain_35707680",
filename = "filtered_feature_bc_matrix.h5",
assay = "Spatial")
Brain_ST <- SCTransform(Brain_ST, assay = "Spatial", verbose = FALSE)
Brain_ST <- RunPCA(Brain_ST, assay = "SCT", verbose = FALSE)
Brain_ST <- FindNeighbors(Brain_ST, reduction = "pca", dims = 1:30)
Brain_ST <- FindClusters(Brain_ST, verbose = FALSE)
Brain_ST <- RunUMAP(Brain_ST, reduction = "pca", dims = 1:30)
p1 <- DimPlot(Brain_ST, reduction = "umap", label = TRUE)
p2 <- SpatialDimPlot(Brain_ST, label = TRUE, label.size = 3)
p1 + p2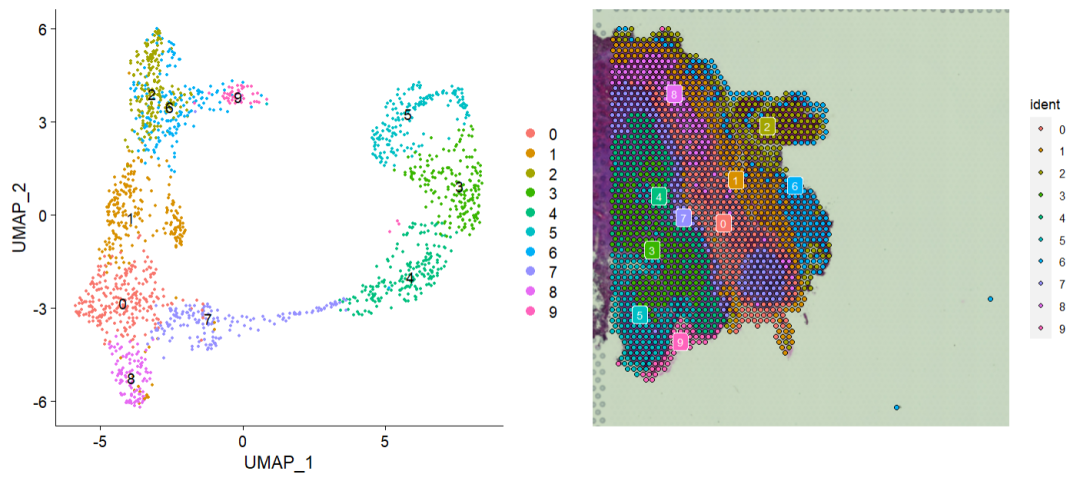
2 单细胞转录组
读取对应样本的单细胞转录组数据,标准分析 ,保存Brain_ST_scRNA.sBio.Rdata 以备后续使用。
注意为了后续对空转数据进行注释,因此这里随意给cluster结果指定了celltype,仅为示例,无实际意义。
Brain_scRNA <- CreateSeuratObject(counts = Read10X("./Brain_35707680/scRNA/"),
project = "scRNA" )
#质控
Brain_scRNA[["percent.mt"]] <- PercentageFeatureSet(Brain_scRNA, pattern = "^MT-")
Brain_scRNA <- subset(Brain_scRNA, subset = nFeature_RNA > 200 &
nFeature_RNA < 5000 &
percent.mt < 30)
#标准分析
Brain_scRNA <- NormalizeData(Brain_scRNA)
Brain_scRNA <- FindVariableFeatures(Brain_scRNA, selection.method = "vst", nfeatures = 2000)
Brain_scRNA <- ScaleData(Brain_scRNA)
Brain_scRNA <- RunPCA(Brain_scRNA, npcs = 50)
Brain_scRNA <- FindNeighbors(Brain_scRNA, dims = 1:30)
Brain_scRNA <- FindClusters(Brain_scRNA, resolution = 0.5)
Brain_scRNA <- RunUMAP(Brain_scRNA, dims = 1:30)
Idents(Brain_scRNA) <- "seurat_clusters"
p11 <- DimPlot(Brain_scRNA, reduction = "umap", pt.size=0.5, label = F,repel = TRUE)
#这里是随意注释的,只是为了后续展示
Brain_scRNA <- RenameIdents(Brain_scRNA,"0"="CD8",
"1"="NK",
"2"="Myeloid",
"3"= "Fibroblast",
"4"= "Tumor",
"5"= "CD4",
"6"= "B"
)
Brain_scRNA@meta.data$celltype <- Idents(Brain_scRNA)
p22 <- DimPlot(Brain_scRNA, reduction = 'umap',
label = TRUE, pt.size = 0.5) + NoLegend()
p11 + p22
#保存结果,以备后续使用
save(Brain_scRNA,Brain_ST,file = "Brain_ST_scRNA.sBio.Rdata")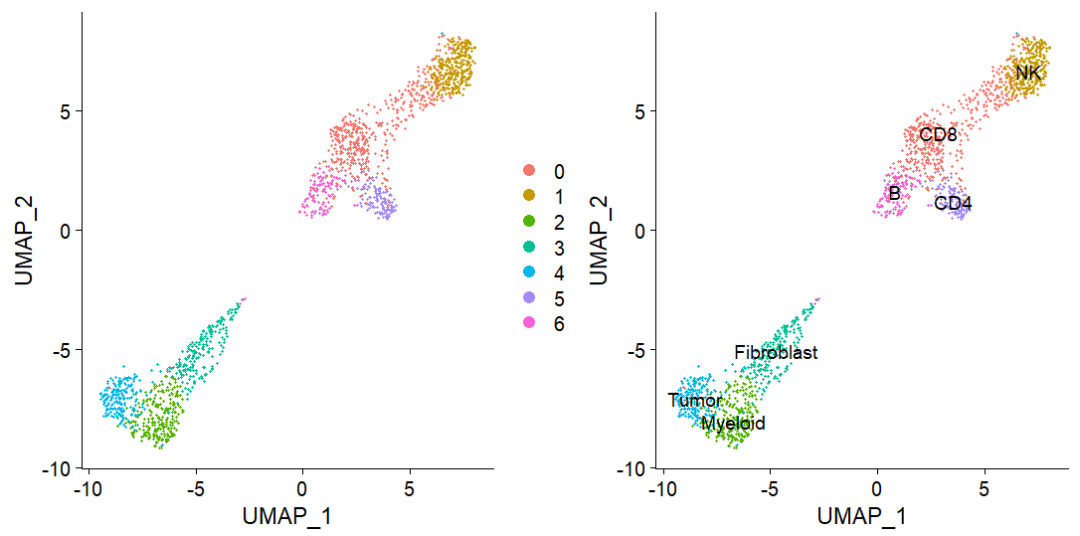
注:celltype是随意注释的,只是为了后续展示 .
二 Seurat - 空间spot注释
这里介绍下使用Seurat结合单细胞转录组进行空间注释的方法,主要函数为FindTransferAnchors 和 TransferData 。
1, FindTransferAnchors
首先使用SCTransform 处理下数据,如果前面是SCTransform处理的则可以省略该步骤。
Brain_scRNA_reference <- SCTransform(Brain_scRNA, ncells = 3000, verbose = FALSE) %>%
RunPCA(verbose = FALSE) %>%
RunUMAP(dims = 1:30)
# the annotation is stored in the 'celltype' column of object metadata
DimPlot(Brain_scRNA_reference, group.by = "celltype", label = TRUE)
# 空转是SCT处理的,因此此处无需再次处理
Brain_ST_2 <- Brain_STFindTransferAnchors 结合
anchors <- FindTransferAnchors(reference = Brain_scRNA_reference,
query = Brain_ST_2,
normalization.method = "SCT")2, TransferData
使用TransferData函数进行注释,其中refdata 即为单细胞转录组中的参考列
predictions.assay <- TransferData(anchorset = anchors,
refdata = Brain_scRNA_reference$celltype,
prediction.assay = TRUE,
weight.reduction = Brain_ST_2[["pca"]],
dims = 1:30)
#查看一下结果
class(predictions.assay)
#[1] "Assay"
#attr(,"package")
#[1] "SeuratObject"
predictions.assay@data[,1:4]
可以看到结果的 每行 是单细胞celltype列中的细胞类型,列为barcode的ID ,会给出每个spot 的celltype 占比,介绍2种保存方式
(1)可以像之前一样添加至metadata.
(2)因为predictions.assay 本身就是SeuratObject ,因此可以单独作为一个新的slot 。
#保存结果
#(1) metadata
predictions.res <- predictions.assay@data
rownames(predictions.res) <- make.names(rownames(predictions.res))
cell_types <- rownames(predictions.res)
for(cell_type in cell_types){
Brain_ST_2 <- AddMetaData(object= Brain_ST_2,metadata = predictions.res[cell_type,],col.name = cell_type)
}
#(2)slot
Brain_ST_2[["predictions"]] <- predictions.assay以上就简单的完成了Seurat中结合单细胞和空转的分析。
三 空转区域差异
得到了每个spot的每种celltype的预测结果,可以查看一下关心的细胞类型分布
DefaultAssay(Brain_ST_2) <- "predictions"
SpatialFeaturePlot(Brain_ST_2, features = c("CD4", "CD8"),
pt.size.factor = 1.6, ncol = 2, crop = TRUE)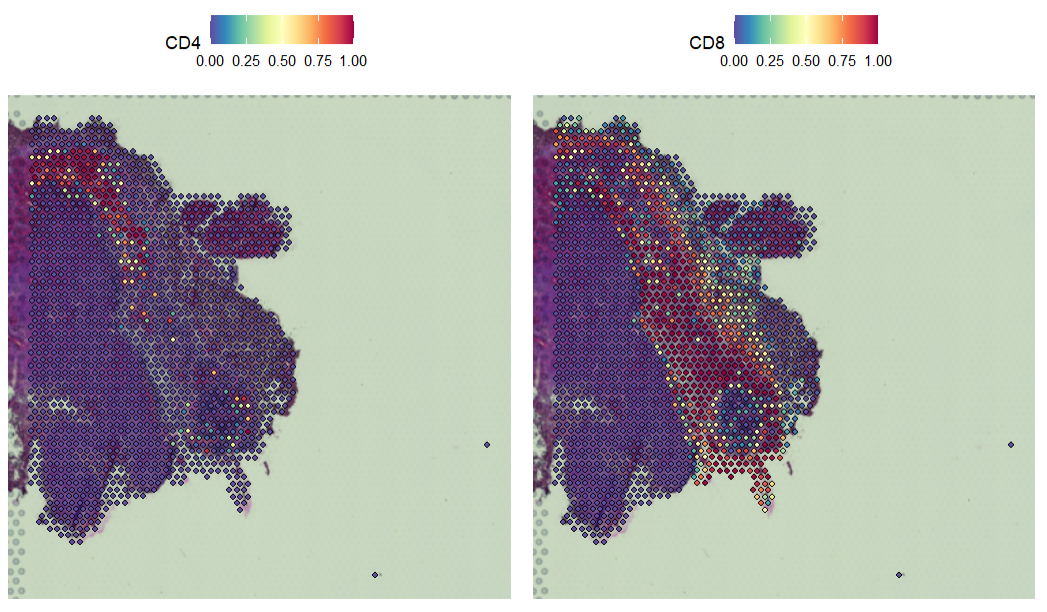
基于这些预测分数,还可以预测其位置受空间限制的细胞类型。使用每个spot 细胞类型的预测分数替代基因表达来作为“marks” 得到spatially variable features
Brain_ST_2 <- FindSpatiallyVariableFeatures(Brain_ST_2,
assay = "predictions",
selection.method = "moransi",
features = rownames(Brain_ST_2),
r.metric = 5,
slot = "data")
top.clusters <- head(SpatiallyVariableFeatures(Brain_ST_2, selection.method = "moransi"), 4)
SpatialPlot(object = Brain_ST_2, features = top.clusters, ncol = 2)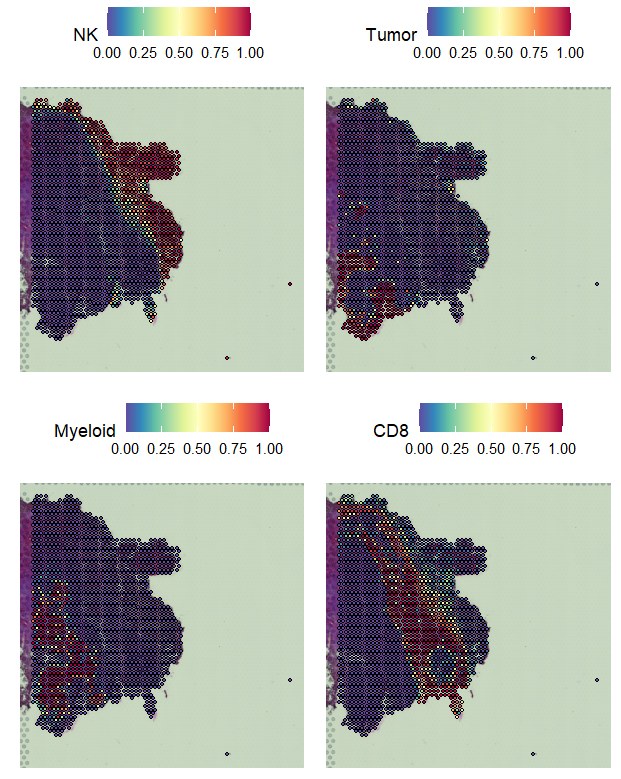
以上分析就完成了,最后给一个彩蛋,使用grid.arrange 函数自定义空转图形的分布,参考2022年Immunity文献。
四 彩蛋- 空转主图可视化
可以学习以下几个点
(1)外层设定统一的theme , 如hide_axis
(2)多张图根据重要性分布 ,HE染色图可以大一些 ,CA9是肿瘤的marker,也可以重点展示
(3)lapply 得到图形的list结果,grid.arrange 来拼图,可以随意调整位置和比例,是直接FeaturePlot得不到的。
(4)图像相关参数随意改,适合文章其他图的风格即可
#hide spatial axes for seurat plots
hide_axis <- theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank())
Idents(Brain_ST_2) <-as.numeric(Idents(Brain_ST_2))
#H&E
he_Brain_ST_2 <- SpatialPlot(Brain_ST_2,repel = F,label = F,
image.alpha=1,
alpha = c(0,0),
pt.size.factor =0.000001) +
geom_point(alpha=0)+
NoLegend() +
DarkTheme() +
hide_axis +
ggtitle("Brain_ST_2")+
theme(text=element_text(size=14))+
theme(text=element_text(face = "bold"))
# celltype score
cell_types <- c("Tumor" , "Fibroblast","CD8" , "Myeloid" ,"NK" ,"CD4", "B","max" )
plot_list_mcp <- lapply(cell_types[c(5,1,2,4,6,3,7,8)],function(x){
plot_list_mcp <- SpatialPlot(Brain_ST_2,
features = x,
image.alpha=0,
pt.size.factor = 1.8) +
DarkTheme() +
hide_axis +
theme(text=element_text(size=14))+
theme(text=element_text(face = "bold"))+
theme(legend.text=element_text(size=7))
})
#CA9 重点marker
ca9 <- SpatialPlot(Brain_ST_2,
features = "CA9",
image.alpha=0,
pt.size.factor = 1.8) +
DarkTheme() +
hide_axis +
theme(text=element_text(size=14))+
theme(text=element_text(face = "bold"))+
theme(legend.text=element_text(size=7))
#########拼图############
plot_list_mcp[[9]] <- ca9
plot_list_mcp[[10]] <- he_Brain_ST_2
#图形摆放
lay <- rbind(c(10,9,1,2,3,4),
c(10,9,5,6,7,8))
grid.arrange(grobs = plot_list_mcp, layout_matrix = lay)
OK !下次介绍使用SPOTlight 解卷积的方式 进行空转spot的注释。
参考资料:
[1]Analysis, visualization, and integration of spatial datasets with Seurat • Seurat (satijalab.org)
[2]Tertiary lymphoid structures generate and propagate anti-tu[]mor antibody-producing plasma cells in renal cell cancer
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-07-03,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

