🤩 Monocle 3 | 太牛了!单细胞必学R包!~(三)(建立单细胞轨迹)
🤩 Monocle 3 | 太牛了!单细胞必学R包!~(三)(建立单细胞轨迹)
生信漫卷
发布于 2023-11-06 15:45:13
发布于 2023-11-06 15:45:13
1写在前面
到周末了,今天去骑车的,感觉还不错。🚴🏻
下面就是今天的内容了:👇
单细胞转录组、蛋白组、表观组学等单细胞技术的发展为研究细胞周期、细胞分化等细胞动态过程提供了新的机会。🤩
使用轨迹推断(TI,trajectory inference)的方法可以根据测序的细胞之间表达模式的相似性对单细胞沿着轨迹进行排序,模拟细胞动态变化的过程。🥸
轨迹推断也称作伪时间分析(pseudotime analysis)。🤩
2用到的包
rm(list = ls())
library(tidyverse)
library(monocle3)
3示例数据
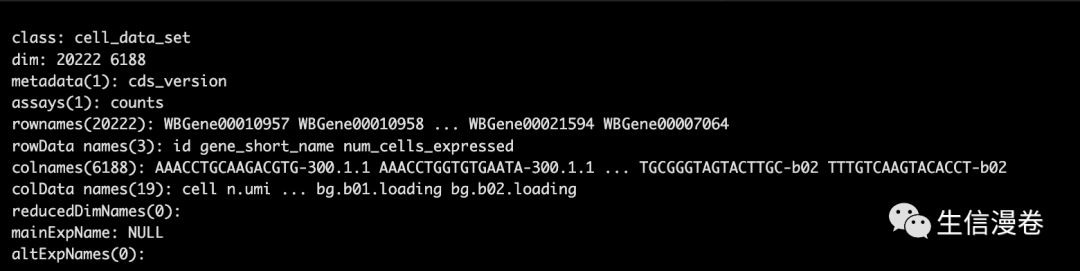
expression_matrix <- readRDS("./packer_embryo_expression.rds")
cell_metadata <- readRDS("./packer_embryo_colData.rds")
gene_annotation <- readRDS("./packer_embryo_rowData.rds")
cds <- new_cell_data_set(expression_matrix,
cell_metadata = cell_metadata,
gene_metadata = gene_annotation)
cds
4预处理数据
我们先处理一下批次效应,这里有一个原文中提供的方法,我们用一下吧。😏
自己的数据还是要自己的方法处理的。😂
cds <- preprocess_cds(cds, num_dim = 50)
cds <- align_cds(cds, alignment_group = "batch", residual_model_formula_str = "~ bg.300.loading + bg.400.loading + bg.500.1.loading + bg.500.2.loading + bg.r17.loading + bg.b01.loading + bg.b02.loading")
5降维并可视化结果
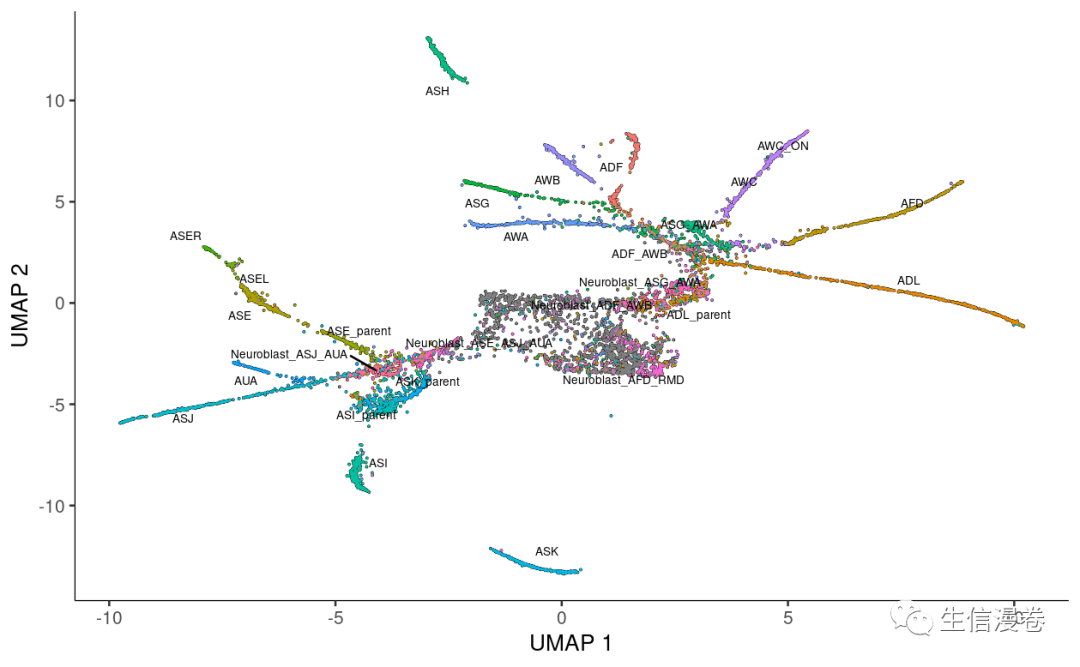
我们只是用了data的一小部分,但Monocle重建了一个有许多分支的轨迹。🥰
叠加人工注释后,这些分支主要由一种细胞类型占据。😋
cds <- reduce_dimension(cds)
plot_cells(cds, label_groups_by_cluster=FALSE, color_cells_by = "cell.type")
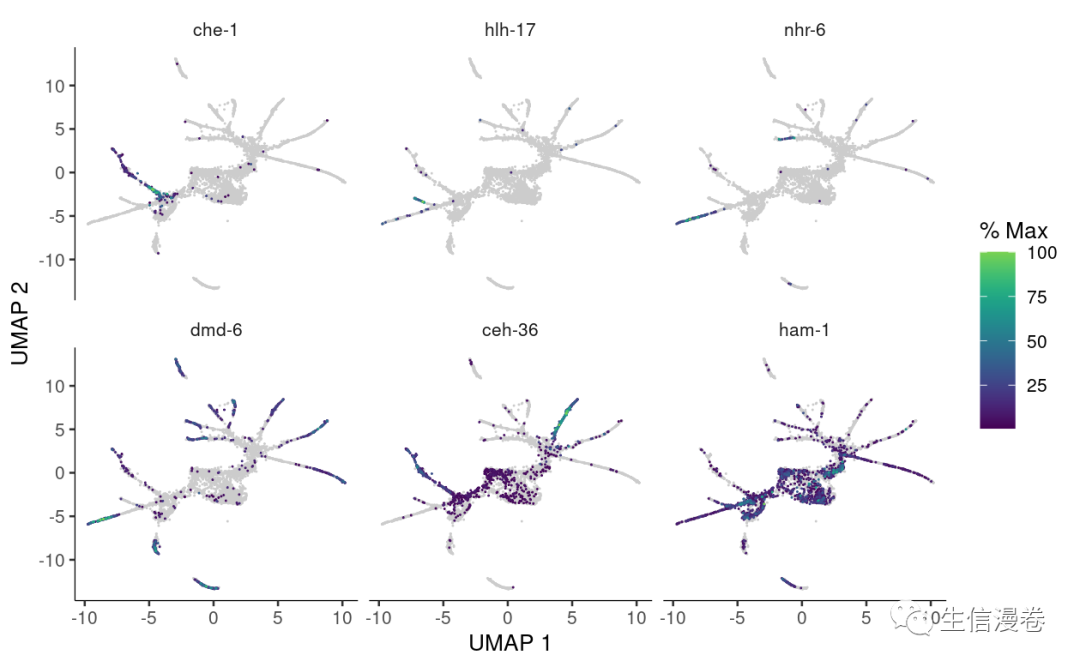
我们再看一下单个基因沿轨迹的变化。🤩
ciliated_genes <- c("che-1",
"hlh-17",
"nhr-6",
"dmd-6",
"ceh-36",
"ham-1")
plot_cells(cds,
genes=ciliated_genes,
label_cell_groups=FALSE,
show_trajectory_graph=FALSE)
6细胞聚类
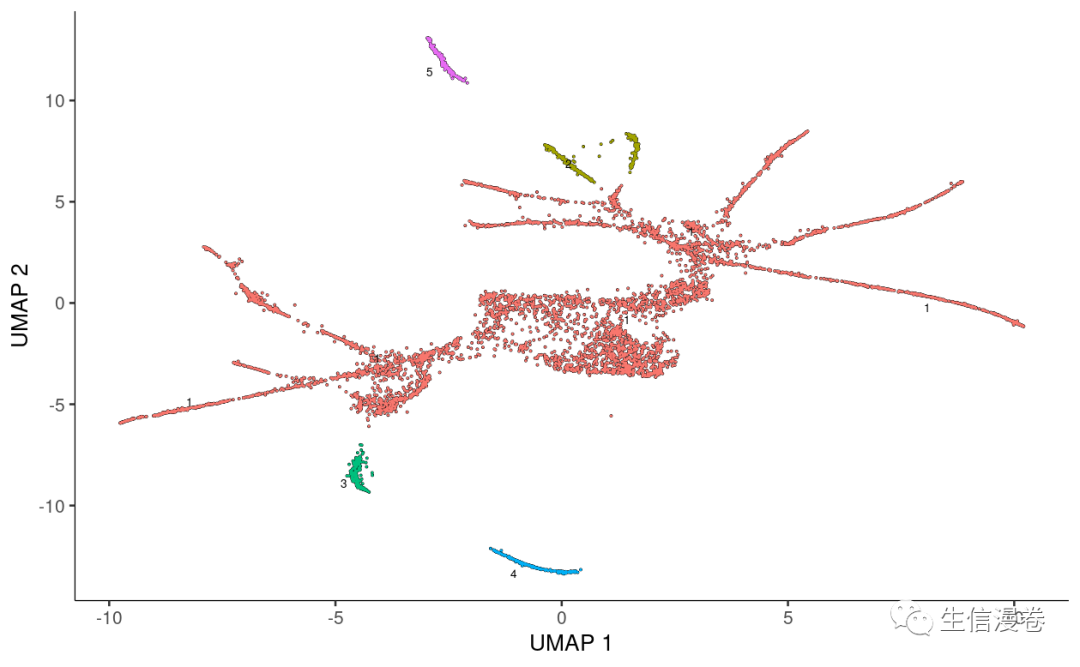
尽管细胞可以连续地从一个状态过渡到下一个状态,之间没有明显边界,但Monocle并不假设数据集中的所有细胞都是从一个共同的转录"祖先"转化而来。😘
事实上可能有多个不同的轨迹。🧐
比如,对感染的反应时,固有免疫细胞和基质细胞会有非常不同的初始转录组,对感染的反应也很不同,所以它们应该是同一轨迹的一部分。😍
细胞会分成一个个partition,然后形成单独的轨迹。😏
cds <- cluster_cells(cds)
plot_cells(cds, color_cells_by = "partition")
拟合主图!~🤩
我们需要使用learn_graph()函数在每个partition内拟合一个主图。🤨
cds <- learn_graph(cds)
plot_cells(cds,
color_cells_by = "cell.type",
label_groups_by_cluster=FALSE,
label_leaves=FALSE,
label_branch_points=FALSE)
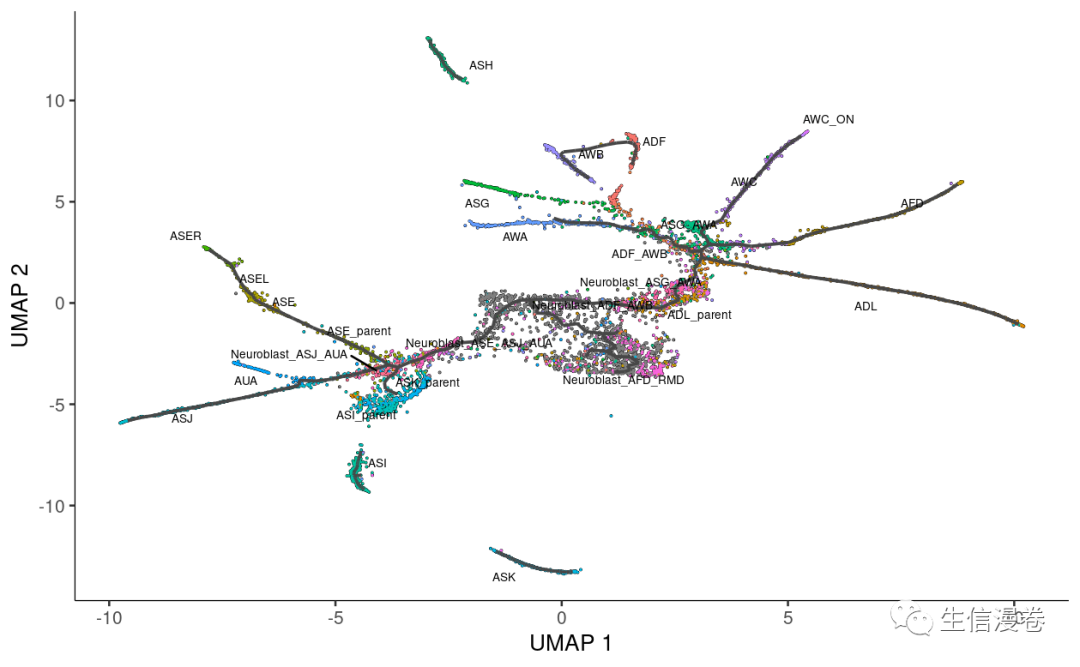
7轨迹排序
首先我们需要找到轨迹的roots,这个可能需要根据你的实验目的来确定。🥸
plot_cells(cds,
color_cells_by = "embryo.time.bin",
label_cell_groups=FALSE,
label_leaves=TRUE,
label_branch_points=TRUE,
graph_label_size=1.5)
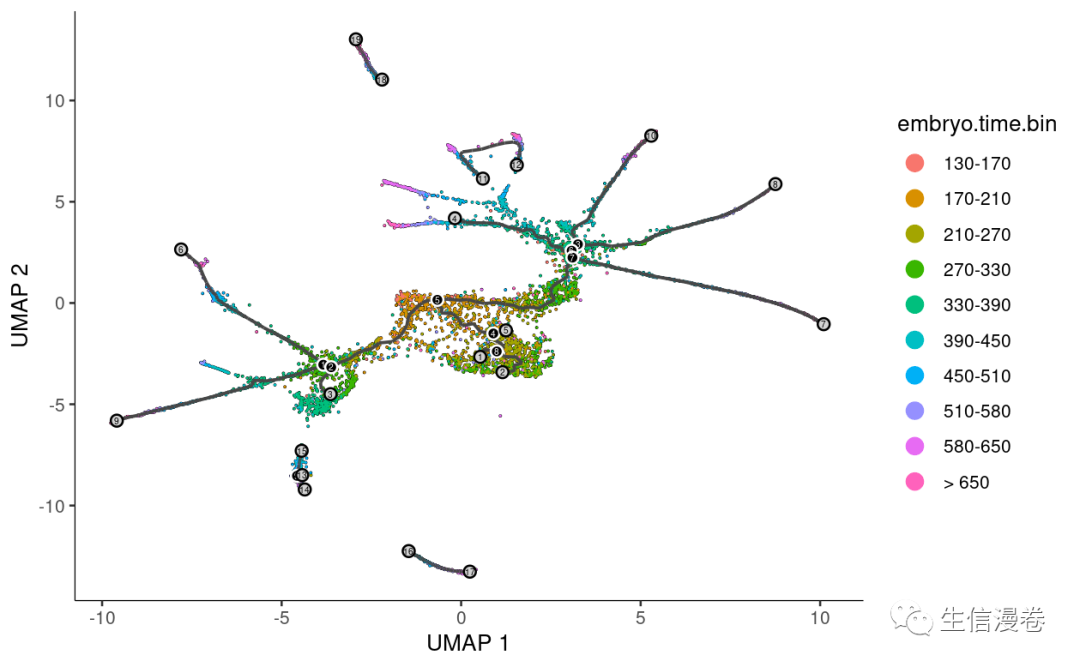
选择root。😀
cds <- order_cells(cds)
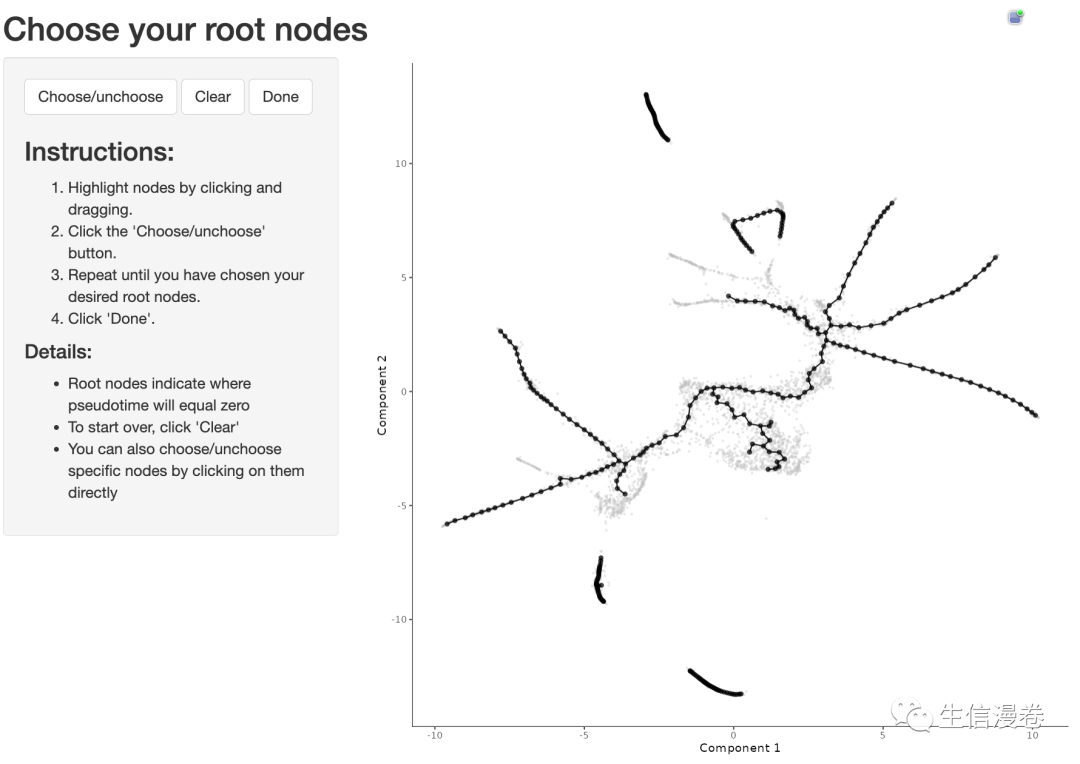
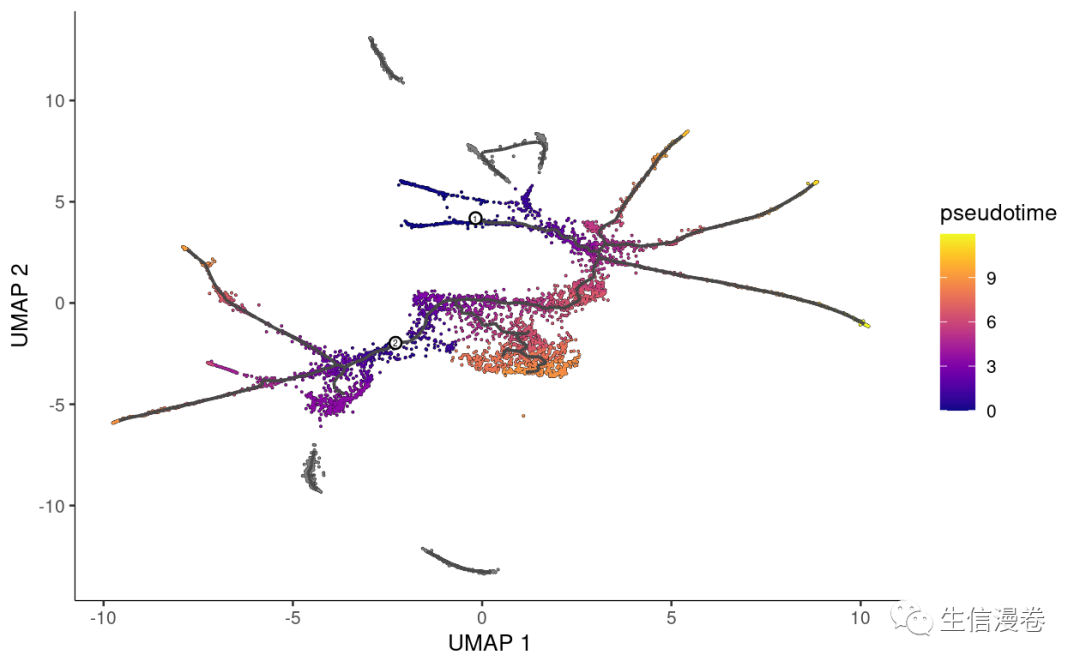
这里我随便标了2个root,我们看一下效果。🥰
plot_cells(cds,
color_cells_by = "pseudotime",
label_cell_groups=FALSE,
label_leaves=FALSE,
label_branch_points=FALSE,
graph_label_size=1.5)
我知道你肯定觉得手动挑选root很累。😋
好的,代码方式来了。😂
get_earliest_principal_node <- function(cds, time_bin="130-170"){
cell_ids <- which(colData(cds)[, "embryo.time.bin"] == time_bin)
closest_vertex <-
cds@principal_graph_aux[["UMAP"]]$pr_graph_cell_proj_closest_vertex
closest_vertex <- as.matrix(closest_vertex[colnames(cds), ])
root_pr_nodes <-
igraph::V(principal_graph(cds)[["UMAP"]])$name[as.numeric(names
(which.max(table(closest_vertex[cell_ids,]))))]
root_pr_nodes
}
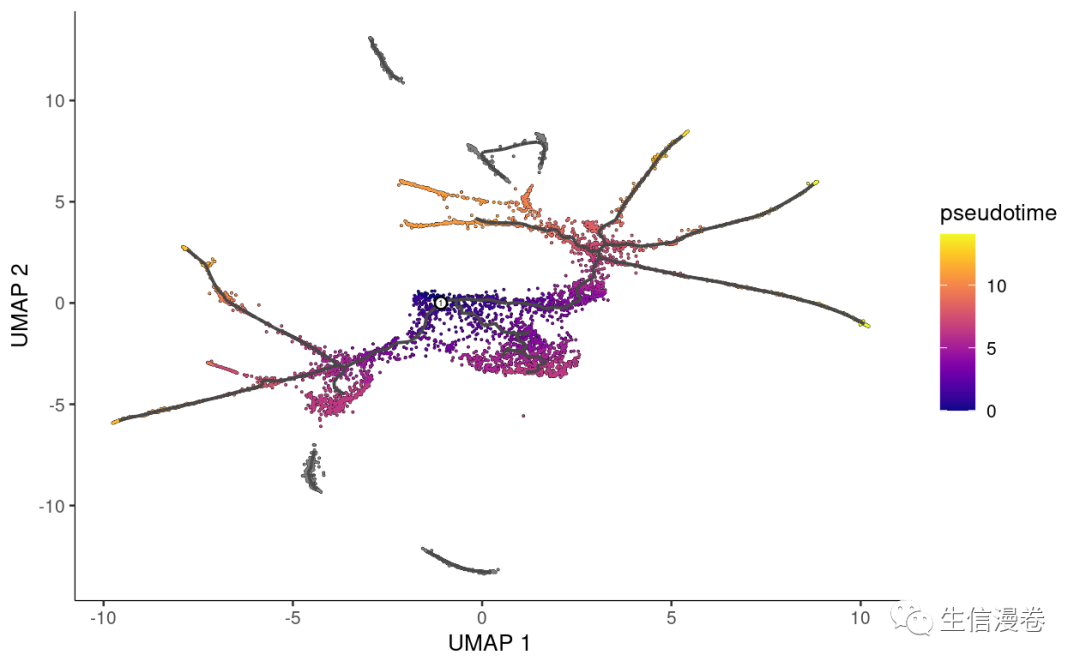
cds <- order_cells(cds, root_pr_nodes=get_earliest_principal_node(cds))
plot_cells(cds,
color_cells_by = "pseudotime",
label_cell_groups=FALSE,
label_leaves=FALSE,
label_branch_points=FALSE,
graph_label_size=1.5)
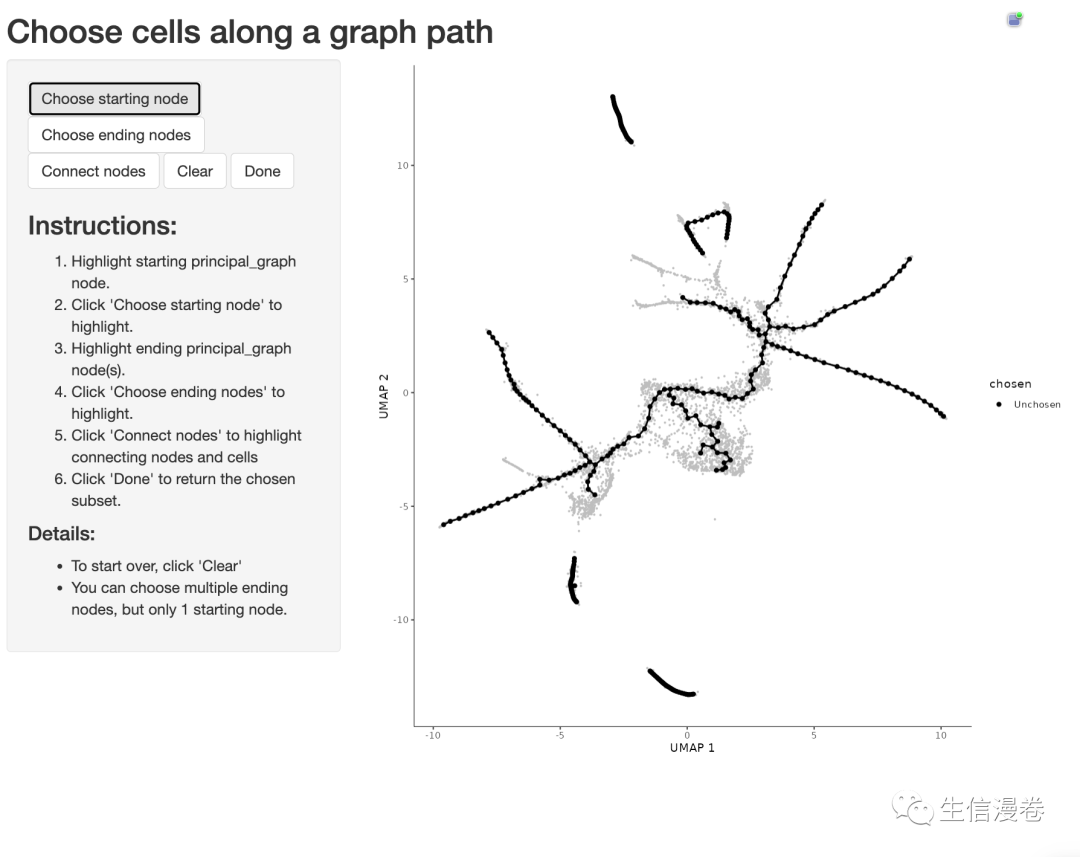
8挑选分支
这里我随便挑选一下分支哦。😜
cds_sub <- choose_graph_segments(cds)
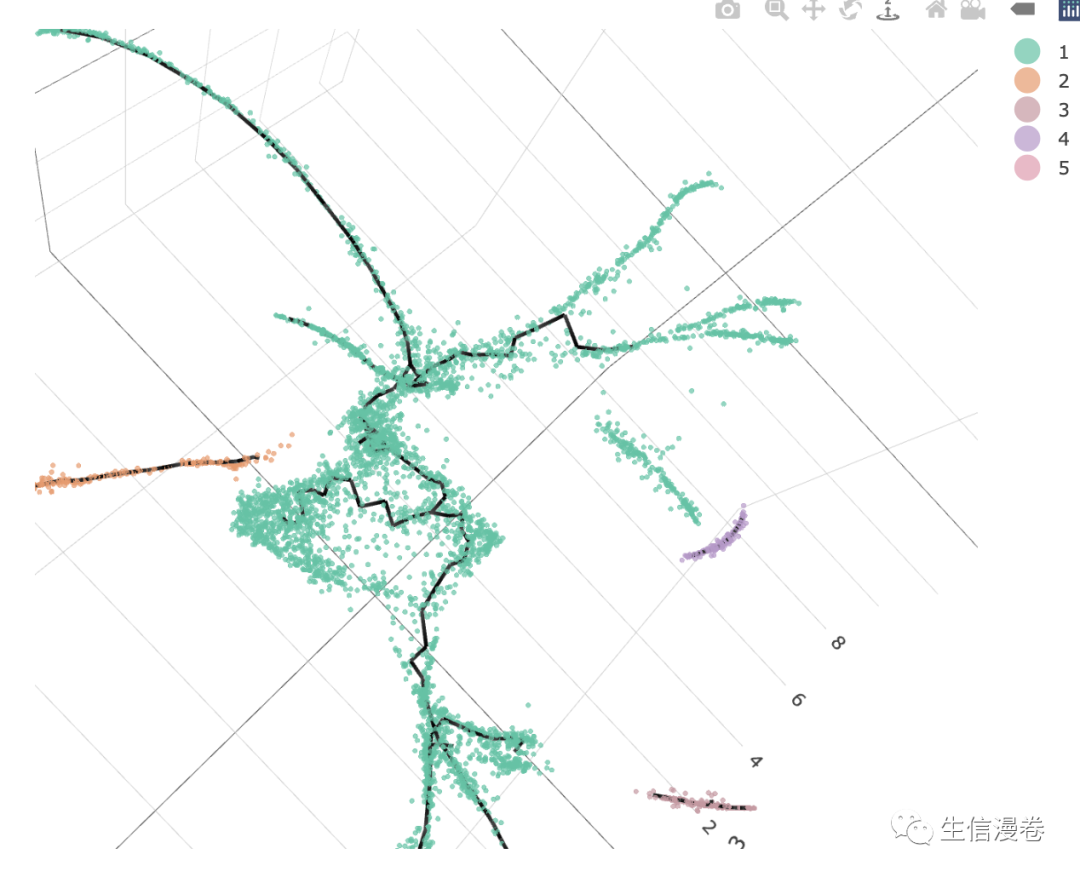
93D展示
cds_3d <- reduce_dimension(cds, max_components = 3)
cds_3d <- cluster_cells(cds_3d)
cds_3d <- learn_graph(cds_3d)
cds_3d <- order_cells(cds_3d, root_pr_nodes=get_earliest_principal_node(cds))
cds_3d_plot_obj <- plot_cells_3d(cds_3d, color_cells_by="partition")
最后祝大家早日不卷!~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-11-04,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

