生物信息学必备工具—SAMtools
工欲善其事必先利其器
1Samtools
Samtools 同样是由李恒博士开发的一套与高通量测序数据交互的程序。它由三个独立的存储库组成:
- Samtools 读/写/编辑/索引/查看SAM / BAM / CRAM格式
- BCFtools 读/写BCF2 / VCF / gVCF文件和调用/过滤/汇总SNP和短插入序列变体
- HTSlib 用于读/写高通量测序数据的AC库
Samtools和BCFtools都在内部使用HTSlib,但这些源包包含它们自己的htslib副本,因此它们可以独立构建。因其具有:
- 多功能性:能够处理、排序、索引和转换SAM/BAM文件。
- 高效性:特别适合于处理大型测序数据集。
- 灵活性:提供多种数据处理选项和参数设置。
- 广泛兼容性:与其他生物信息学工具和流程兼容。
- 易于集成:可以轻松集成到自动化的生物信息学分析流程中。
- 强大的数据过滤和查询功能:能够高效地过滤和查询特定的数据。
- 开源:开放源代码,方便用户修改和定制。
这些优势使Samtools成为生物信息学领域研究人员广泛使用的关键工具之一。
2发表文章
题目:The Sequence Alignment/Map format and SAMtools ;The Sequence Alignment/Map format and SAMtools 发表日期:2009/8/15;2021/1/29 期刊:Bioinformatics ;GigaScience 作者&单位:李恒;哈佛医学院生物医学信息学系和Dana-Farber癌症研究所数据科学系 官网:https://www.htslib.org/ 旧版本网址:https://samtools.sourceforge.net/ 主要编程语言:C语言

3简要用途
Samtools是一套实用工具,用于处理SAM(序列比对/映射)、BAM和CRAM格式的比对数据【通常是短序列比对工具如bwa,bowtie2,hisat2,STAR等等产生的】。它可以在这些格式之间进行转换,执行排序、合并和构建索引,还能快速检索任何区域的读取数据。其包含有许多子命令:
sort用于对文件进行排序index生成索引文件view主要用途有两个,一是文件SAM和BAM之间的格式转换,二是查看二进制文件stats:生成关于比对数据的统计信息,如测序覆盖度、比对质量等faidx对fasta文件建立索引,生成的索引文件以.fai后缀结尾。该命令也能依据索引文件快速提取fasta文件中的某一条(子)序列tview查看reads比对到基因组的情况,类似基因组浏览器的功能markdup标记重复序列,在duplicate read上标注,并没有将它从sam文件中去除merge用于合并多个已排序的比对文件,生成一个包含所有输入记录的单一排序输出文件,同时保持现有的排序顺序。
.......
4如何安装
conda 安装
首选推荐Conda安装,非常简单
#codna create -n wes #先创建小环境,如果已经创建,可以忽略
conda activate wes
conda install -y samtools
二进制安装
也可以采用二进制包编译安装,比起conda稍微复杂一点,不过安装也很顺利。
wget -c https://github.com/samtools/samtools/releases/download/1.18/samtools-1.18.tar.bz2
tar -xf samtools-1.18.tar.bz2
##./configure #默认位置是安装在 /usr/local/bin ,非管理员用户是没有这个权限的,所以我们需要指定自己有读写权限的目录来安装
./configure --prefix=/home/data/username/software/samtools ##自定义安装位置,注意需要时绝对路径
make
make install
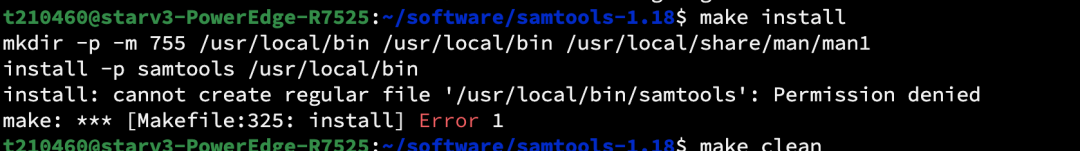
未指定目录安装,非管理员用户会报错
5高频用法
samtools 有39个子命令,但是最常用的功能就是对bam文件排序后构建索引,然后进行后续的生物信息学分析。也就是我们常说的 samtools三步曲:sam转bam,bam排序,bam建索引(旧版本),但是目前samtoools 对sam 文件进行 sort 排序的时候是可以直接输出bam的,因此可以缩减为2步——sam排序输出bam、bam建索引
## 示例
## sam排序,输出bam
samtools sort -@ 2 -o ${outdir}/${sample}.sort.bam
## 建索引
samtools index d0_sort.bam
BWA本身不直接输出BAM文件。它通常输出SAM格式的文件,用于存储序列比对结果。但是SAM文件比较占用空间,为了得到BAM格式的文件(一种更紧凑的二进制格式),通常通道符叠加使用samtools 将BWA的输出从SAM格式转换为BAM格式
##和bwa联用示例
id=d0
bwa mem -M -t 4 \
-R "@RG\tID:${id}\tSM:${id}\tLB:WXS\tPL:Illumina" \
~/database/GATK/hg38/bwa-mem1-index/gatk_hg38 \
~/sam_test/d0_1.fastq.gz \
~/sam_test/d0_2.fastq.gz \
| samtools sort -@ 4 -m 1G -o ~/sam_test/bwa_bam/d0_sort.bam -
为什么要转换为bam文件
BAM是一种压缩的二进制格式,占用更少的存储空间;同时由于其压缩性质,BAM文件在数据检索时通常比SAM文件更高效。
##简单对比感受一下sam和bam占用存储空间大小的差别
## 原始文件大小
2.5G 12月 12 11:04 d0_1.fastq.gz
2.7G 12月 12 11:05 d0_2.fastq.gz
## 产生的sam文件大小
27G 12月 12 12:00 d0.sam
## 产生的bam文件大小
3.1G 12月 12 15:37 d0.bam
## 以示例来说存储空间相差了9倍
6其余子命令参数及用法
sort
samtools sort -@ 4 d0.sam -o ./d0_sort.bam
-T #设置临时文件前缀,将临时文件写入PREFIX.nnnn.bam(排序过程中会产生好多临时文件)
-@ #定义命令执行所用的n个线程(排序和压缩)
-o #将最终排序输出写入FILE,而非标准输出,设定排序后的输出文件名
-O #将最终输出写为sam、bam或cram格式(文件名后缀也可以自动识别)
-m #每个线程大约需要的最大内存,单位为字节或带K、M、G后缀。(对于处理大数据时,如果内存够用,则设置大点的值,以节约时间)
-no-PG:#不在输出文件的头部添加@PG行
-l INT:#设置最终输出文件的压缩级别,范围从0(无压缩)到9(最佳压缩但写入最慢)
-u:#设置压缩级别为0,即无压缩输出
index
用于快速随机访问的索引创建
- 必须对bam文件进行排序后,才能进行index。否则会报错。
- 建立索引后将产生后缀为.bai的文件,用于快速的随机处理。很多情况下需要有bai文件的存在,特别是显示序列比对情况下。比如samtool的tview命令就需要。
- BAI索引格式支持最长512 Mbp(2^29碱基)的单个染色体。如果输入文件可能包含映射到更远位置的读取,需要使用CSI索引。
samtools index d0_sort.bam
#设定线程
samtools index -@ 2 d0_sort.bam
samtools index -b -@ 2 d0_sort.bam
samtools index -b -@ 2 d0_sort.bam -o d0_sort.bam.bai
-b #对bam文件生成BAI-format的索引,系统默认
-c #对bam文件生成CSI-format的索引
-@ #设定线程数
-o #将输出索引写入FILE。仅在索引单个比对文件时可用
view
主要用于将SAM、BAM或CRAM格式转换;以及区域过滤查看
##查看BAM文件
samtools view d0_sort.bam|less -SN
##SAM文件转BAM
samtools view -b -h d0.sam > test.bam
samtools view -b -h d0.sam -o test.bam
##BAM文件转SAM
samtools view -h d0.bam > test.sam
samtools view -h d0.bam -o test.sam
-b #view命令默认下输出是 SAM 格式文件,该参数设置输出为 BAM 格式
-H #仅仅输出文件的头部信息
-h #默认下输出的 sam 格式文件不带 header,该参数设定输出sam文件时带 header 信息
-@ #指定线程
-o #设定输出文件
-1 #启用快速压缩,更改默认输出格式为BAM
satas
从 BAM 文件收集统计信息,并以文本格式输出,可以使用 plot-bamstats 以图形方式可视化输出。详细统计信息见:https://www.htslib.org/doc/samtools-stats.html
samtools stats d0.bam > test.bam.stats
-@ #指定线程
faidx
#对参考基因组建立索引
samtools faidx ~/database/Homo_sapiens_assembly38.fasta -o ./Homo_sapiens_assembly38.fasta.fai
#由于有索引文件,可以使用以下命令很快从基因组中提取到fasta格式的子序列
samtools faidx ~/database/Homo_sapiens_assembly38.fasta chr1 -o ./hg38_chr1.fasta
tview
查看reads比对到基因组的情况,类似基因组浏览器的功能
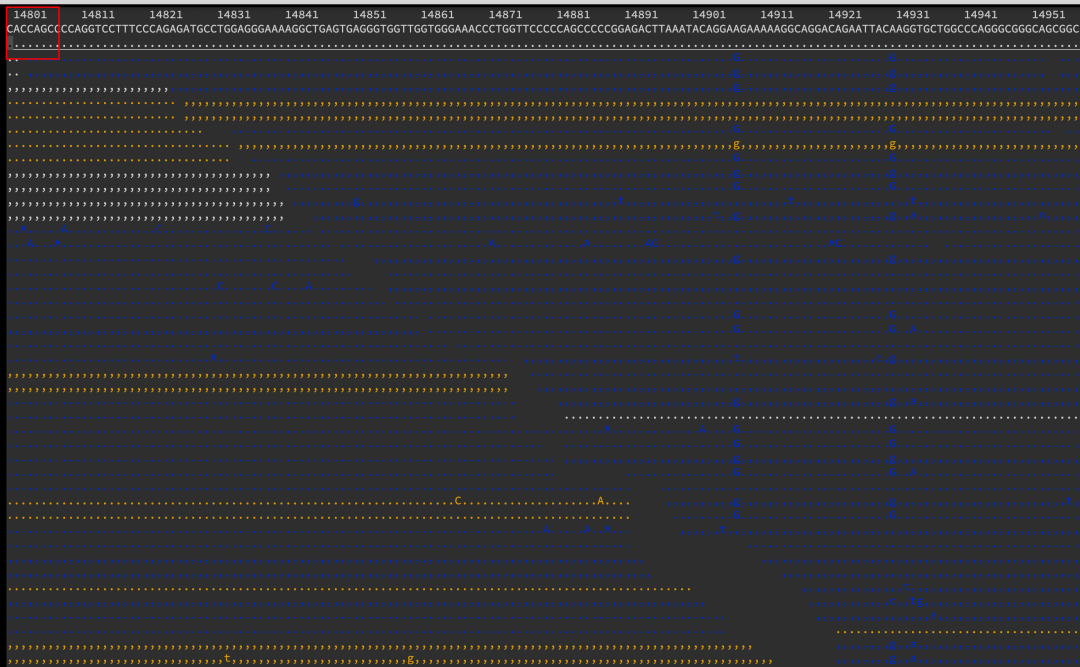
- 顶部显示的是参考序列,如果未知则显示为'N'。参考序列下方是由序列比对得出的共识序列。共识序列下面是序列比对记录。大写和小写用于区分序列链,大写代表正向链。
- 按下 g ,则提示输入要到达基因组的某一个位点。例子“chr1:14800"表示到达1号染色体,第14800个碱基位点处。
- 当参考序列已知时,共识序列和比对记录序列会使用点标记法显示。在这种显示方式中,与参考序列匹配的碱基会用点(
.)表示在正向链,或逗号(,)表示在反向链。与参考序列不匹配的碱基和缺失的碱基则会以它们的碱基符号显示。例如,在一个特定位置,如果所有比对到的序列都与参考序列匹配,那里就会显示点(.)或逗号(,)。如果有不匹配或缺失的碱基,它们会以实际的碱基符号(如A、T、C、G)显示。此显示模式可以通过按下“.”键进行切换。这种显示方式有助于快速识别序列比对中的一致性和差异性。 - 按
?获取帮助文档
###注意:bam和genome基因组(fasta文件都要先建立索引
samtools tview d0.bam ~/database/Homo_sapiens_assembly38.fasta #输入bam文件和genome(参考基因组)文件
-p chr:pos #直接到达这个基因的位置
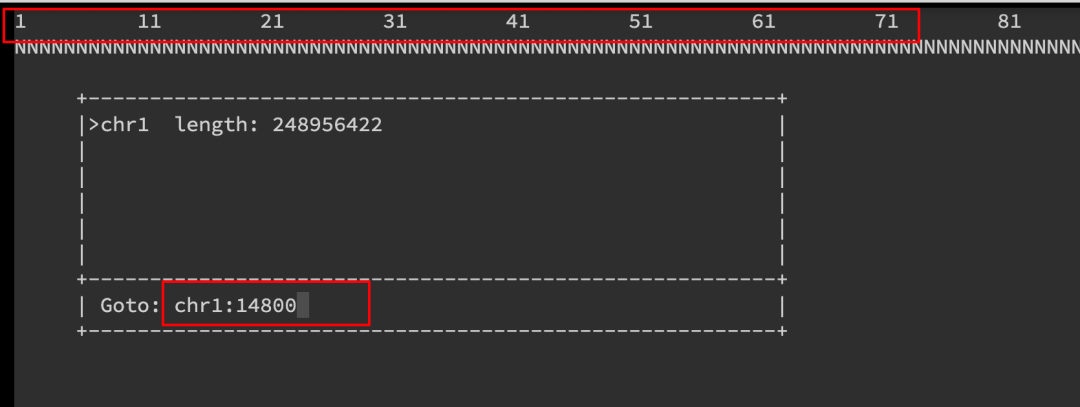
按g 输入位置
markdup
识别并标记那些在进行基因组坐标排序后被视为重复的比对记录(默认情况下并没有将它从sam文件中去除)
samtools markdup test.bam markdup.bam #是在duplicate read上标注,并没有将它从sam文件中去除
samtools markdup -r test.bam markdup.bam #将duplicate read从sam文件中去除
-@ #指定线程数
-r #删除重复读取
-T #指定临时文件前缀,将临时文件写入prefix.samtools.nnnn.nn.tmp
-l #最大读取长度(默认300个碱基)
-s #打印基本的统计信息
-f #将统计数据写到指定文件
merge
用于合并多个已排序的比对文件,生成一个包含所有输入记录的单一排序输出文件,同时保持现有的排序顺序。输出文件可以用-o指定。如果没有使用-h选项,输入文件的@SQ头部将被合并为一个综合头部。
samtools merge -@ 4 -o merge_out.bam test_sort.bam test2_sort.bam
-@ #指定线程数
-o FILE:#将合并输出写入FILE
-b FILE:#输入BAM文件列表,每行一个文件
-f:#如果输出文件已存在,强制覆盖
-h FILE:#使用FILE中的行作为输出文件的`@`头部
-R STR:#仅合并指定区域STR的文件。
-c :#当多个输入文件包含相同ID的@RG头部时,仅输出第一个。
-p :#对于每个@PG ID,仅使用第一个文件中的@PG行。
-L FILE:#用BED文件指定合并执行的多个区域
mpileup
mpileup以前为pileup;用于对bam文件进行处理,生成mpileup, VCF或BCF文件,再使用bcftools或varscan2进行SNP和Indel变异位点的检测(相较于GAT流程,其灵敏度并不高,不建议使用,在此不做演示)
注:另有更多用法,请参考官方文档:https://www.htslib.org/doc/samtools.html
参考:
- https://www.htslib.org/
- https://www.htslib.org/doc/samtools.html
- https://zhuanlan.zhihu.com/p/89896205
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-12-12,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

