DSSP教程:一步步教你预测蛋白质二级结构
最近在帮实验室的学姐分析一些蛋白质序列,然后就接触到了DSSP这个算法。于是写一篇小笔记,仅此来记录一下本次的使用记录。
一.DSSP简介
背景介绍:
二级结构是由生物大分子在原子分辨率结构中所观察到的氢键来定义的。蛋白质的二级结构通常是以主链中氨基之间的氢键模式来定义〈与主链-侧链间以及侧链-侧链间的氢键无关〉,亦即DSSP的定义。而核酸的二级结构是以碱基之间的氢键来定义。
课外知识:
对生物大分子的二级结构含量可以以光谱来初步估计。对于蛋白质,最常用的方法是圆二色性(Circular dichroism), (利用长紫外线,波长范围170-250nm)。在获得的光谱吸收曲线上,α螺旋结构会在208nm及222nm两处同时出现极小值,而204nm和207nm处出现单个极小值则分别表示存在无规卷曲和β折叠结构。
DSSP 是用于对蛋白质结构中的氨基酸残基进行二级结构构像分类的标准化算法,由Wolfgang Kabsch和Chris Sander设计。
——来自百度百科
The DSSP algorithm is the standard method for assigning
secondary structure to the amino acids of a protein,
given the atomic-resolution coordinates of the protein.
The abbreviation is only mentioned once in the 1983 paper
describing this algorithm,[2] where it is the name of the
Pascal program that implements the algorithm
Define Secondary Structure of Proteins. DSSP---来自维基百科
DSSP也有一个数据库:
DSSP数据库是由此算法生成的一个存放蛋白质二级结构分类数据的数据库,其中包括了PDB数据库(Protein Data Bank)中的所有条目。算法名称Define Secondary Structure of Proteins由作者在其原始论文中,作为实现该算法的Pascal语言程序名称所提及。DSSP数据库英文全称则为Definition of Secondary Structure of Proteins。
二.DSSP软件
GIthub地址:
https://github.com/PDB-REDO/dssp
项目介绍:
但是这个dssp在windows是无法编译和安装的,亲测多次失败。然后在ubuntu服务器上测试安装成功:
安装指令:
sudo apt-get install dssp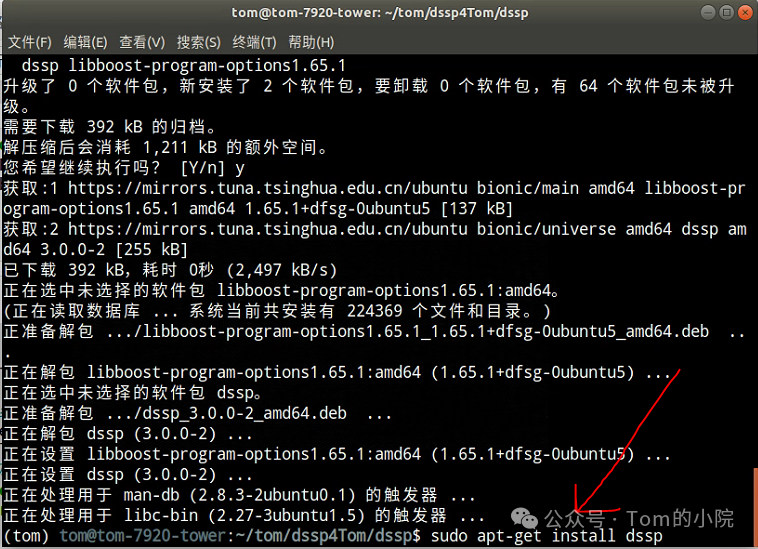
然后就安装成功了
三.使用dssp对蛋白质序列进行分析,
这里的31条蛋白质序列,我已经使用alphafold进行了预测,所以已经得到其pdb文件。所以这次我们的分析,是建立在pdb文件的基础上的。
1.导包
from Bio.PDB import PDBParser
from Bio.PDB.DSSP import DSSP
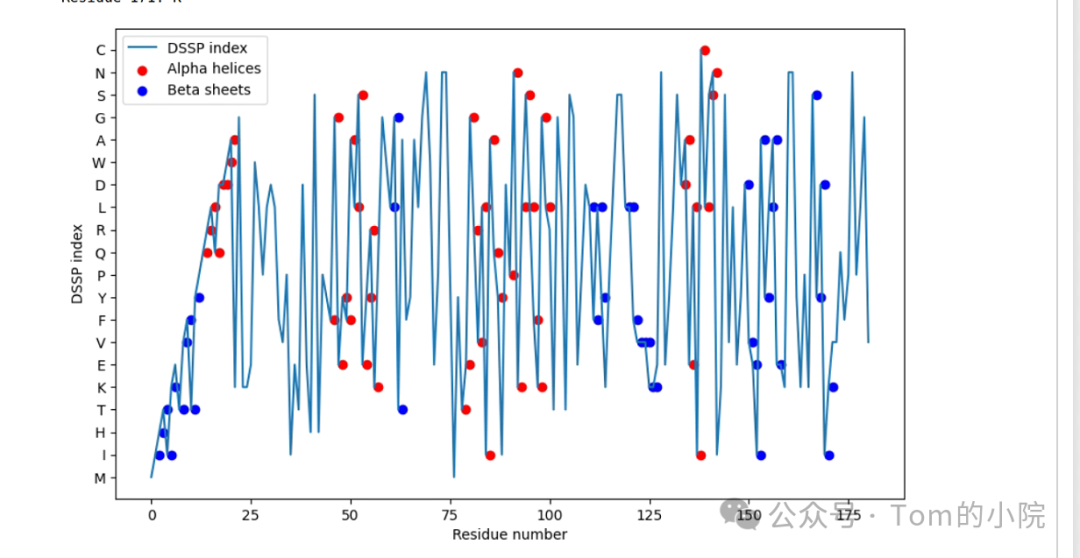
import matplotlib.pyplot as plt2.开始分析蛋白质文件,这里我们主要实现了打印出蛋白质对应的结构的片段序列。例如,下列代码实现了打印一个pdb文件中所有阿尔法螺旋的片段,还有贝塔折叠的片段信息。最后使用dssp的指标进行画图,并且在图中标注,做到可视化的功能。
# 解析PDB文件
p = PDBParser()
structure = p.get_structure("protein_name", "/home/01.pdb")
# 使用DSSP分析二级结构
model = structure[0]
dssp = DSSP(model, "/home/01.pdb")
# 提取阿尔法螺旋和贝塔折叠的片段
helices = [res for res in dssp if res[2] == 'H'] # 阿尔法螺旋
sheets = [res for res in dssp if res[2] == 'E'] # 贝塔折叠
# 打印片段信息
print("Alpha helices:")
for res in helices:
print(f"Residue {res[0]}: {res[1]}")
print("
Beta sheets:")
for res in sheets:
print(f"Residue {res[0]}: {res[1]}")
# 可视化
plt.figure(figsize=(10, 6))
plt.plot([res[1] for res in dssp], label='DSSP index')
plt.scatter([res[0] for res in helices], [res[1] for res in helices], color='r', label='Alpha helices')
plt.scatter([res[0] for res in sheets], [res[1] for res in sheets], color='b', label='Beta sheets')
plt.xlabel('Residue number')
plt.ylabel('DSSP index')
plt.legend()
plt.savefig("01.png")
plt.show()
首先程序的输出是阿尔法螺旋的氨基酸片段:

然后是贝塔折叠的片段:
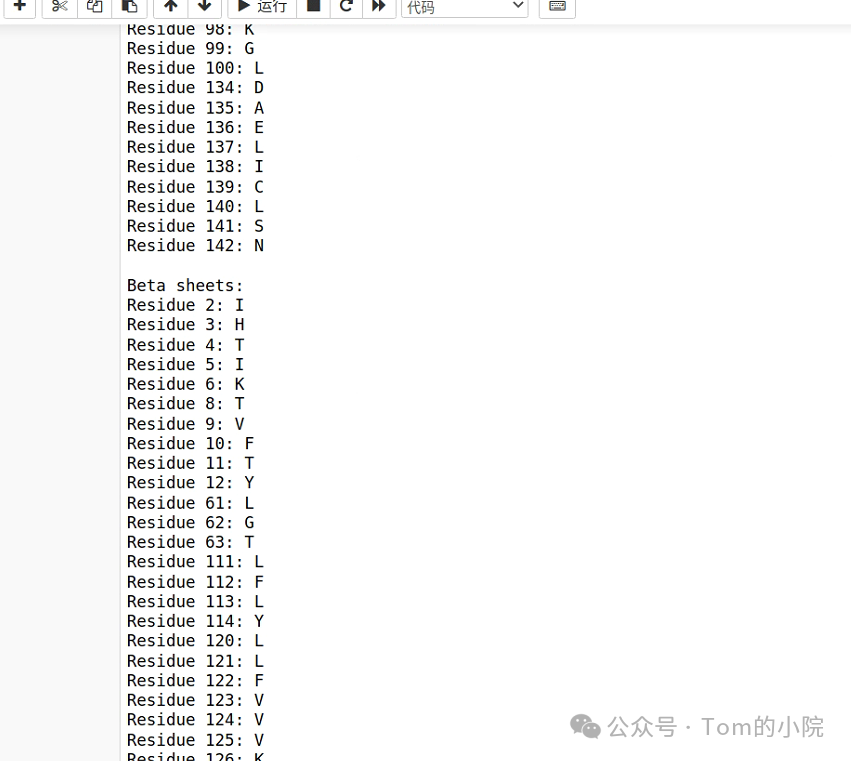
最后是一个可视化的图,显示了哪些片段是对应的结构:
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-03-19,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读


