scRNA|使用scMetabolism完成单细胞代谢激活分数估计
scRNA|使用scMetabolism完成单细胞代谢激活分数估计

之前介绍过 scRNA分析|使用AddModuleScore 和 AUcell进行基因集打分,然后可视化目标基因集合的打分 ,这里介绍scMetabolism包-整合了多个可以完成细胞代谢相关通路评估方法的R包。
一 载入R包,数据
首先根据官网GitHub - wu-yc/scMetabolism: Quantifying metabolism activity at the single-cell resolution的介绍安装相关的R包,需要注意的是VISION要安装v2.1.0版本。
然后使用之前注释过的sce.anno.RData数据 ,为节省资源,每种细胞类型随机抽取30%的数据。
install.packages(c("devtools", "data.table", "wesanderson", "Seurat", "devtools",
"AUCell", "GSEABase", "GSVA", "ggplot2","rsvd"))
#Please note that the version would be v2.1.0
devtools::install_github("YosefLab/VISION@v2.1.0")
devtools::install_github("wu-yc/scMetabolism")
# 加载R包
library(scMetabolism)
library(tidyverse)
library(rsvd)
library(Seurat)
library(pheatmap)
library(ComplexHeatmap)
library(ggsci)
# 加载数据
load("sce.anno.RData")
sce2@meta.data$CB <- rownames(sce2@meta.data)
# 按照细胞类型抽取一定比例的数据
sample_CB <- sce2@meta.data %>%
group_by(celltype) %>%
sample_frac(0.3)
# 提取数据
sce3 <- subset(sce2,CB %in% sample_CB$CB)
head(sce3,2)
二 计算代谢得分
该包比较简单,主函数可以选择sc.metabolism.Seurat 输入Seurat的单细胞对象(推荐),也可以选择 sc.metabolism 输入矩阵(作者不太建议)。
Idents(sce3) <- "celltype"
countexp.Seurat <- sc.metabolism.Seurat(obj = sce3, #Seuratde单细胞object
method = "AUCell",
imputation = F,
ncores = 2,
metabolism.type = "KEGG")其中obj是一个包含 UMI 计数矩阵的 Seurat 对象,记得指定Idents 。
method支持VISION、AUCell、ssgsea和gsva四种,默认的是VISION 方法。
metabolism.type支持KEGG和REACTOME,分别对应不同的代谢相关通路。
1,查看函数
可以用过View(sc.metabolism.Seurat) 查看函数的主体,结构还是比较清楚的,(1)预设了KEGG和REACTOME中代谢相关通路,(2)根据VISION、AUCell、ssgsea和gsva 四种常见方法计算代谢通路相关的得分。
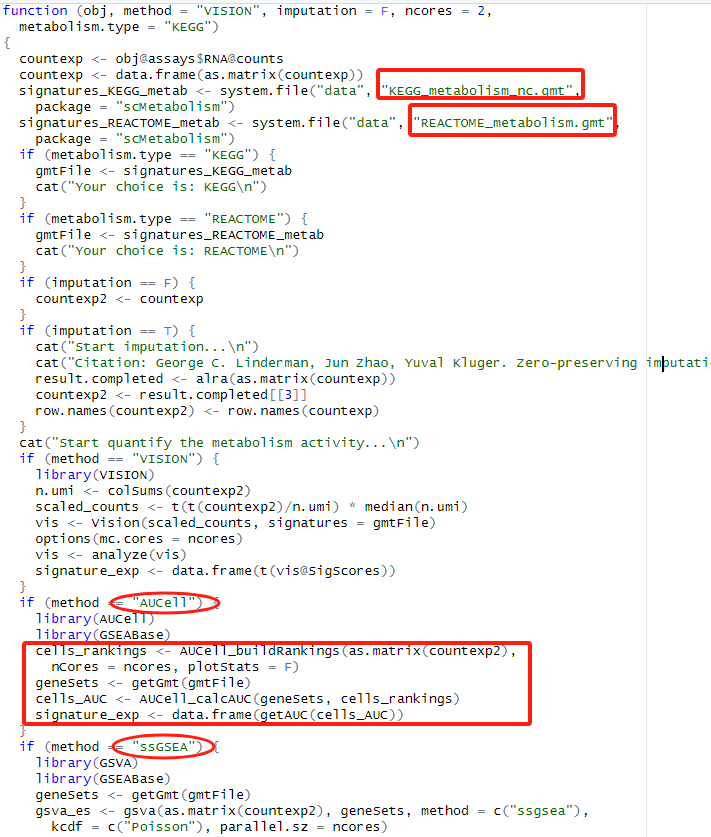
注:gmt可以改为你课题需要的通路,然后放到signatures_KEGG_metab输出的路径下。
也可以如 scRNA分析|使用AddModuleScore 和 AUcell进行基因集打分使用相关方法的函数直接计算 。
signatures_KEGG_metab <- system.file("data", "KEGG_metabolism_nc.gmt",
package = "scMetabolism")
signatures_KEGG_metab
#[1] "C:/Users/XXX/AppData/Local/R/win-library/4.3/scMetabolism/data/KEGG_metabolism_nc.gmt"2,提取结果-添加至meta信息
代谢评分的结果存放在新的assays -- METABOLISM中 ,可以通过如下方式得到每个基因的代谢通路的活性分数。
如截图所示细胞barcode的"-1"变为了".1",通过str_replace_all简单处理后添加至meta中,以备后面可能的相关分析。
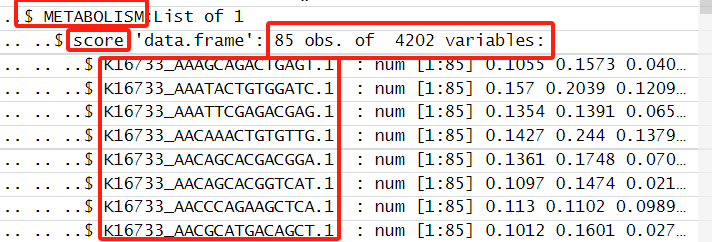
#提取score结果
score <- countexp.Seurat@assays$METABOLISM$score
score[1:4,1:4]
#将score中barcode的点转为下划线
score_change <- score %>%
select_all(~str_replace_all(., "\\.", "-")) #基因ID不规范会报错,下划线替换-
#确定细胞barcode椅子
identical(colnames(score_change) , rownames(countexp.Seurat@meta.data))
#[1] TRUE
countexp.Seurat@meta.data <- cbind(countexp.Seurat@meta.data,t(score_change) )
#可以直接使用Seurat的相关函数
p1 <- FeaturePlot(countexp.Seurat,features = "Glycolysis / Gluconeogenesis")
p2 <- VlnPlot(countexp.Seurat,features = "Glycolysis / Gluconeogenesis")
p1 + p2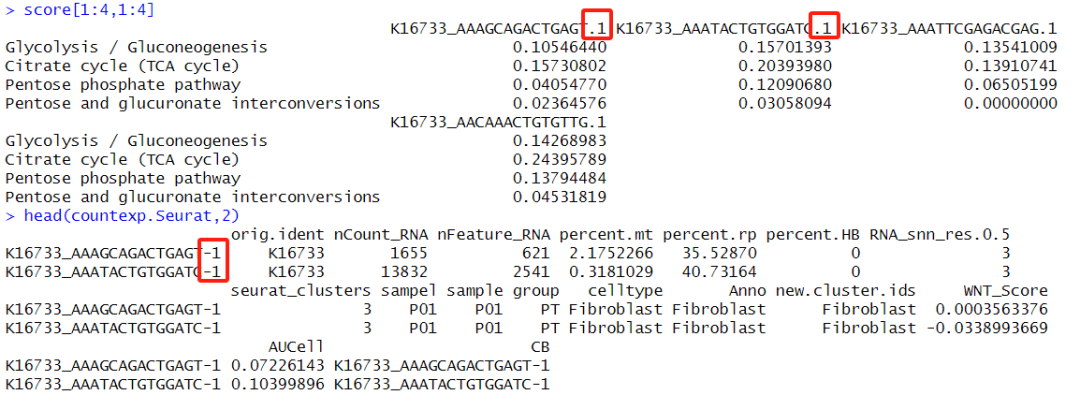
三 可视化
可以使用scMetabolism自带的函数完成一些可视化展示。
1,umap展示某条通路的代谢得分
DimPlot.metabolism(obj = countexp.Seurat,
pathway = "Glycolysis / Gluconeogenesis",
dimention.reduction.type = "umap",
dimention.reduction.run = F, size = 1)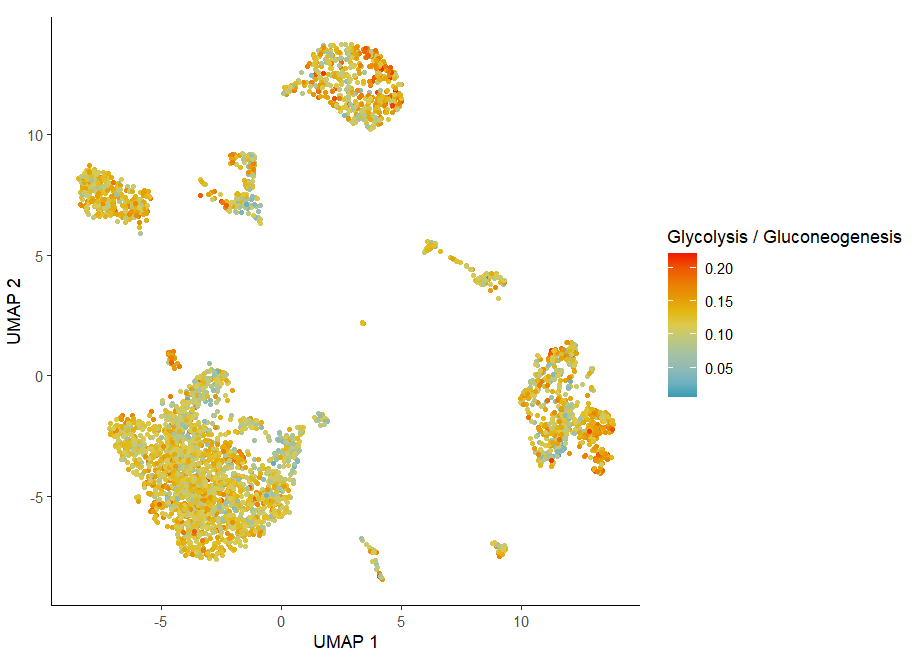
2,指定通路-细胞类型点图
可以选择直接指定目标通路 或者 展示前几个,注意将phenotype 参数改为需要展示的列。
#直接指定
input.pathway<-c("Glycolysis / Gluconeogenesis",
"Oxidative phosphorylation",
"Citrate cycle (TCA cycle)")
#展示前10个
input.pathway <- rownames(countexp.Seurat@assays$METABOLISM$score)[1:10]
DotPlot.metabolism(obj = countexp.Seurat,
pathway = input.pathway,
phenotype = "celltype", #更改phenotype 参数
norm = "y")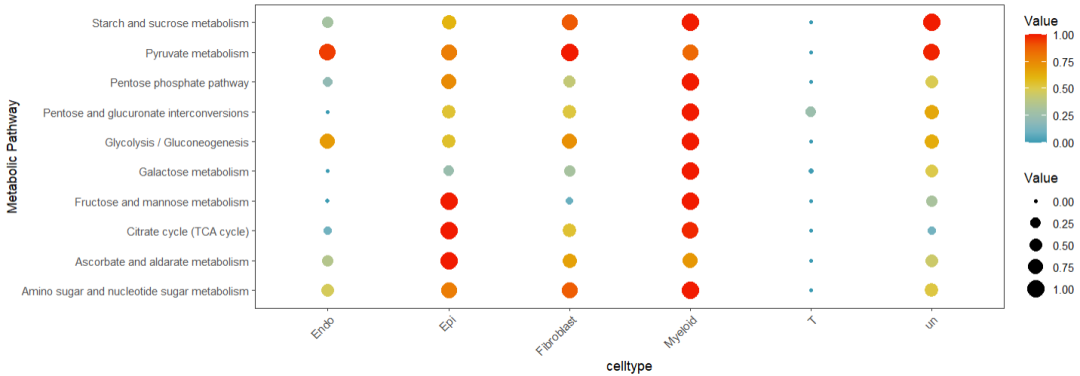
3,指定通路-箱线图
可以使用ggsci 包修改一下颜色
BoxPlot.metabolism(obj = countexp.Seurat,
pathway = input.pathway[1:4],
phenotype = "celltype",
ncol = 2) +
scale_fill_nejm()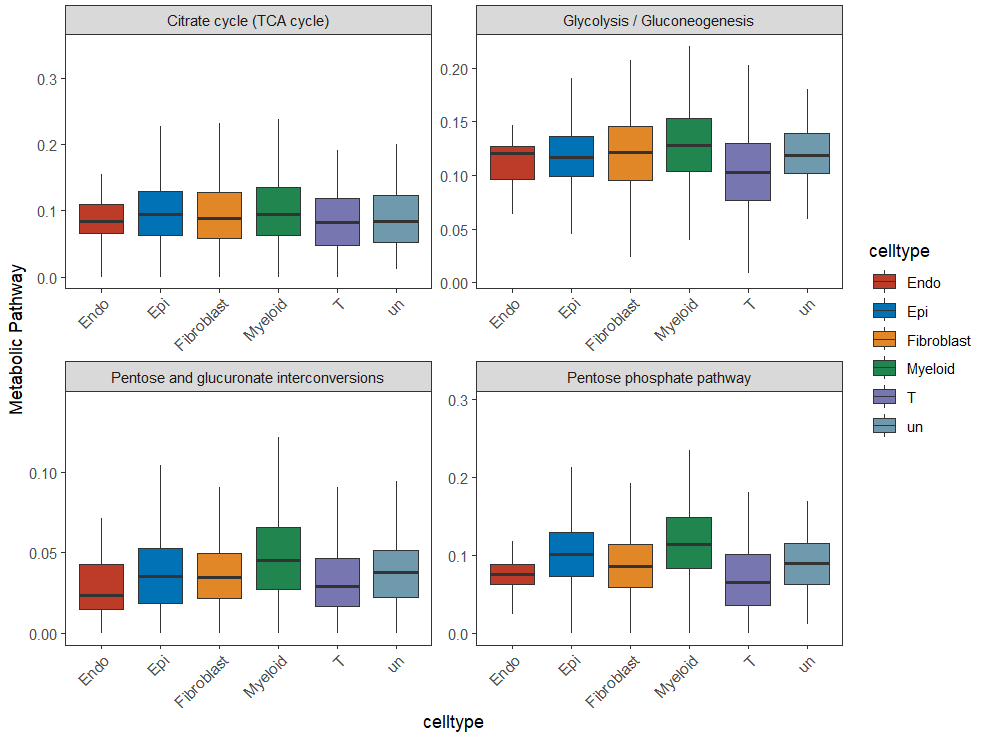
4,自定义热图
首先计算每种细胞类型的相关代谢通路得分的均值,然后可以使用pheatmap 直接绘制热图,或者参照scRNA|ComplexHeatmap自定义单细胞转录组celltype-level 热图可视化绘制复杂热图
#可以计算celltype均值,然后绘制
df <- countexp.Seurat@meta.data
#19列开始是代谢通路的得分,按照celltype计算均值
avg_df = aggregate(df[,19:ncol(df)],
list(df$celltype),
mean)
#热图需要转为矩阵
avg_df <- avg_df %>%
select(1:20) %>% #展示前20个
column_to_rownames("Group.1")
avg_df[1:4,1:4]
也可以手动选择想展示的代谢通路。
(1)直接pheatmap绘制
pheatmap(t(avg_df),
show_colnames = T,
scale='row',
cluster_rows = T,
color=colorRampPalette(c('#1A5592','white',"#B83D3D"))(100),
cluster_cols = T)(2)组合复杂热图
作为复杂热图的一个组件。为使图形更好看,我们先手动对数据进行标准化。
exp <- apply(avg_df, 2, scale)
rownames(exp) <- rownames(avg_df)
# 组件
h_state <- Heatmap(t(exp),
column_title = "state_gsva",
col = colorRampPalette(c('#1A5592','white',"#B83D3D"))(100),
name= "gsva ",
show_row_names = TRUE,
show_column_names = TRUE)
h_state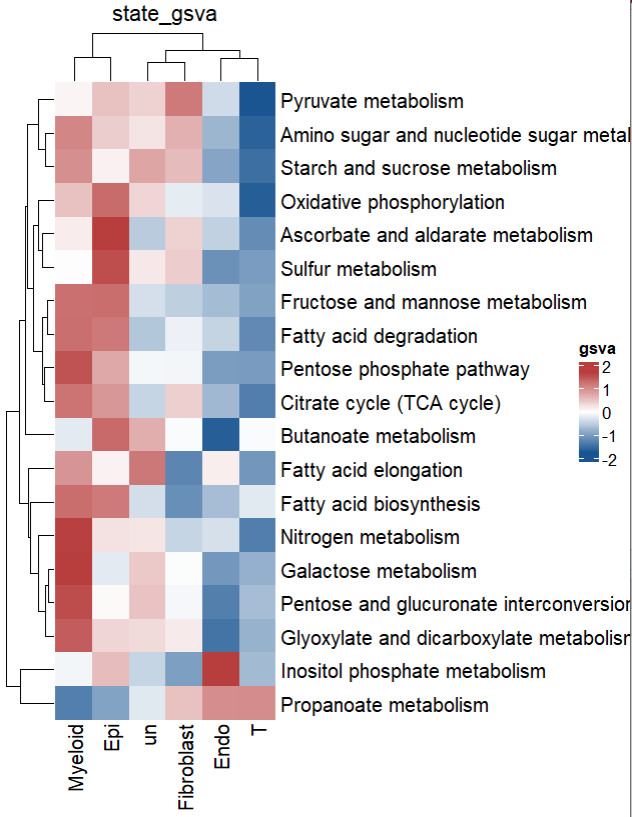
然后可以scRNA|ComplexHeatmap自定义单细胞转录组celltype-level 热图可视化添加更多的信息组合绘制下面的图 。
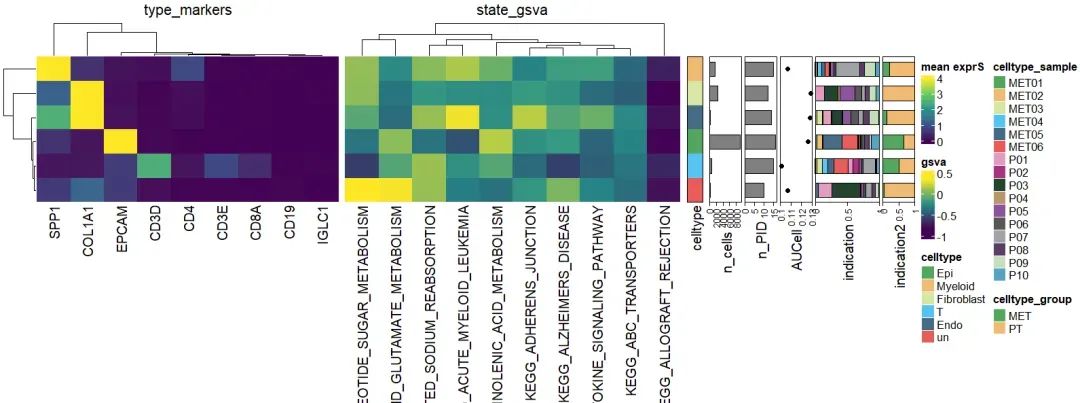
参考资料:
GitHub - wu-yc/scMetabolism: Quantifying metabolism activity at the single-cell resolution
◆ ◆ ◆ ◆ ◆

