String/GeneMANIA两种常用蛋白质网络预测工具数据库简单介绍及操作流程展示
原创String/GeneMANIA两种常用蛋白质网络预测工具数据库简单介绍及操作流程展示
原创
凑齐六个字吧
发布于 2024-08-28 16:20:33
发布于 2024-08-28 16:20:33
1. STRING (Search Tool for the Retrieval of Interacting Genes/Proteins)
STRING 是一个广泛使用的数据库和资源,主要用于预测和可视化蛋白质-蛋白质相互作用网络。它结合了实验数据、计算预测以及已知的生物学知识来预测蛋白质相互作用。
数据来源: STRING 的数据来源包括实验验证的相互作用、计算预测的相互作用、文献挖掘以及来自其他数据库的信息。
功能:
● 提供蛋白质之间的直接(物理)和间接(功能)相互作用信息。
● 允许用户查询特定基因或蛋白质的相互作用网络。
● 可视化相互作用网络并进行拓扑分析。
网址: STRING(https://string-db.org/)
2. GeneMANIA
GeneMANIA 是一个灵活且用户友好的工具,用于预测基因功能和构建基因相互作用网络。它通过整合多个来源的数据(包括基因共表达、蛋白质-蛋白质相互作用、遗传相互作用、通路、物理相互作用等),帮助研究人员找到与输入基因相关的基因。
数据来源: GeneMANIA 整合了多种数据类型,如基因共表达、蛋白质-蛋白质相互作用、遗传相互作用、共定位、路径、物理相互作用、蛋白质结构等。
功能:
● 快速生成基因相互作用网络,并通过多个网络权重模型进行预测。
● 提供可视化工具,可以查看输入基因及其相互作用伙伴的网络图。
● 支持多种生物物种。
网址: GeneMANIA(https://genemania.org/)
数据库操作流程
String
1、进入页面之后主要关注Search界面上的这两个红色方框内容。
左边红框可以选择是单一/多种蛋白输入模式,右边红框是输入蛋白(基因)名称并选择物种。
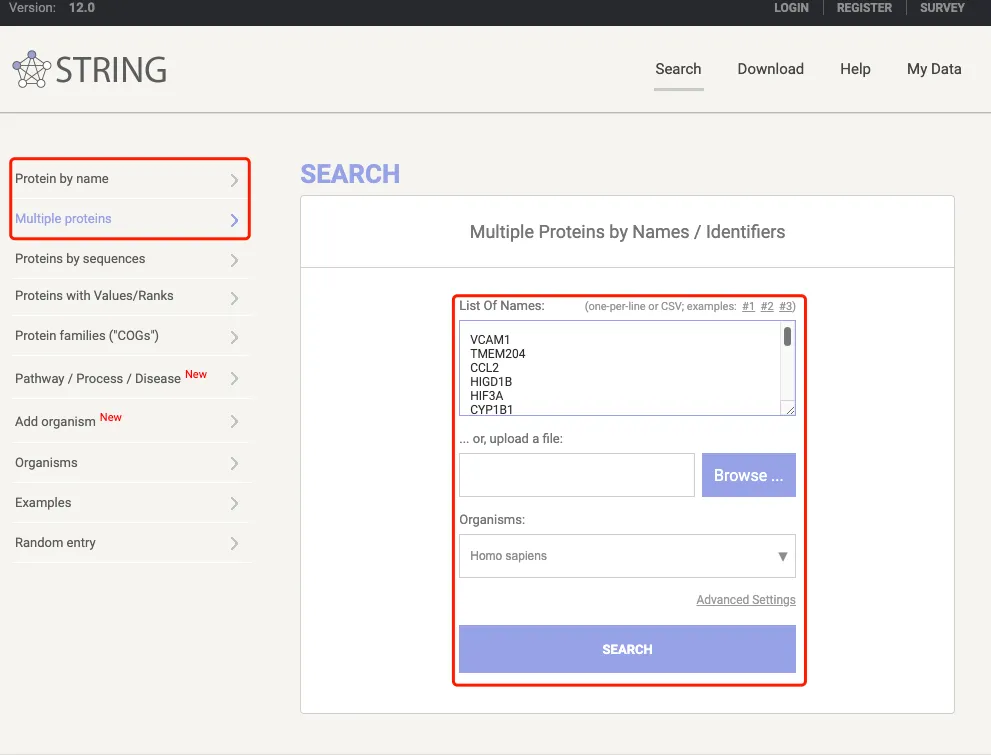
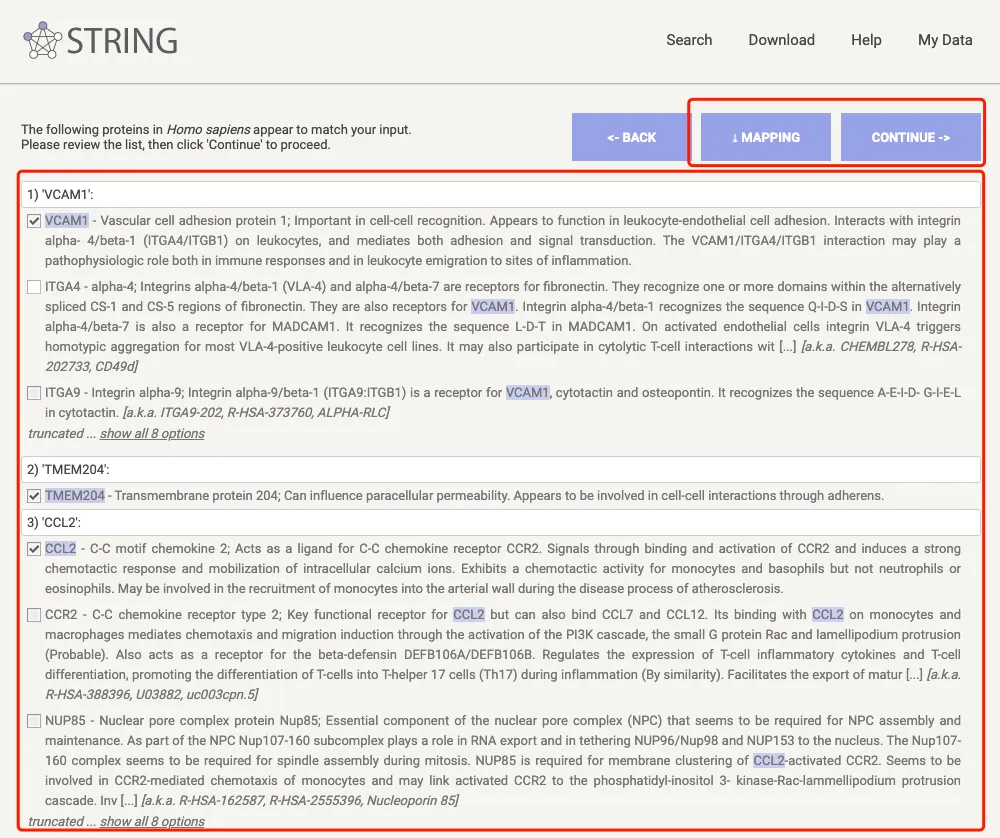
2、点击SEARCH后会进入下一步的界面,这里对输入的蛋白(基因)会有详细的解释。同时也可以把对应的信息从MAPPING处下载下来。
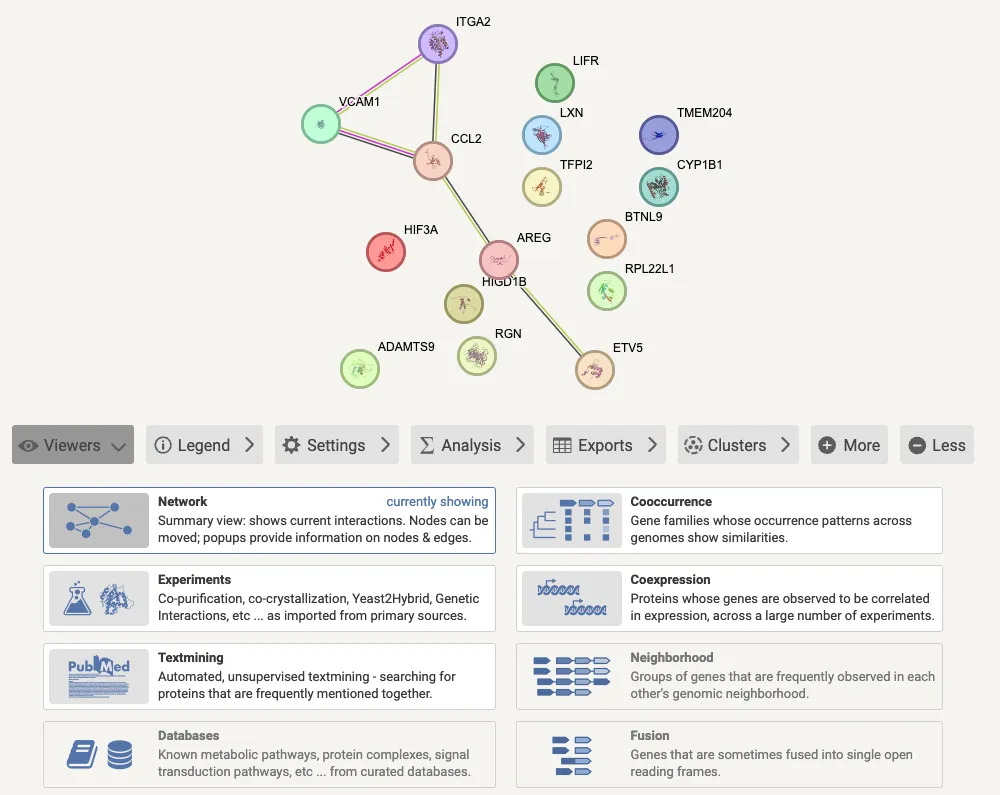
3、Viewers界面可以看到蛋白互作的证据来源。
4、Legend显示了网络中的核心内容信息。
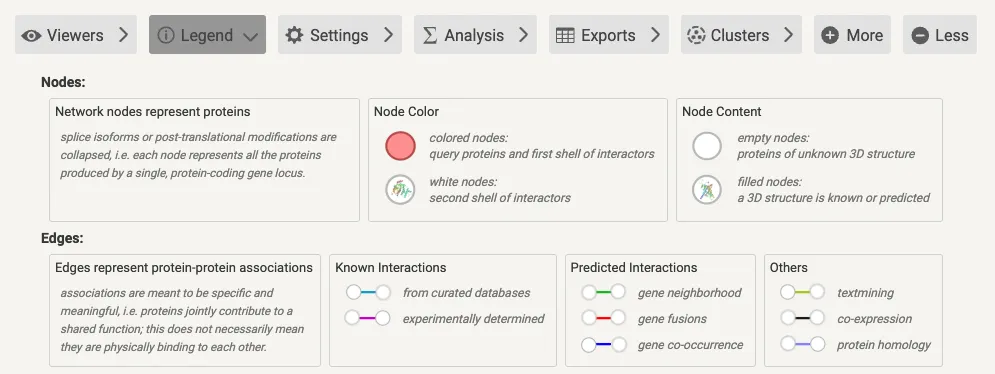
STRING数据库的网络图由两大元素组成:点(Nodes) 和 线(Edges)。
● 点(Nodes):每个点代表一个蛋白质,气泡内显示该蛋白的3D结构。点击气泡可以查看更多注释信息。不过,在这个网络图中,不同的剪接异构体或翻译后修饰的蛋白质被合并处理,每个点代表由同一基因位点产生的所有蛋白质变体。
● 线(Edges):连线表示蛋白质之间的相互作用(PPI)。STRING通过整合7种类型的证据(如基因邻域、基因融合、共现、实验数据、共表达、数据库信息、文本挖掘),对每对蛋白的互作关系进行评分。这些证据的连线颜色各不相同,且综合得分越高,表示该蛋白质互作关系的可信度越高。点击任意连线可以查看详细的互作信息和证据评分。
在PPI网络图中,不同颜色的连线代表了7种证据类型:
- 邻域(Neighborhood):根据基因在染色体上的位置,评估它们是否可能相互作用。
- 融合(Fusion):通过全基因组分析,找出融合基因,推测其他基因间可能的关联。
- 共现(Co-occurrence):在进化过程中,经常一起出现的基因对可能相互作用。
- 实验(Experiments):基于已发表的实验数据,直接证明蛋白质间的相互作用。
- 共表达(Co-expression):分析基因或蛋白质的表达模式,寻找表达相似的基因对。
- 数据库(Databases):利用已有的生物数据库信息,如通路和蛋白质复合物,来支持相互作用的可能性。
- 文本挖掘(Textmining):通过分析科学文献中的关键词,找到可能的蛋白质互作证据。
这些证据通过不同颜色的连线展示,相互作用的可信度通过综合得分来表示。 5、Exports界面可以把数据导出,后续可用比如Cytoscape等软件进一步分析
GeneMANIA
1、进入官网后发现界面很简洁,左上角可输入蛋白(基因)名称
2、输入名称并分析之后会在官网中间出现网络图。
右侧有网络类别的解释(示例内容仅出现了三种):
共表达(Co-expression):基因表达水平在不同条件下相似,表明它们可能相关联。数据主要来自GEO数据库。
物理相互作用(Physical Interaction):如果两个蛋白质在实验中被发现相互作用,它们就是相关联的。数据来自BioGRID和PathwayCommons。
遗传/基因相互作用(Genetic Interaction):一个基因的改变影响了另一个基因的功能,表明它们在功能上相关。数据来自基础研究和BioGRID。
共享蛋白域(Shared Protein Domains):两个基因产物具有相同的蛋白结构域,表明它们有关联。数据来自InterPro、SMART和Pfam。
共定位(Co-localization):基因在同一组织表达或其产物在同一细胞位置出现,表明它们相关联。
通路(Pathway):两个基因产物参与同一通路反应,表明它们相关联。数据来自PathwayCommons。
其他(Other):不属于上述任何类别的相互作用网络。

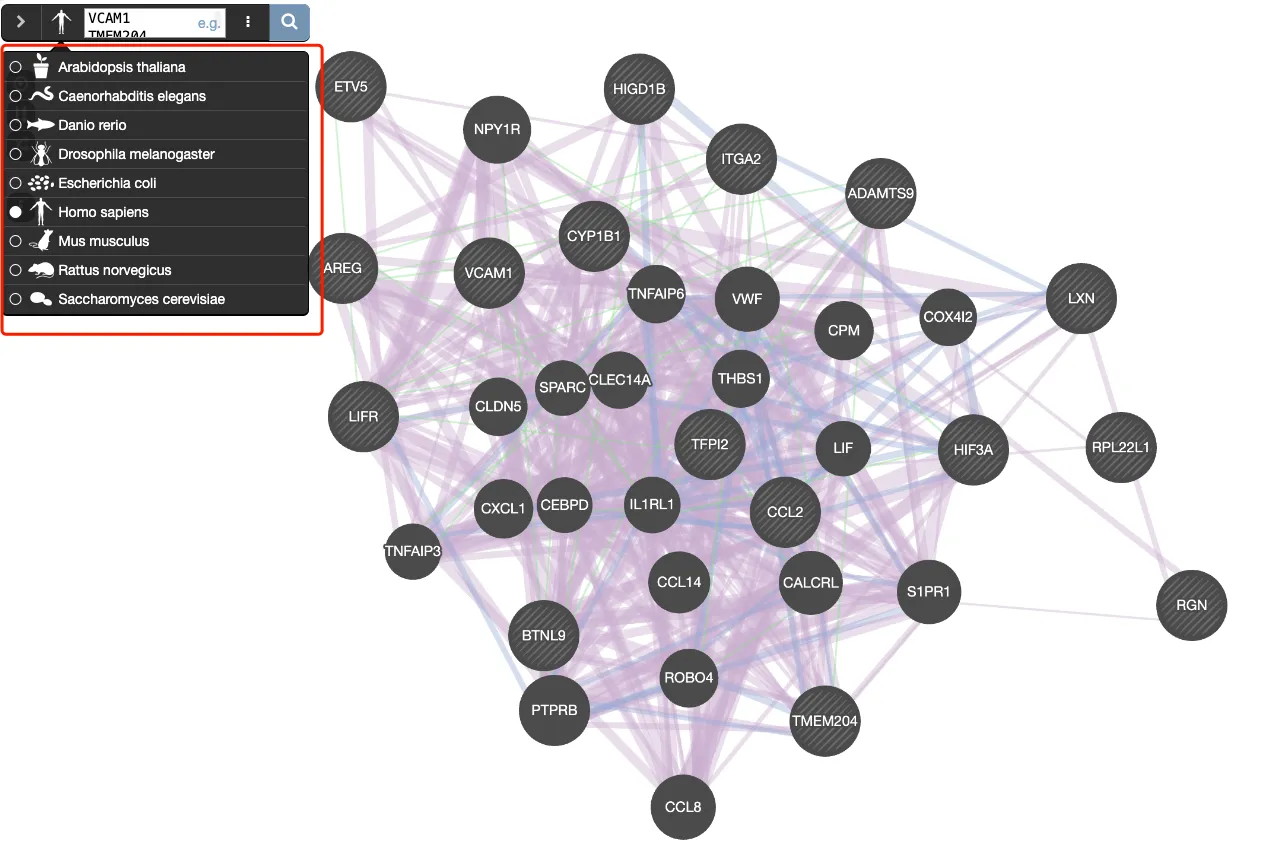
3、可以选择不同的物种
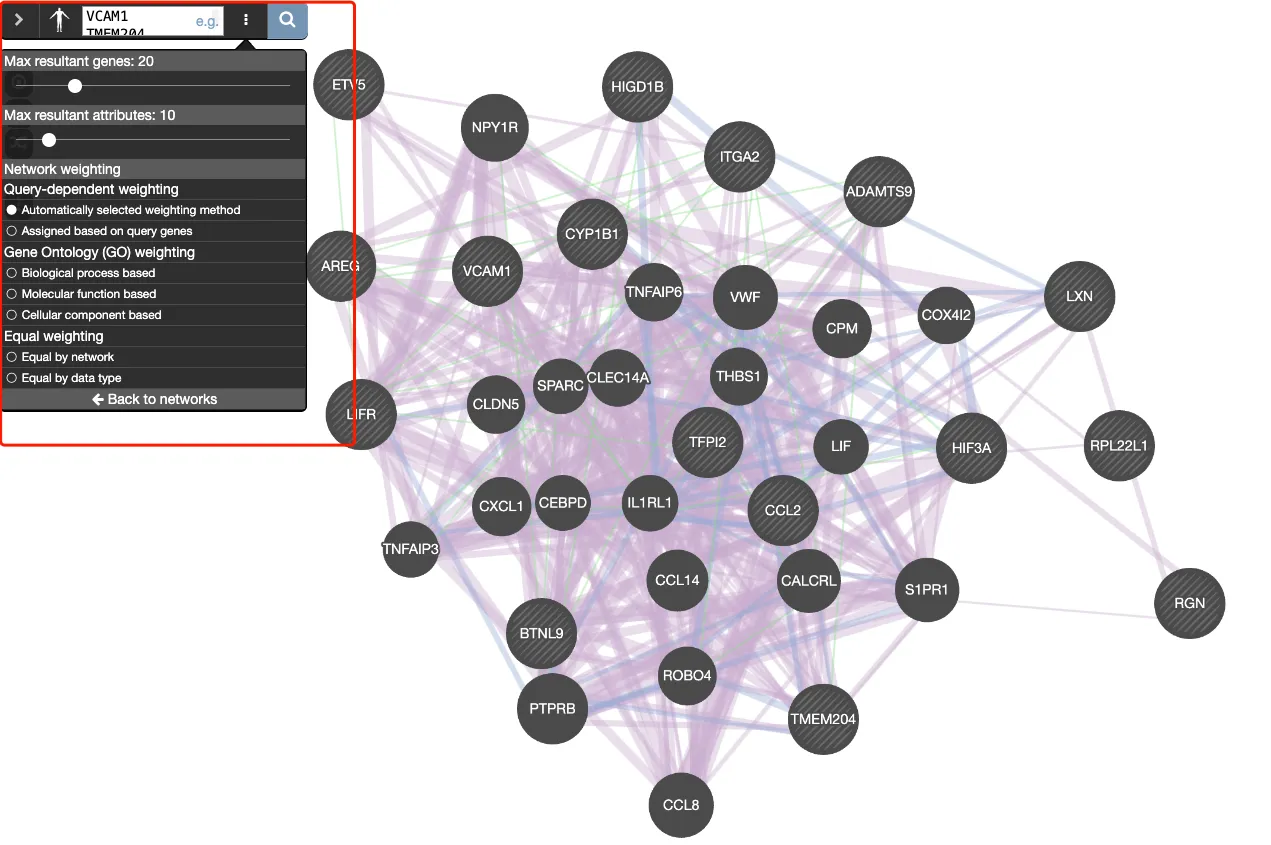
4、左上角也有高级选项可以选择,里面主要包括了三种加权分析的方法:
Query-dependent weighting(自动优化加权) :
● Automatically selected weighting (default):根据输入基因数量自动选择权重。如果输入基因少于5个,默认采用基于Gene Ontology (GO) 生物过程的加权方法;若输入基因在5个或以上,则采用线性回归方法,以最大化输入基因间的连接性。
● Assigned based on query:使用线性回归自动分配权重,使输入基因之间的相互作用最大化,同时减少与非输入基因的相互作用。
Ontology-based weighting(基于基因本体论加权) :
● Biological Process based:基因与GO生物过程相关。
● Molecular Function based:基因与GO分子功能相关。
● Cellular Component based:基因与GO细胞成分相关。
Equal weighting(均等加权) :
● Equal by network:为所有网络分配相等的权重,适合查看连接输入基因的所有网络。
● Equal by data type:为所有网络类别分配相等权重,并在类别内的网络中均匀分布。
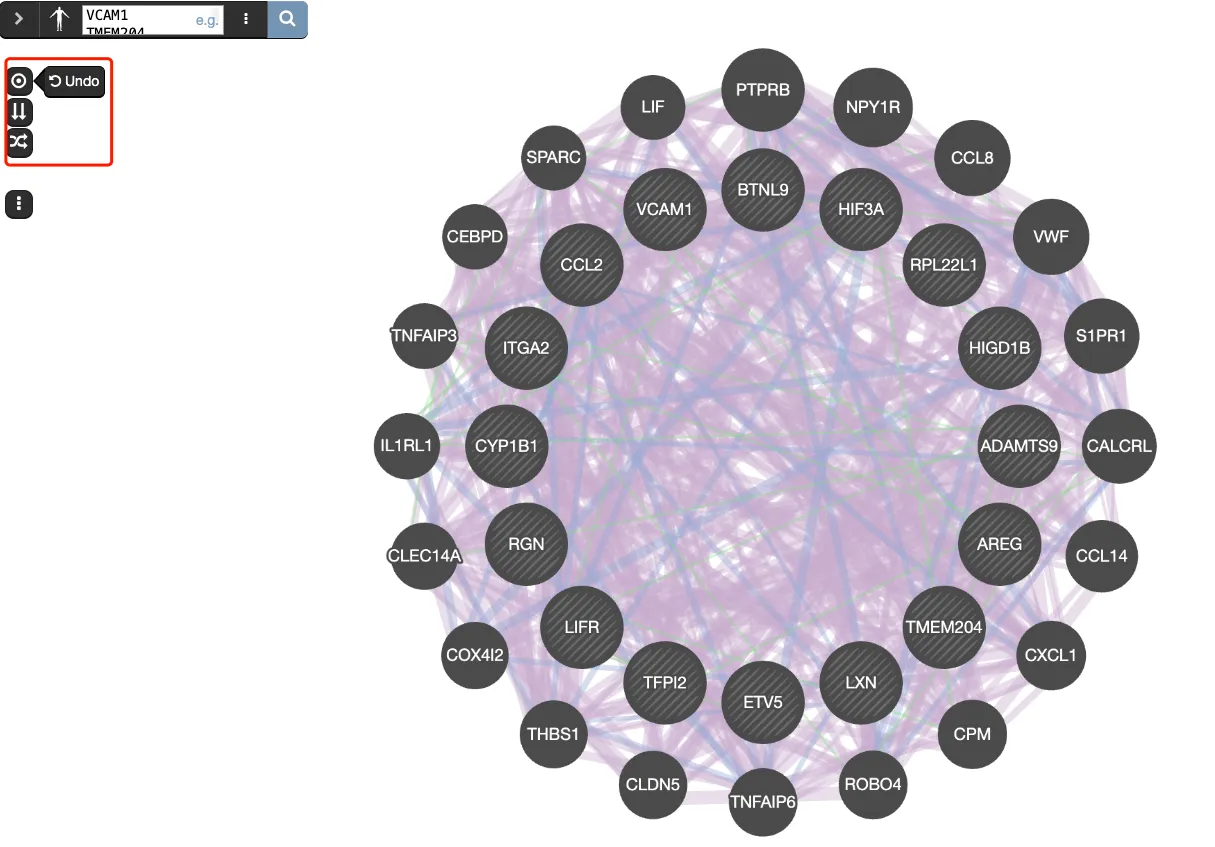
5、左边三个按钮可以让网络图产生不同的形状
6、Functions 选项显示了网络中基因的富集结果
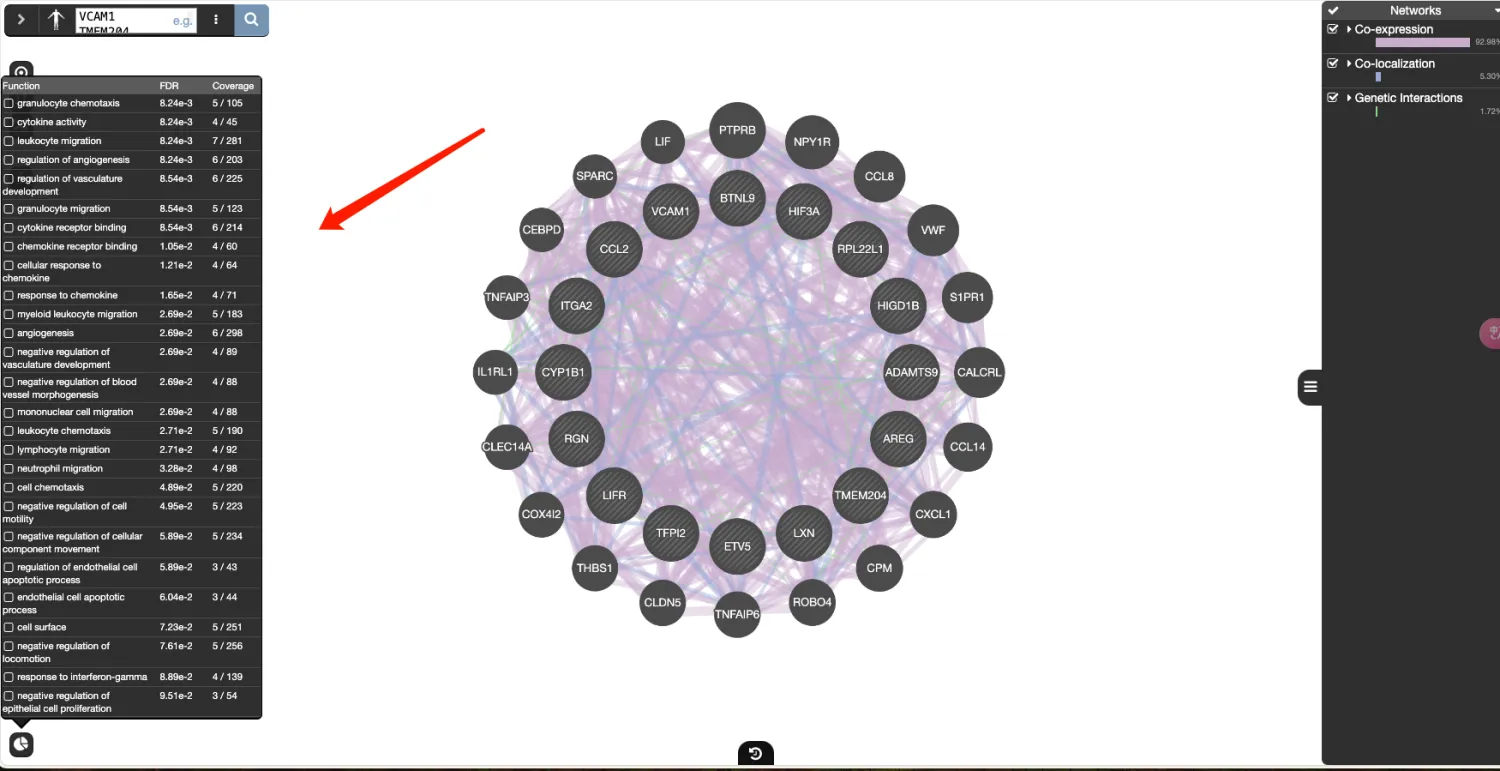
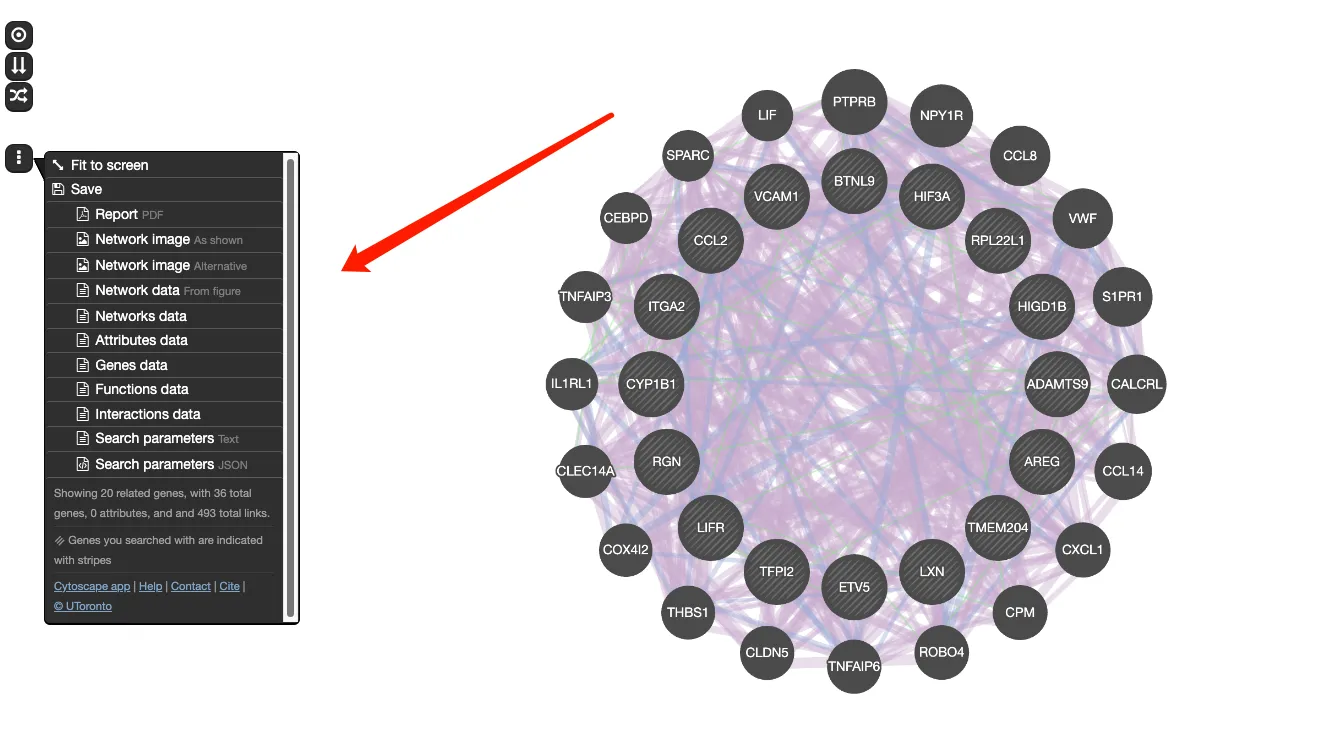
7、下载界面可以把数据导出进行进一步分析
注:若对内容有疑惑或者有发现明确错误的朋友,请联系后台(欢迎交流)。更多内容可关注公众号:生信方舟
- END -
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

