J Pharm Anal|化学自然语言引导基于扩散的生成式类药分子编辑
扩散模型已成为分子设计与优化中的一种前沿范式。然而,大多数基于扩散的分子生成模型主要聚焦于2D图形或3D几何结构的建模,对分子序列扩散模型的研究相对较少。国际纯粹与应用化学联合会 (IUPAC) 命名系统与化学自然语言更为相似,而非有机化合物的SMILES编码。
2024年11月2日发表在Journal of Pharmaceutical Analysis上的研究论文Diffusion-based generative drug-like molecular editing with chemical natural language提出了一种基于IUPAC名称引导的条件扩散模型DiffIUPAC,用于从化学自然语言到化学语言 (SMILES) 的分子编辑。

该研究探索了扩散模型的预训练生成性能能否迁移至化学自然语言领域。研究人员开发的DiffIUPAC是一种可控的分子编辑扩散模型,能够将IUPAC名称转换为SMILES字符串。评估结果显示,DiffIUPAC模型在语义规则捕捉方面优于现有方法,成功掌握了两种化学语言的语义规则。为了展示该模型在药物设计中的潜在应用,还进行了官能团编辑、类似物设计和Linker设计的案例研究。
背景
药物发现与开发对于克服人类疾病至关重要。在先导小分子药物的设计与优化阶段,往往需要探索庞大的类药化学空间。因此,高效搜索类药化学空间并筛选具有理想特性的药物仍是重大的挑战。传统的计算机辅助药物设计(CADD)技术,如相似性搜索、药效团模型、骨架跳跃、分子对接、定量构效关系(QSAR)模型、虚拟筛选和分子动力学等,被广泛应用于加速药物设计与筛选。随着硬件、计算方法和人工智能技术的发展,传统工具的性能得到了提升,并催生出新的研究范式。
生成式深度学习在药物发现和开发中正发挥着越来越重要的作用。深度生成模型在提取药物特征、化学空间搜索和分子优化方面展现了巨大潜力。这些模型旨在捕捉训练集中分子及其性质的概率分布,提取独特的分子表示,并最终生成具有理想特性的创新分子。现有分子表示方法包括分子指纹、分子描述符、简化分子线性输入系统(SMILES)、分子图、国际纯粹与应用化学联合会(IUPAC)命名、IUPAC国际化学标识符(InChI)、分子图像和基于神经网络的表示。基于这些分子表示,分子生成模型可分为SMILES字符串生成模型、IUPAC名称生成模型、分子图生成模型、分子图像生成模型和3D分子生成模型。目前,深度分子生成模型主要采用序列(SMILES)和分子图进行训练。尽管SMILES字符串便于训练,但缺乏3D信息;分子图可以弥补这一缺陷,但涉及高维信息处理。两种表示方式虽然适合机器处理,但对人类而言难以精确编辑。
近年来,大型语言模型(LLMs)在化学、药物发现、药物设计与优化以及材料科学等领域表现出色。LLMs在理解和生成类人文本方面具有显著潜力,有助于更直观、有效的人机交流。它们能协助解读科学文献、自动化化合物设计,并基于自然语言描述预测分子特性。这一能力至关重要,因为它让研究人员能够使用自然语言与计算模型互动,从而提升药物发现过程的可及性和效率。目前,分子主要通过化学语法(SMILES字符串)和化学自然语法(IUPAC名称)来描述。SMILES字符串广泛应用于分子的序列表示,设计上便于计算机化学信息处理;而IUPAC命名在有机化学中发挥重要作用,许多化学期刊和规范性协议要求对发表的有机结构使用IUPAC名称。图1展示了同一分子的SMILES字符串和IUPAC名称之间的差异。SMILES字符串通过原子和键表示分子,不适合随意添加或人类修改,而IUPAC名称由字母数字和特殊字符组成,更接近自然语言,便于人类理解和操作。基于Transformer架构的IUPAC名称生成模型已经存在。IUPAC名称允许领域专家根据具体应用编辑分子,是一种对人类友好的化学自然语言,便于片段或亚结构的编辑与设计。
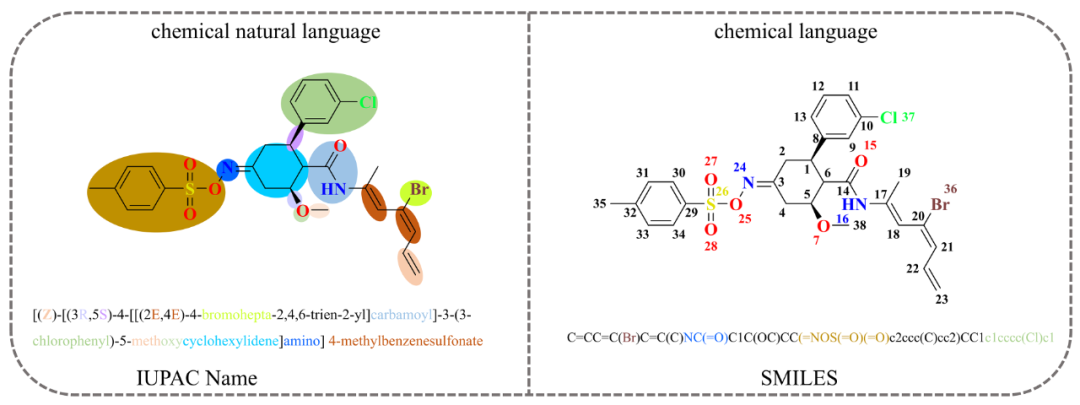
图1. 图示展示了同一分子在国际纯粹与应用化学联合会 (IUPAC) 命名和简化分子线性输入系统 (SMILES) 表示之间的差异。
深度分子生成模型可大致分为六类:自回归模型(包括循环神经网络(RNN)、长短期记忆(LSTM)网络和Transformer模型)、变分自编码器(VAEs)、生成对抗网络(GANs)、流模型、扩散模型和混合模型。近年来,扩散模型作为新兴生成建模范式在生物信息学、计算生物学、分子设计以及图像和视频生成中取得了显著成功。然而,大多数基于扩散的分子生成模型主要聚焦于2D图或3D几何结构,针对序列的扩散模型研究较少。尽管已有基于SMILES的扩散分子生成模型,但尚未出现基于IUPAC名称的扩散生成模型。结合条件扩散模型与化学自然语言和化学语言的序列到序列分子生成模型,将有助于弥合扩散模型与分子编辑之间的差距。
研究人员利用条件扩散模型进行从化学自然语言到化学语言的分子编辑,探索扩散模型的卓越生成性能是否能迁移到化学自然语言领域。我们提出了DiffIUPAC,一种可控的分子编辑扩散模型,能够从IUPAC名称生成SMILES字符串。结果显示,我们提出的模型优于参考方法,成功捕捉了两种化学语言的规则及其映射关系。DiffIUPAC能够基于给定的IUPAC名称及其遮蔽部分生成分子结构。化学空间与骨架分析表明,该模型在特定约束条件下生成具有多样骨架的相似分子。在蛋白质-蛋白质相互作用(PPI)调节剂的类似物设计和连接体编辑中,DiffIUPAC生成了候选化合物,具有更好的类药性和对接亲和力。案例研究展示了DiffIUPAC在类似物设计和Linker编辑中的优势和应用潜力。
数据与方法
数据准备和Tokenizer
所有分子的规范化SMILES字符串和IUPAC名称对均从PubChem数据库获取。数据清洗之后,使用RDKit计算了所有分子的以下理化性质:分子量 (MolWt)、正辛醇/水分配系数 (LogP)、可旋转键 (ROTB)、氢键供体 (HBD)、氢键受体 (HBA)、拓扑极性表面积 (TPSA)、合成可达性评分 (SAscore) 和药物相似性定量估计 (QED)。为获得高质量的类药数据集,研究人员按照以下标准进行筛选:100≤MolWt≤900、-5≤LogP<8、SAscore<4、ROTB<10、HBD<5、HBA<10、TPSA<150和QED≥0.3。
研究人员首先对序列进行了分词处理。分词器将IUPAC名称和SMILES字符串分解为可作为离散元素处理的信息块,从而使化学信息的分析和建模更加高效和准确。研究人员构建了一个基于规则的IUPAC分词器,将IUPAC名称转换为表示片段、官能团、子结构、立体化学和分子的词元。IUPAC分词器排除了非常罕见或不重要的名称,排除标准基于其在化学信息学中的频率和相关性,确保研究重点集中在重要且常见的化学结构上。对于SMILES字符串,我们采用基于字符的分词方式。
模型架构
扩散模型是一种生成模型,包括一个迭代的前向扩散过程和一个学习得到的逆扩散过程。在前向扩散过程中,随机噪声逐渐加入真实样本中,逐步破坏训练数据,直到真实样本变得不可识别。在逆扩散过程中,随机噪声被逐步去噪以恢复真实样本。训练完成后,模型可以通过从正态分布(随机噪声)生成特征,并对这些特征进行去噪,来创建新的高质量样本。
研究人员提出的受控分子编辑扩散模型DiffIUPAC。为了将扩散模型应用于IUPAC名称到SMILES字符串的设置中,使用编码器-解码器Transformer架构扩展了SeqDiffuSeq文本扩散模型。
前向扩散过程逐步向真实的SMILES数据添加噪声,使其逐渐变为随机噪声,以便在训练中破坏数据结构。这一过程将SMILES字符串通过嵌入函数映射为向量表示,并在每一步加入高斯噪声,最终生成完全随机的噪声序列,为后续的逆向扩散提供基础。
逆向扩散过程通过学习到的去噪过程逐步去除噪声,以恢复原始SMILES序列或生成新的SMILES样本。该过程条件化于输入的IUPAC名称,以确保生成的SMILES与输入相关联。通过在每个时间步从噪声状态逐步去噪,逆向扩散最终恢复或生成具有高保真度的SMILES序列。
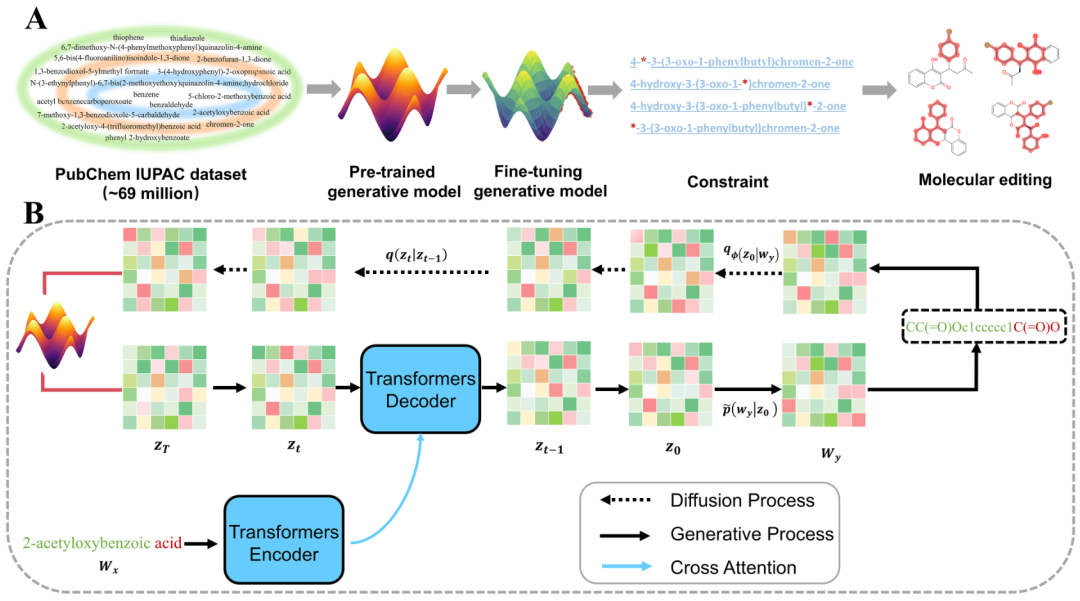
图2:DiffIUPAC架构概述
基线方法
研究人员将DiffIUPAC与以下基线方法进行了比较:
C5T5:C5T5使用IUPAC名称作为分子表示,通过变体条件Transformer训练一个自监督的预训练T5模型,以实现有机分子的控制生成。C5T5首先生成新的IUPAC名称,然后将其转换为SMILES字符串。
TransAntivirus:TransAntivirus通过连接两个变体条件Transformer模型,根据IUPAC名称生成SMILES字符串。
iupacGPT:iupacGPT使用IUPAC名称作为分子表示,基于GPT-2化学自然模型开发轻量级的分子生成和性质预测模型。
DiffSeqMol:DiffSeqMol以SMILES字符串作为分子表示,提出了基于扩散模型的分子序列到序列生成模型。
结果与讨论
可控制的分子生成
为了展示DiffIUPAC如何让用户和领域专家根据自身直觉和具体需求选择分子的编辑位置,用户可以通过在IUPAC名称中用“”替换可以修改的位置。模型在填充完整分子的同时,会自动替换局部子结构。这种功能在药物发现中的先导化合物优化等任务中非常有用。我们以DKY709为例,这是一种用于癌症免疫治疗的选择性IKZF2分子胶降解剂。DKY709的IUPAC名称为3-[6-(1-benzylpiperidin-4-yl)-3-oxo-1H-isoindol-2-yl]piperidine-2,6-dione。将“piperidine-2,6-dione”屏蔽为“*”后,得到的输入IUPAC名称为“3-[6-(1-benzylpiperidin-4-yl)-3-oxo-1H-isoindol-2-yl]*”,用于生成新分子(图3)。这种方法允许DiffIUPAC在保留结构的前提下,通过用不同的官能团替换被屏蔽的区域,生成新颖的分子。
模型生成的有效化合物显示,指定的子结构保持不变,而屏蔽区域被化学上可行的各种官能团替换。图3展示了一些代表性结构,展示了用于替换屏蔽区域的多样官能团。这种精确控制分子不同部分的能力使DiffIUPAC成为化学家优化先导化合物或探索新化学实体的有力工具,特别适用于进行目标性官能团修饰。
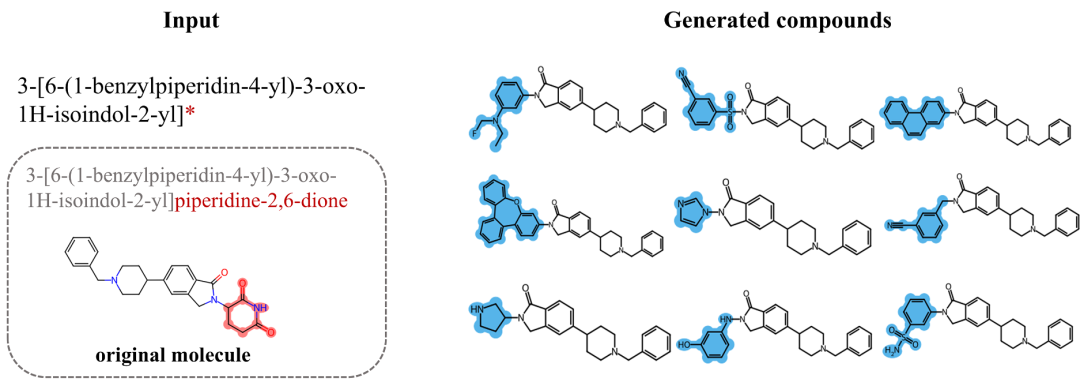
图3 模型生成的分子插图
类似物设计
在类似物设计中,通过生成从少量活性分子衍生的类药类似物库,以便进一步的虚拟筛选。我们利用DiffIUPAC模拟了药物类似物的设计过程。以BCL-xL抑制剂A-1331852为例,该药物通过阻断蛋白-蛋白相互作用(PPI)诱导肿瘤细胞凋亡。通过屏蔽其IUPAC名称的特定部分,生成了新的类似物。模型生成了3,000个有效分子,并筛选出与A-1331852具有相似结构的674个分子。随后,我们通过分子对接筛选潜在的BCL-xL候选抑制剂,结果显示549个类似物的对接评分高于A-1331852。最终,368个分子表现出更好的PPI靶向类药特性(QEPPI)和对接得分,表明DiffIUPAC生成的结构改进了A-1331852与BCL-xL的相互作用特性。经过可视化分析,选取了一个对接表现优异的分子进行进一步研究。雷达图分析表明,生成的命中化合物具有更好的PPI靶向类药特性,具备进一步实验评估的潜力。
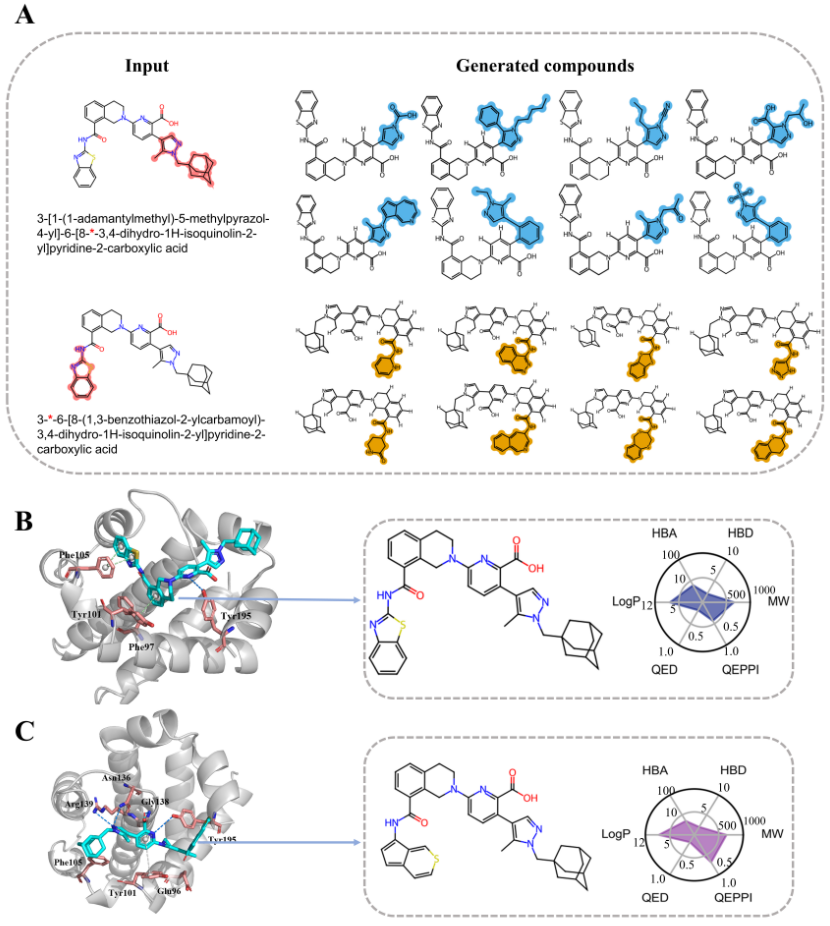
图4 (A)输入屏蔽的国际纯粹与应用化学联合会(IUPAC)名称A-1331852,用于生成类药物类似物(突出显示IUPAC名称中屏蔽的子结构,替换为“*”)。(B)Bcl-xL与A-1331852抑制剂的对接姿势和A-1331852物理化学性质的雷达图。(C)Bcl-xL与生成的模拟物的对接姿态,以及物理化学性质的雷达图。氢键显示为蓝色虚线。π-阳离子相互作用显示为灰色虚线。
Linker设计
片段药物发现(FBDD)常用于引领化合物的发现和优化,其中一种策略是通过连接片段优化分子的性质和亲和力。以破坏menin-MLL相互作用的抑制剂Ziftomenib为例,通过屏蔽其IUPAC名称中的连接子部分生成新分子。我们采样了1,000个有效分子,筛选出572个与Ziftomenib结构相似的分子。在对接筛选中,10个新分子的对接得分优于Ziftomenib。通过QEPPI评估类药性,7个化合物的QEPPI评分超过了Ziftomenib。进一步的对接分析显示,生成的命中化合物在menin结合位点表现出增强的相互作用,雷达图分析也显示出较好的类药性和PPI靶向性,表明新连接子在维持药物性质的同时增强了结合亲和力。这些结果表明,生成的新连接子在克服PPI药物发现中的挑战方面具有潜力。
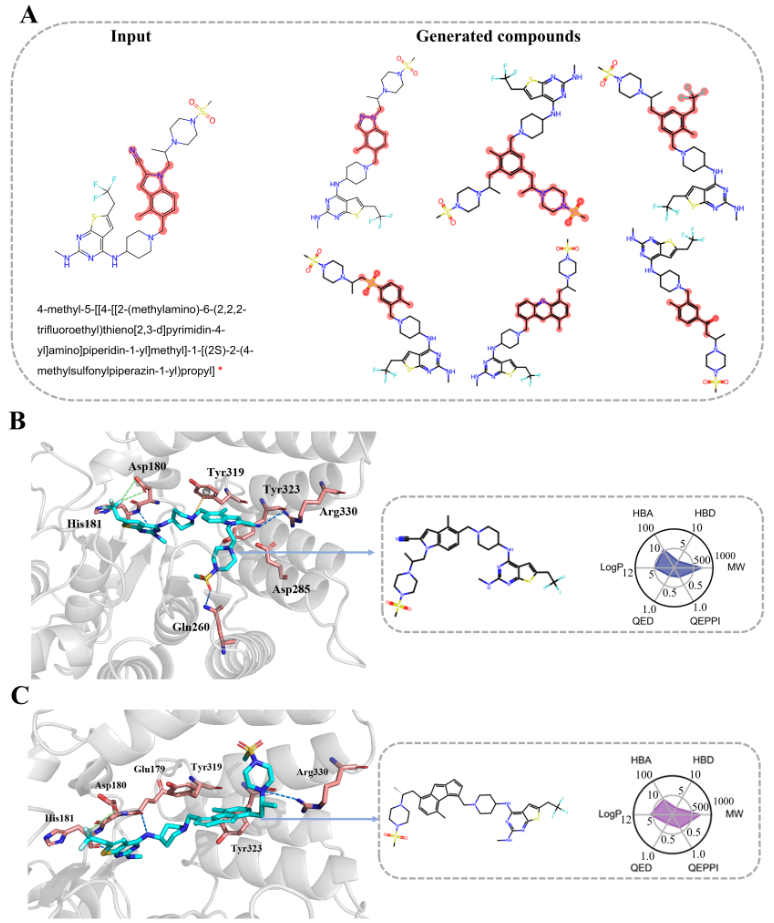
图5 (A)输入屏蔽的国际纯粹与应用化学联合会(IUPAC)名称Ziftomenib以生成接头(突出显示IUPAC名称中屏蔽的接头,替换为“*”)。(B)Menin与Ziftomenib的对接姿态,以及Ziftomeni物理化学性质的雷达图。(C)梅宁与生成化合物的对接姿态,以及物理化学性质的雷达图。氢键显示为蓝色虚线,卤素键显示为石灰虚线,π-阳离子相互作用显示为灰色虚线。
结论
本研究提出了一种从化学自然语言(IUPAC名称)到化学语言(SMILES)的受控分子编辑条件扩散模型。该模型可以根据屏蔽的IUPAC名称生成具有不同官能团约束的全新分子,为先导化合物设计的早期阶段提供候选结构。然而,先导化合物的设计和优化,尤其是生物相互作用的考虑,还需要超出DiffIUPAC能力范围的计算或实验步骤。
综合评估显示,DiffIUPAC模型成功捕捉了两种化学语言的规则及其映射关系,实现了受控的约束分子编辑。在分子生成中,DiffIUPAC在有效性、新颖性和多样性等指标上超越了基线方法。与基于SMILES的分子生成模型相比,IUPAC名称可以用于局部分子编辑,帮助领域专家进行分子设计和优化。在基于IUPAC名称的生成模型中,DiffIUPAC优于基于Transformer的模型,并首次探索了基于IUPAC名称的序列扩散生成建模。通过DiffIUPAC,我们展示了可以通过编辑IUPAC名称设计新的类似物和连接片段。我们结合条件扩散模型与Transformer神经网络来识别IUPAC名称和SMILES字符串规则之间的映射模式。结果表明,DiffIUPAC可以根据屏蔽的IUPAC名称生成具有子结构约束的新分子,并可用于先导化合物设计和优化过程,如构建虚拟库、设计类似物和创建连接子。
未来,将更多维的数据和方法(如强化学习)结合起来,有望满足多目标药物优化和设计需求。结合3D/4D分子结构数据将实现从IUPAC名称到3D结构的分子或材料设计。
参考资料
Jianmin Wang, Peng Zhou, Zixu Wang, Wei Long, Yangyang Chen, Kyoung Tai No, Dongsheng Ouyang, Jiashun Mao, Xiangxiang Zeng, Diffusion-based generative drug-like molecular editing with chemical natural language, Journal of Pharmaceutical Analysis,2024,101137.
https://doi.org/10.1016/j.jpha.2024.101137
--------- End ---------
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-12-30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读


