在R语言中的 ATACseq 数据分析全流程实战(一)
前面我们给大家介绍了非常好的 ATACseq数据分析 pipeline指导综述:综述:ATAC-Seq 数据分析工具大全。
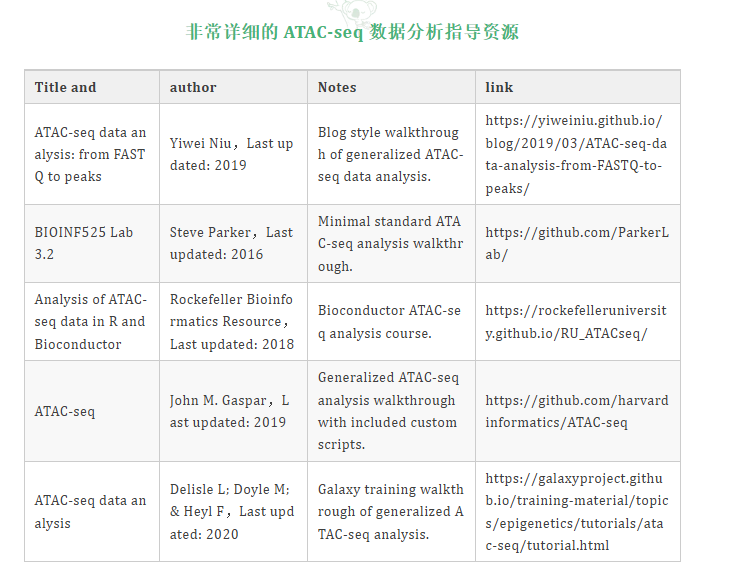
里面的全流程已经给大家介绍完了第一个:ATAC-seq data analysis: from FASTQ to peaks
今天来学习第二个:Analysis of ATAC-seq data in R and Bioconductor
https://rockefelleruniversity.github.io/RU_ATACseq/
这个教程主要介绍在R语言中的 ATACseq 数据分析。本课程包括两个部分。
1、常规的ATAC-seq分析工作流程的每一步:包括比对(alignment)、质量控制(QC)、peaks 识别(peak calling)、测试基因组中富集情况、基序富集(motif enrichment)以及差异peaks。
2、在所有子章节之后都包括了练习和答案表,以供练习技巧并提供未来参考示例。
0.环境配置
0.1.安装IGV
从这里下载进行安装:https://www.broadinstitute.org/igv/
0.2.安装MACS2
MACS2以及新版的MACS3信息可以在这里获取:https://macs3-project.github.io/MACS/
需要Linux or MacOS系统。这里教程中建议使用 Herper,一个R包以便于在R中安装和管理conda环境。我这里直接在服务器终端安装:
# 创建一个名字为chip的codna环境并激活
conda create -n chip -y python=3.10
conda activate chip
# 安装 MACS2
conda install bioconda::macs2 -y
# 看下是否安装成功
macs2 -h
0.3.安装R与Rstudio
R官网:http://www.r-project.org/,选择一个适合自己的电脑系统安装包。
Rsdudio网址:https://rstudio.com/products/rstudio/download/
0.4.安装需要的R包
## 使用西湖大学的 Bioconductor镜像
options(BioC_mirror="https://mirrors.westlake.edu.cn/bioconductor")
options("repos"=c(CRAN="https://mirrors.westlake.edu.cn/CRAN/"))
# 本课程的包
install.packages('BiocManager')
BiocManager::install('RockefellerUniversity/RU_ATACseq',subdir='atacseq')
# CRAN and Bioconductor
install.packages('BiocManager')
BiocManager::install('methods')
BiocManager::install('ggplot2')
BiocManager::install('rmarkdown')
BiocManager::install('ShortRead')
BiocManager::install('ashr')
BiocManager::install('ChIPQC')
BiocManager::install('DiffBind')
BiocManager::install('BSgenome.Hsapiens.UCSC.hg19')
BiocManager::install('Rsubread')
BiocManager::install('Rbowtie2')
BiocManager::install('R.utils')
BiocManager::install('Rsamtools')
BiocManager::install('BSgenome.Hsapiens.UCSC.hg38')
BiocManager::install('rtracklayer')
BiocManager::install('ChIPseeker')
BiocManager::install('soGGi')
BiocManager::install('GenomicAlignments')
BiocManager::install('TxDb.Hsapiens.UCSC.hg19.knownGene')
BiocManager::install('DESeq2')
BiocManager::install('BSgenome.Mmusculus.UCSC.mm10')
BiocManager::install('TxDb.Hsapiens.UCSC.hg38.knownGene')
BiocManager::install('tracktables')
BiocManager::install('clusterProfiler')
BiocManager::install('TxDb.Mmusculus.UCSC.mm10.knownGene')
BiocManager::install('devtools')
BiocManager::install('tidyr')
BiocManager::install('DT')
BiocManager::install('dplyr')
BiocManager::install('rGREAT')
BiocManager::install('MotifDb')
BiocManager::install('Biostrings')
BiocManager::install('GenomicRanges')
BiocManager::install('pheatmap')
BiocManager::install('universalmotif')
BiocManager::install('seqLogo')
BiocManager::install('org.Mm.eg.db')
BiocManager::install('ATACseqQC')
BiocManager::install('JASPAR2020')
BiocManager::install('motifmatchr')
BiocManager::install('chromVAR')
BiocManager::install('ggseqlogo')
BiocManager::install('TFBSTools')
BiocManager::install('motifStack')
BiocManager::install('knitr')
BiocManager::install('testthat')
BiocManager::install('yaml')
# 查看是否都能加载
library('methods')
library('ggplot2')
library('rmarkdown')
library('ShortRead')
library('ashr')
library('ChIPQC')
library('DiffBind')
library('BSgenome.Hsapiens.UCSC.hg19')
library('Rsubread')
library('Rbowtie2')
library('R.utils')
library('Rsamtools')
library('BSgenome.Hsapiens.UCSC.hg38')
library('rtracklayer')
library('ChIPseeker')
library('soGGi')
library('GenomicAlignments')
library('TxDb.Hsapiens.UCSC.hg19.knownGene')
library('DESeq2')
library('BSgenome.Mmusculus.UCSC.mm10')
library('TxDb.Hsapiens.UCSC.hg38.knownGene')
library('tracktables')
library('clusterProfiler')
library('TxDb.Mmusculus.UCSC.mm10.knownGene')
library('devtools')
library('tidyr')
library('DT')
library('dplyr')
library('rGREAT')
library('MotifDb')
library('Biostrings')
library('GenomicRanges')
library('pheatmap')
library('universalmotif')
library('seqLogo')
library('org.Mm.eg.db')
library('ATACseqQC')
library('JASPAR2020')
library('motifmatchr')
library('chromVAR')
library('ggseqlogo')
library('TFBSTools')
library('motifStack')
library('knitr')
library('testthat')
library('yaml')
0.5.下载好上课的材料
下载地址:https://github.com/RockefellerUniversity/RU_ATACseq/archive/master.zip
这个你下载有困难,可以直接找我发给你,微信:Biotree123
PPT资料:https://rockefelleruniversity.github.io/RU_ATACseq/presentations/slides/RU_ATAC.html#1
代码:https://rockefelleruniversity.github.io/RU_ATACseq/presentations/r_code/RU_ATAC.R
1.背景介绍
ATAC-seq使用转座酶在测序之前高效地切割可接近的DNA,提供了一种在全基因组范围内绘制可接近/开放染色质的方法。
与其他技术相比,ATAC-seq的优点有:
- 少量的组织样本即可(>10000个细胞)
- 实验周期短(~4hrs)
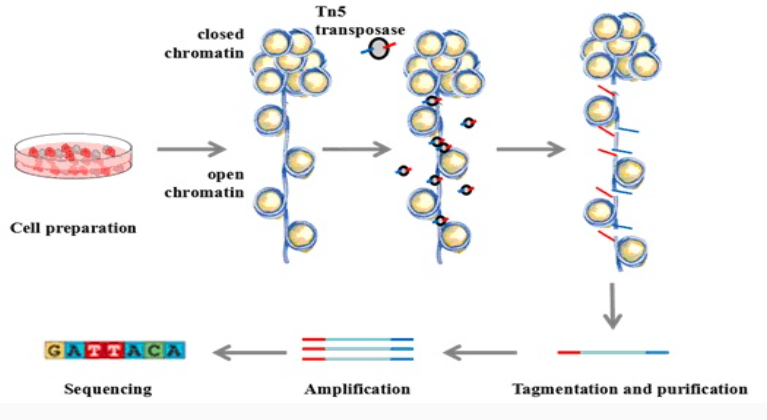
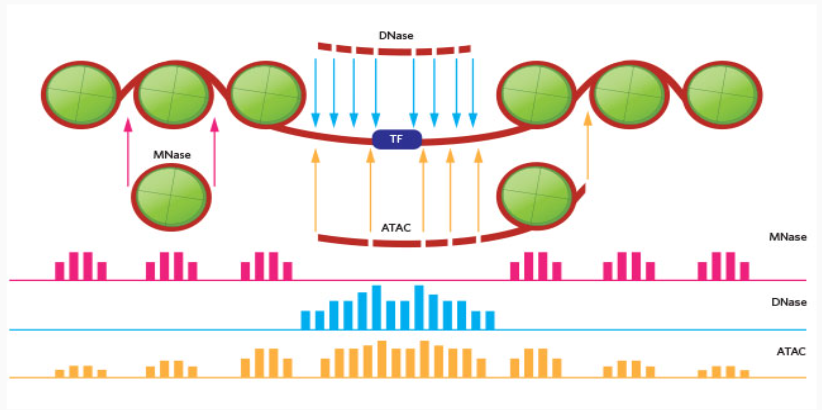
- DNaseseq:酶消化法从转录因子结合位点周围的开放染色质中提取信号
- MNaseseq:酶消化法提取代表核小体定位的信号
- ATACseq:使用转座酶,并提供了一种从单个样品的转录因子结合位点和核小体位置同时提取信号的方法
2.使用数据说明
本教程将介绍三个已发表的公共数据作为练习。
数据一:
来自文献:Transposition of native chromatin for multimodal regulatory analysis and personal epigenomics Jason D. Buenrostro, Paul G. Giresi, Lisa C. Zaba, Howard Y. Chang, and William J. Greenleaf.
数据编号为:GSE47753 https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE47753
使用其中一个样本:ATACseq_50k_Rep2 sample GEO - GSM1155958
fq下载:https://www.ebi.ac.uk/ena/browser/view/PRJNA207663
conda activate chip
conda install -y hcc::aspera-cli=3.9.6
# 下载
key=/nas2/zhangj/biosoft/miniconda3/envs/rna/etc/asperaweb_id_dsa.openssh
ascp -v -QT -l 300m -P33001 -k1 -i $key era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR891/SRR891269/SRR891269_1.fastq.gz ./
ascp -v -QT -l 300m -P33001 -k1 -i $key era-fasp@fasp.sra.ebi.ac.uk:/vol1/fastq/SRR891/SRR891269/SRR891269_2.fastq.gz ./
数据二:
数据来自项目ENCODE consortium的一部分,为小鼠的组织:ENCODE Project Consortium. An integrated encyclopedia of DNA elements in the human genome. Nature 2012 Sep 6;489(7414):57-74. PMID: 22955616
- Liver day 12:https://www.encodeproject.org/experiments/ENCSR302LIV/
- Kidney day 15:https://www.encodeproject.org/experiments/ENCSR023QZX/
- Hindbrain day 12:https://www.encodeproject.org/experiments/ENCSR088UYE/
下载:
## Liver day 12:https://www.encodeproject.org/experiments/ENCSR302LIV/
# 样本1
r1:https://www.encodeproject.org/files/ENCFF409BPW/@@download/ENCFF409BPW.fastq.gz
r2:https://www.encodeproject.org/files/ENCFF016UWL/@@download/ENCFF016UWL.fastq.gz
# 样本2
r1:https://www.encodeproject.org/files/ENCFF772GVP/@@download/ENCFF772GVP.fastq.gz
r2:https://www.encodeproject.org/files/ENCFF987EVS/@@download/ENCFF987EVS.fastq.gz
## Kidney day 15:https://www.encodeproject.org/experiments/ENCSR023QZX/
## Hindbrain day 12:https://www.encodeproject.org/experiments/ENCSR088UYE/
数据三:
来运:Christina Leslie's lab at MSKCC,处理的bam:T-Reg - ENCSR724UJS:https://www.encodeproject.org/experiments/ENCSR724UJS/
fq:
read1:https://www.encodeproject.org/files/ENCFF175VOD/@@download/ENCFF175VOD.fastq.gz
read2:https://www.encodeproject.org/files/ENCFF447BGX/@@download/ENCFF447BGX.fastq.gz
bam:https://www.encodeproject.org/files/ENCFF053CGD/@@download/ENCFF053CGD.bam
处理后的数据
上面的数据为原始fq格式,预处理需要耗费一定的时间,作者这里还提供了中间的文件:
bam和index文件:
https://s3.amazonaws.com/rubioinformatics/ATAC_Workshop/ATAC_Data/ATAC_BAM/Sorted_ATAC_50K_2.bam
https://s3.amazonaws.com/rubioinformatics/ATAC_Workshop/ATAC_Data/ATAC_BAM/Sorted_ATAC_50K_2.bam.bai
Small BAM, peak calls and directory structure:
https://s3.amazonaws.com/rubioinformatics/ATAC_Workshop_Essential.zip
Additional workshop files and directory structure:
https://s3.amazonaws.com/rubioinformatics/ATAC_Workshop.zip
Bigwigs for IGV:
https://s3.amazonaws.com/rubioinformatics/ATAC_bigWigs.zip
3.参考序列数据
ATACseq数据分析需要 reference data,包括:
- fa格式的参考基因组序列,作者使用 来自 BSGenome Bioconductor annotation R包,额到时候可能网速是个很大的问题啊!
- Gene模型:也是 R包 TxDb Bioconductor annotation packages
- Blacklists:参考基因组中的Artifact regions,在这里下载:https://www.encodeproject.org/annotations/ENCSR636HFF/
# human GRCh38
https://www.encodeproject.org/files/ENCFF356LFX/@@download/ENCFF356LFX.bed.gz
# 小鼠 mm10
https://www.encodeproject.org/files/ENCFF547MET/@@download/ENCFF547MET.bed.gz
4.ATACseq数据比对
在比对之前,作者建议我们做一下fastqc,看看数据质量:一些基本的质量控制检查可以帮助我们发现测序过程中是否存在任何偏差,例如reads质量的意外下降,或者非随机的GC含量。方法可参考:https://rockefelleruniversity.github.io/RU_ChIPseq/presentations/slides/ChIPseq_In_Bioconductor.html#6
4.1 创建参考基因组
我们这里分析的数据为human,使用hg19,R代码创建方式如下:
## 4.1 创建参考基因组
library(BSgenome.Hsapiens.UCSC.hg19)
mainChromosomes <- paste0("chr",c(1:21,"X","Y","M"))
mainChrSeq <- lapply(mainChromosomes,
function(x)BSgenome.Hsapiens.UCSC.hg19[[x]])
names(mainChrSeq) <- mainChromosomes
mainChrSeqSet <- DNAStringSet(mainChrSeq)
writeXStringSet(mainChrSeqSet, "BSgenome.Hsapiens.UCSC.hg19.mainChrs.fa")
这一步运行花个10来分钟,会在当前工作目录中生成一个fa文件:BSgenome.Hsapiens.UCSC.hg19.mainChrs.fa
4.2 创建 Rsubread 索引
## 4.2 创建 Rsubread 索引
library(Rsubread)
buildindex("BSgenome.Hsapiens.UCSC.hg19.mainChrs",
"BSgenome.Hsapiens.UCSC.hg19.mainChrs.fa",
indexSplit = TRUE,
memory = 1000)
总共运行了:Total running time: 48.9 minutes. 后面可以增加 memory 参数来提高速度,这里的单位还是M,1000M连一个G都没有干到~
生成的index文件包括fa在内总的文件大小共有18G,需要注意自己的磁盘空间大小~
4.3 比对 subread
ATACseq数据一般都为双端测序数据,文件名中一般具有_1和_2,_R1和_R2 关键词。
如我们前面下载的数据一里面的那个样本:
read1 <- "GSE47753/SRR891269_1.fastq.gz"
read2 <- "GSE47753/SRR891269_2.fastq.gz"
读取fq:
library(ShortRead)
read1 <- readFastq("GSE47753/SRR891269_1.fastq.gz")
read2 <- readFastq("GSE47753/SRR891269_2.fastq.gz")
id(read1)[1:2]
## BStringSet object of length 2:
## width seq
## [1] 59 HISEQ:236:HA03EADXX:1:1101:1147:2237 1:Y:0:TAAGGCGACTCTCTAT
## [2] 59 HISEQ:236:HA03EADXX:1:1101:1201:2248 1:N:0:TAAGGCGACTCTCTAT
id(read2)[1:2]
## BStringSet object of length 2:
## width seq
## [1] 59 HISEQ:236:HA03EADXX:1:1101:1147:2237 2:Y:0:TAAGGCGACTCTCTAT
## [2] 59 HISEQ:236:HA03EADXX:1:1101:1201:2248 2:N:0:TAAGGCGACTCTCTAT
# 其他的探索
sread(read1)[1:2]
sread(read2)[1:2]
quality(read1)[1:2]
quality(read2)[1:2]
read1 和 read2 之间的 距离非常重要,它可以使我们能够区分来自短片段还是长片段的 reads,分别表明了无核小体和有核小体的信号部分。
insert size:为 read1 与 read2 的起始位置之间的距离
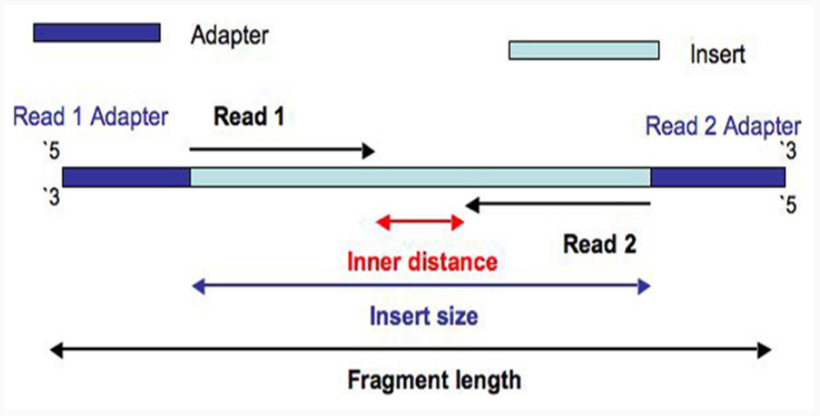
比对参数:maxFragLength
需要增加允许的最大片段长度,以捕获代表多核小体信号的长片段。这里设定的最大允许片段长度是基于 Greenleaf 研究中使用的参数,就是这个数据对应的那个文献。
作者还设置了unique参数为TRUE,以便只包含唯一比对的reads。
此外 type=1:dna (or 1; genomic DNA-seq data such as WGS, WES, ChIP-seq data etc.).
# 比对
read1 <- "GSE47753/SRR891269_1.fastq.gz"
read2 <- "GSE47753/SRR891269_2.fastq.gz"
align("subread_index/BSgenome.Hsapiens.UCSC.hg19.mainChrs",
readfile1=read1,
readfile2=read2,
output_file = "ATAC_50K_2.bam",
nthreads=64,
type=1,
unique=TRUE,
maxFragLength = 2000)
nthreads:最大只能设置 64! 有点不能忍受为什么要在R里面做上游!!!
让它且跑着吧, 明天继续(未完待续~)
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-03-13,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

