基于蛋白质谱数据的WGCNA分析以及模块网络可视化
前面我们给大家分享了一个将文章分析的数据和代码整理为在线书籍形式的文献,见:分享一篇将代码整理成在线book形式的顶刊文献(Q1/IF: 58.7),今天就来学习其中的Figure2,这里我修改优化了作者提供的一些代码,并增加了一些缺少的分析代码。
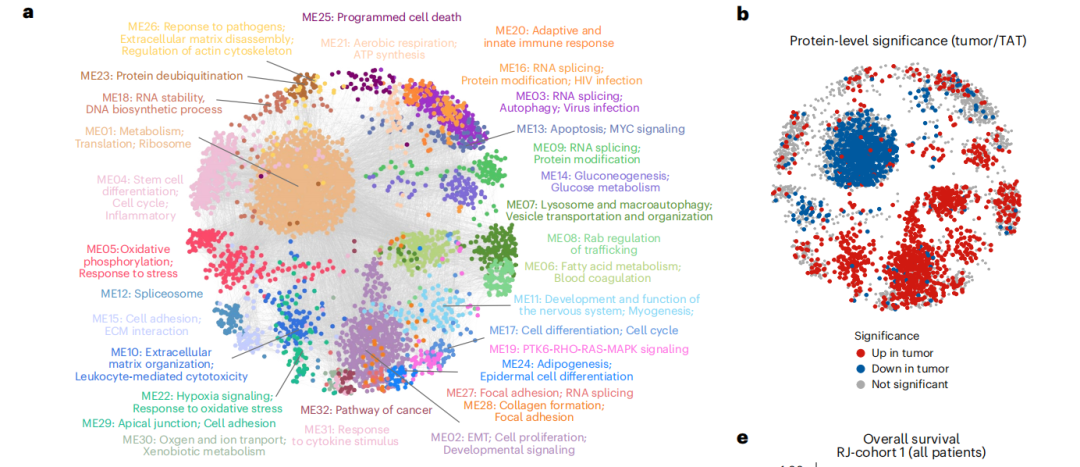
图二:PDAC的蛋白质组学图谱及功能蛋白模块的临床相关性
a,WGCNA识别出32个在肿瘤样本的蛋白质组学数据中富集的功能蛋白模块(ME01–32)。
b,将肿瘤与TATs之间显著上调/下调的蛋白质叠加到网络节点上。
「代码和数据」
文献中所用的数据可以在下面两个地方下载得到:
https://www.biosino.org/node/project/detail/OEP004100
https://doi.org/10.6084/m9.figshare.24863049
代码分享:http://www.genetictargets.com/PDAC2BOOKLET/index.html
WGCNA分析
这里对肿瘤样本的蛋白质组学数据进行 WGCNA 分析,共鉴定出32个功能模块。
Step 1: 加载蛋白表达矩阵和设置参数
读取蛋白表达矩阵 20230412_PDAC_PRO_exp.rds 以及 样本表型信息 20230412_PDAC_PRO_meta.rds
rm(lisst=ls())
library(WGCNA)
dir.create("./results/Figure2/WGCNA",recursive = T)
# Step 1: 加载蛋白的表达矩阵和设置参数
# 肿瘤样本
protein.nona.tumor <- readRDS("./PDAC2_data_results/data/proteomics/wgcna/20230412_PDAC_PRO_Tumor_exp.rds")
dim(protein.nona.tumor)
protein.nona.tumor[1:5,1:5]
# 相关性分析方法
corType = "pearson"
corFnc = ifelse(corType=="spearman", cor, bicor)
maxPOutliers = ifelse(corType=="pearson",1, 0.05)
# 输入数据标准化
plot.mat <- t(protein.nona.tumor - rowMeans(protein.nona.tumor)) # row: 样本 | col: 基因
plot.mat[1:5,1:5]

Step 2: 异常样本检测
首先对样本进行层次聚类分析:
# Step 2: identification of outlier samples
# 样本聚类
sampleTree = hclust(dist(plot.mat), method = "ward.D")
pdf(paste0("./results/Figure2/WGCNA/1.tree.pdf"), width = 20, height = 9)
plot(sampleTree, main = "Sample clustering to detect outliers", sub="", xlab="")
dev.off()
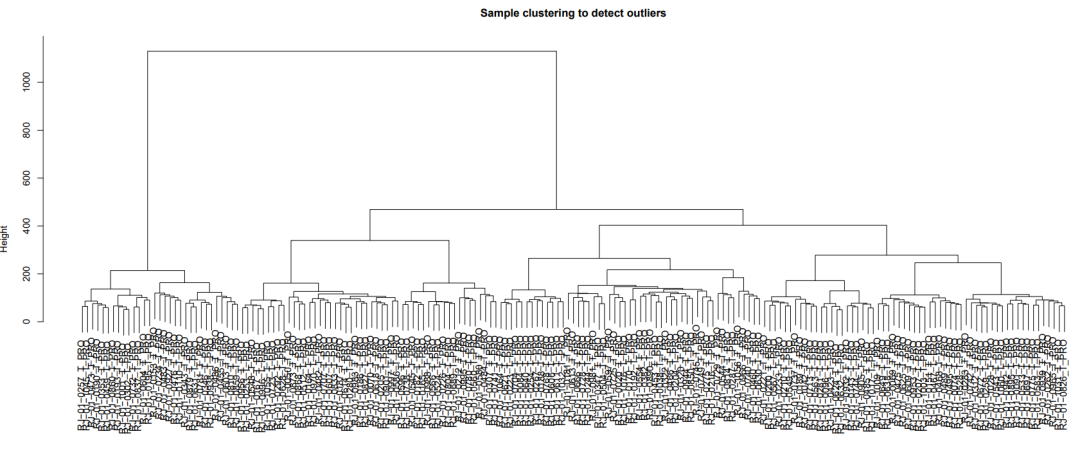
看起来没有异常离群样本,这个聚类结果想当nice哇!
Step 3-4: 软阈值选择
这里设置一系列软阈值,并计算每个软阈值的指标,「选择第一个达到0.85的那个power」。
# Step 3: analysis of network topology
# 设置软阈值
powers = c(c(1:10), seq(from = 12, to = 30, by = 2))
powers
# 计算每个power对应的指标
sft = pickSoftThreshold(plot.mat, powerVector=powers, networkType="unsigned", verbose=5)
# Plot the results:
pdf(paste0("./results/Figure2/WGCNA/2.power.pdf"), width = 12, height = 8)
par(mfrow = c(1,2))
cex1 = 0.9
plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",
ylab="Scale Free Topology Model Fit, signed R^2",type="n",
main = paste("Scale independence"))
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,cex=cex1,col="red")
abline(h=0.85,col="red") # 阈值线为0.85
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Soft Threshold (power)",ylab="Mean Connectivity", type="n",
main = paste("Mean connectivity"))
text(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers,
cex=cex1, col="red")
dev.off()
# Step 4: soft threshold : power
power = sft$powerEstimate
power
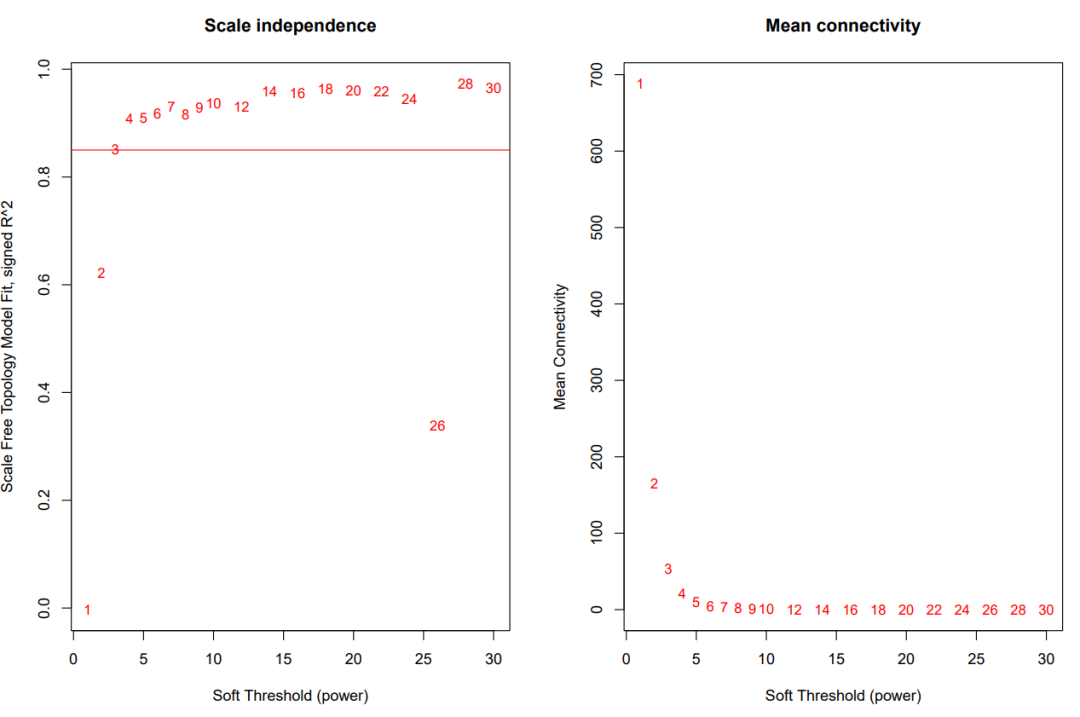
power为3。
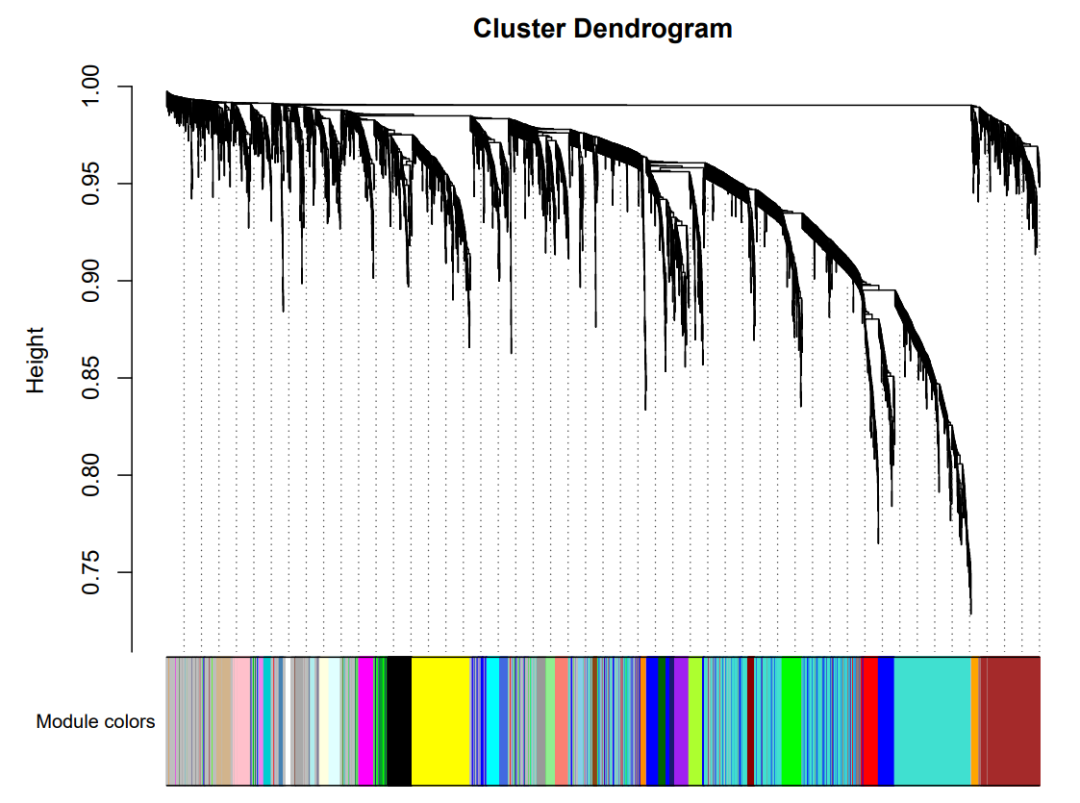
Step 5: 一步构建network和模块检测
这里共检测到32个module。
# Step 5: One-step network construction and module detection
net = blockwiseModules(plot.mat, power = power, maxBlockSize = ncol(plot.mat),
TOMType = "signed", minModuleSize = 20,
reassignThreshold = 0, mergeCutHeight = 0.0001,
numericLabels = TRUE, pamRespectsDendro = FALSE,
saveTOMs=TRUE, corType = corType,
maxPOutliers = 1, loadTOMs=TRUE,
randomSeed = 931, # seed
saveTOMFileBase = paste0("./results/Figure2/WGCNA/WGCNA.tom"),
verbose = 3, pearsonFallback = "none", deepSplit = 3 )
# module:
table(net$colors) # 0 corresponds to unclassified genes
# Convert [number] labels to [colors] for plotting
moduleLabels = net$colors
moduleColors = labels2colors(moduleLabels)
# plot
pdf(paste0( "./results/Figure2/WGCNA/3.module.pdf"), width = 8, height = 6)
plotDendroAndColors(net$dendrograms[[1]], moduleColors[net$blockGenes[[1]]],
"Module colors",
dendroLabels = FALSE, hang = 0.03,
addGuide = TRUE, guideHang = 0.05)
dev.off()
Step 6: 模块可视化
接下来可视化模块形成的网络,但是这里的网络数据不知道作者从哪里搞来的哇(wgcna/2D-node.data.rds 和 wgcna/2D-edge.data.rds),先画再说!
# Step 6: Vasualization
## data
node.data <- readRDS("./PDAC2_data_results/data/proteomics/wgcna/2D-node.data.rds")
head(node.data)
edge.data <- readRDS("./PDAC2_data_results/data/proteomics/wgcna/2D-edge.data.rds")
head(edge.data)
# 32个模块对应的颜色
color.module <- c("#CCCCCC","#ecb888","#af88bb","#a032cb","#efbed6","#fc496a","#b6d37f","#589336","#7fd68e","#52c465"
,"#3372e0","#84d7f6","#5394c3","#6376b3","#7f6cd7","#c4ceff","#fc9d40","#5c95e0","#cd7560","#ff70e4",
"#ff8738", "#ffcead","#1cbf8b", "#b76d38", "#1584ff", "#7f006d", "#ffd35f","#E66F73","#F57F20","#1DBB95"
,"#9CB79F","#F0B8D2","#A0485E","#A0688E","#C7E1DF","#51B1DF","#6D97D7","#5D6193","#CEC3E0","#A9917E"
,"#7C7D80","#F4E192","#ADD666")
names(color.module) <- paste0("ME", seq(0,length(color.module)-1) %>%
str_pad(width = 2,side = "left",pad = "0"))
color.module
## plot network
gg <- ggplot() +
geom_segment(mapping = aes(x = from.x, y = from.y, xend = to.x, yend = to.y),
color = "#CCCCCC", size = 0.01, data = edge.data) + # draw a straight line
geom_point(mapping = aes(x = pos.x, y = pos.y, color = Module),
size = 2, data = node.data) + # add point
scale_size(range = c(0, 6) * 2) + # specifies the minimum and maximum size
theme_void() +
labs(x = "", y = "", title = paste0("PPI")) +
scale_colour_manual(values = color.module)
gg
ggsave(paste0("./results/Figure2/2A-PRO-modules.png"), gg, width = 10, height = 8,bg = "white")
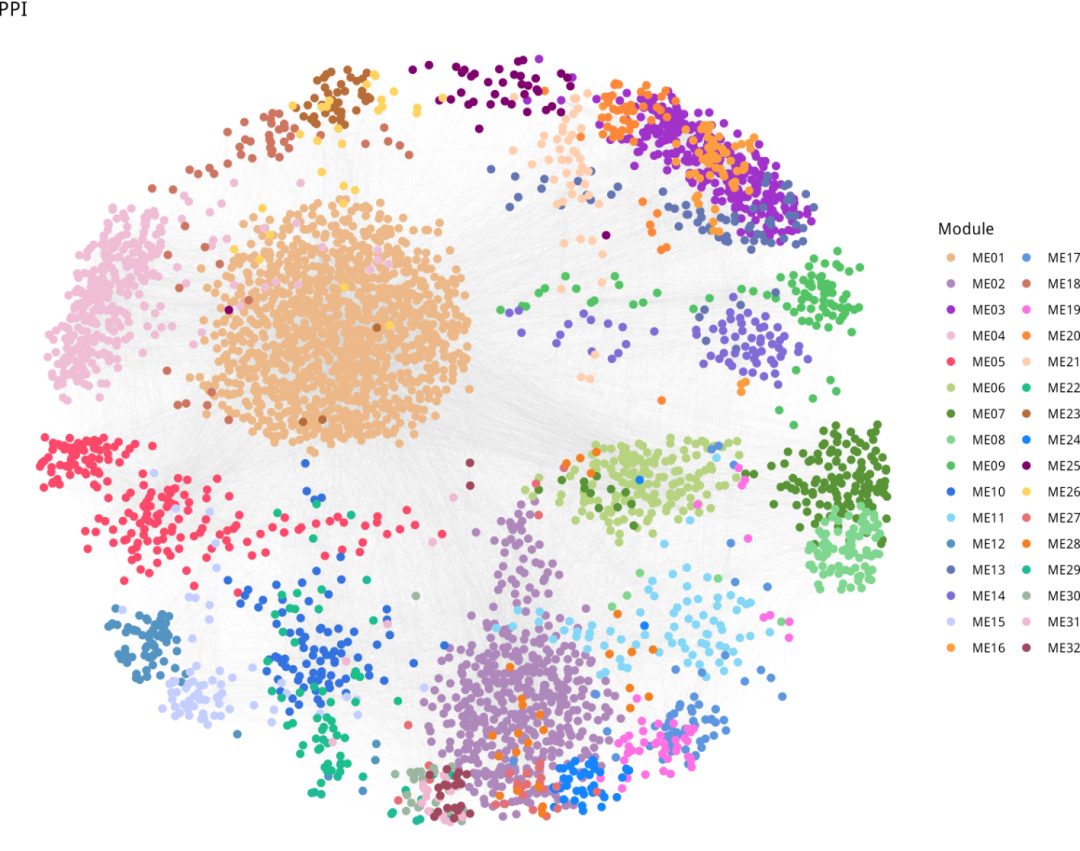
step7:模块功能富集分析
如果需要跟文献中一样,需要知道每个module对应的功能,「还需要对每个模块进行功能富集分析,这里是补充的代码」,作者并没有提供这些代码。
## 每个模块对应的基因以及功能富集分析
library(clusterProfiler)
library(org.Hs.eg.db)
library(ggplot2)
library(stringr)
save(net, file = "net.RData")
module <- data.frame(ID=names(net$colors), module=net$colors) # 0 corresponds to unclassified genes
head(module)
module <- module[module$module!="0", ]
table(module$module)
gcSample <- split(module$ID, module$module)
str(gcSample)
# 将基因symbol转为entrezid
gcSample = lapply(gcSample, function(y){
y=mapIds(org.Hs.eg.db, keys = y, keytype = 'SYMBOL', column = 'ENTREZID')
y=as.character(na.omit(y))
y
})
str(gcSample)
# 然后是GO数据库的BP,CC,MF的独立注释
# Run full GO enrichment test for BP
# GO BP
formula_res <- compareCluster(gcSample,fun="enrichGO", OrgDb="org.Hs.eg.db",
ont = "BP", pAdjustMethod = "BH", pvalueCutoff = 0.05, qvalueCutoff = 0.05 )
#KEGG
res_kegg <- compareCluster(gcSample, fun="enrichKEGG", organism="hsa", pvalueCutoff=0.05)
# 提取富集结果
res <- res_kegg@compareClusterResult
head(res)
## 通路筛选Top5
enrich <- res %>%
group_by(Cluster) %>%
top_n(n = 5, wt = -pvalue)
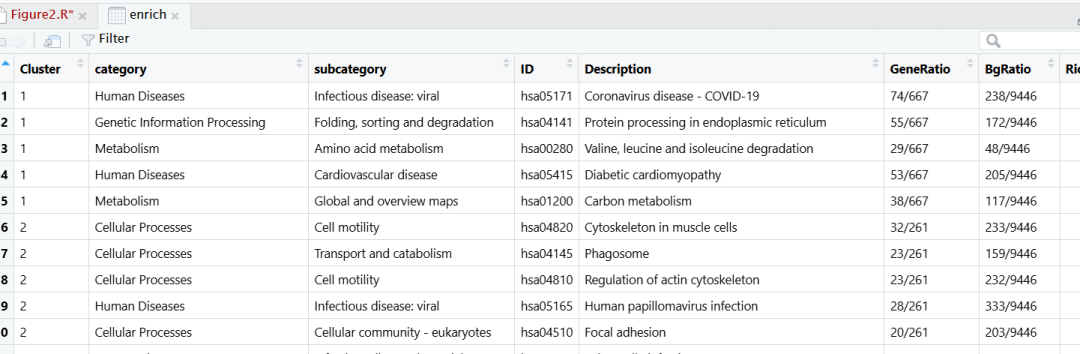
将得到的每个模块的功能按照显著性选取top1-3个通路后期使用AI工具标注在上图中!「这里的通路作者进行了精心挑选,但是我没有从文献中看到细节,靠各位自己根据生物学背景去挑了!」
今天分享到这~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-08,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

