多分组差异分析结果的两种展示形式
最近分析了一批RNA-seq的测序数据,发现DEseq2分析后有多了比较组。之前我们会绘制多个火山图或Upset图去呈现结果。但是,由于这两种方式被大家用太多了,所以我们想换几种另外的展示方式。我们在网上差了很多资料,其中有两个图个人感觉很不错,于是,就有了这一期的文案。下面我们直接进入今天的主题分享:
1. 利用TBtools的DEGs Dist Plot功能可视化多分组差异分析的结果
1.1 打开TBtools
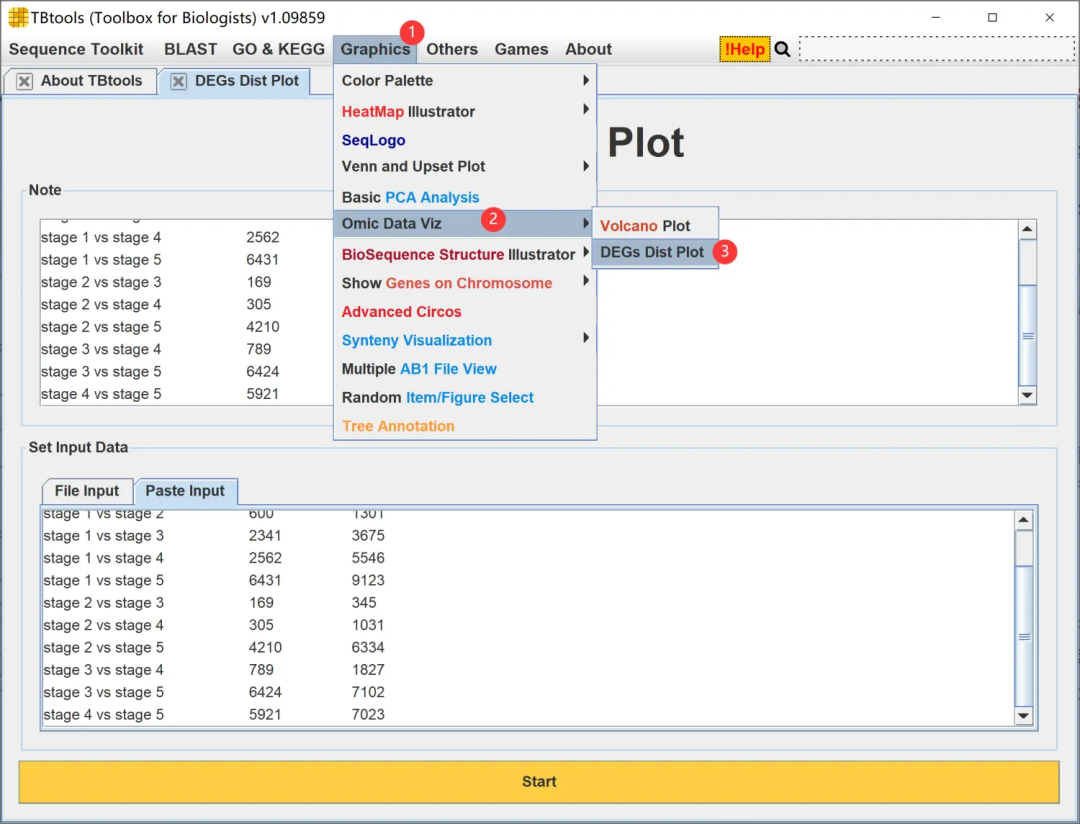
1.2 点击Graphics,选择Omic Data Viz → DEGs Dist Plot
1.3 界面打开即有示例数据,参照示例数据整理自己的结果
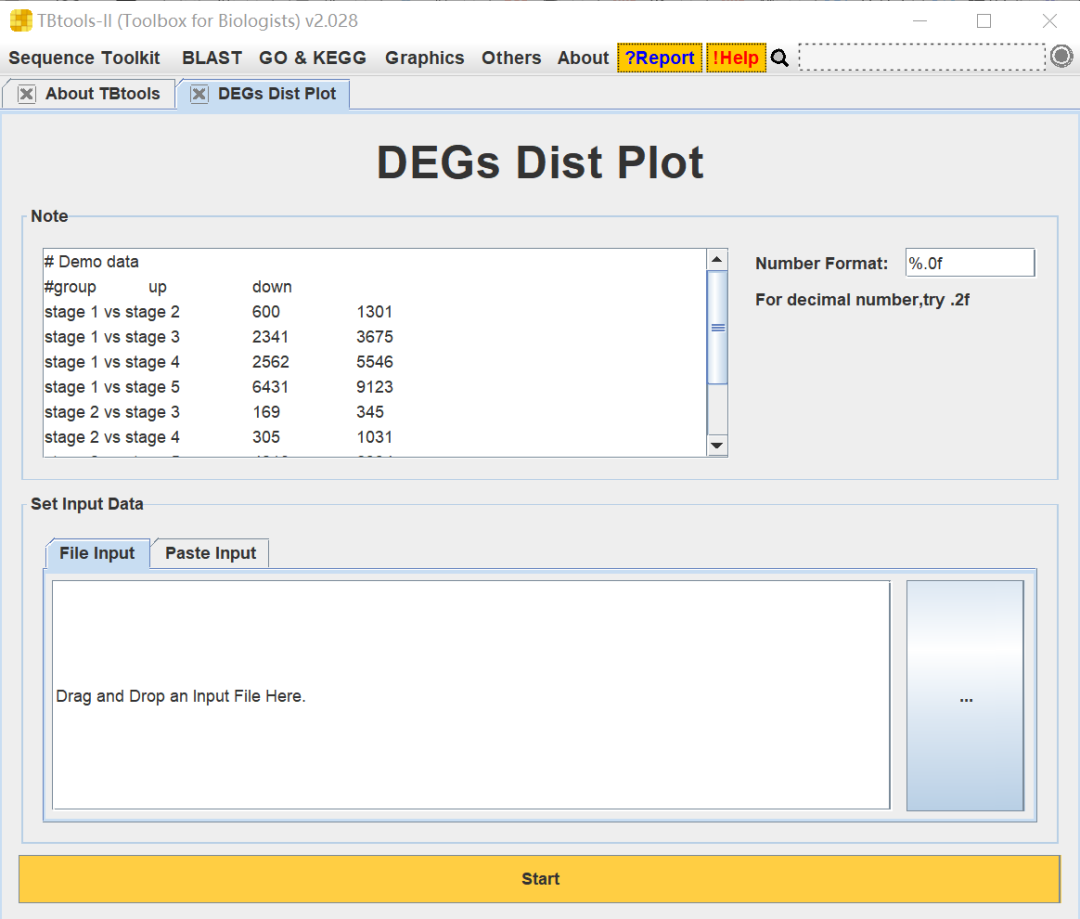
1.4 文件包括3列数据,以此为比较组合、上调基因数、下调基因数,以制表符分隔(Tab)

1.5 准备好文本文件,或者直接黏贴文本置入即可
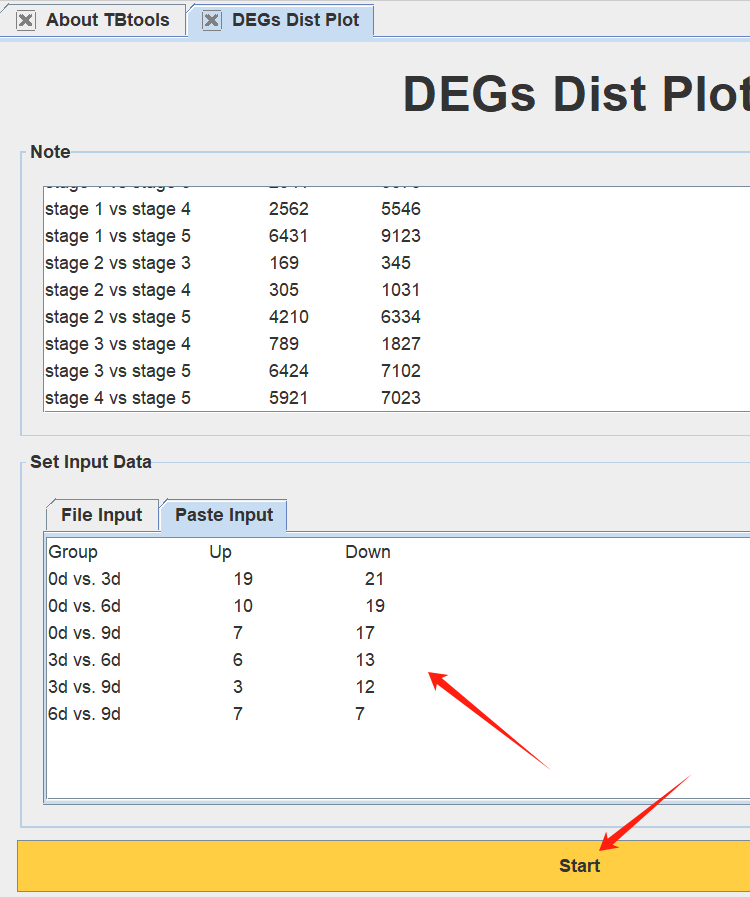
1.6 保存结果
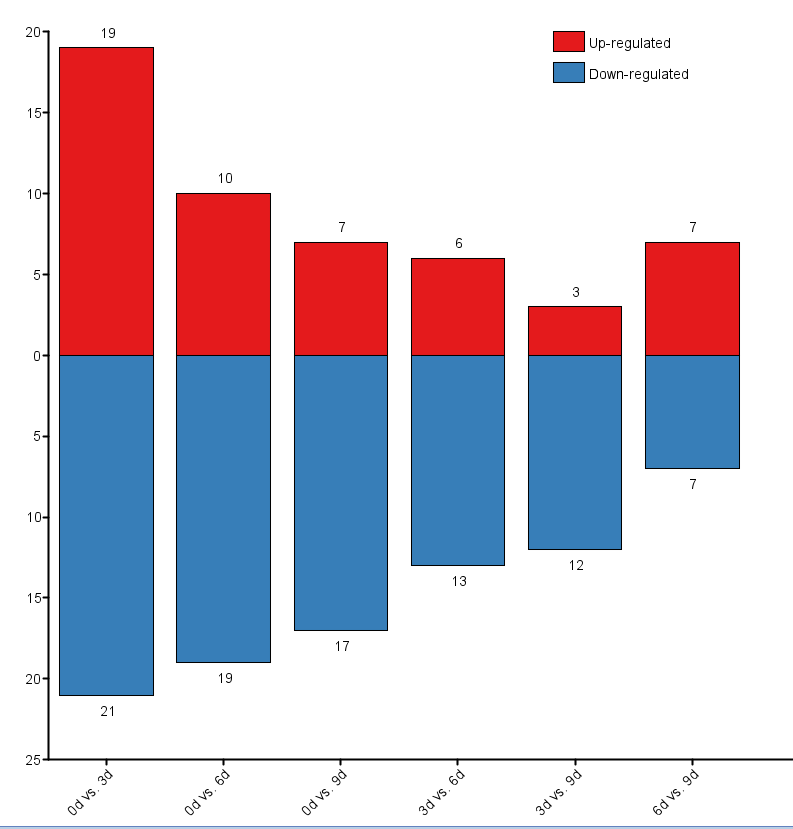
当然,上述图用R也可以实现,但我个人认为TBtools可视化结果更方便!
2. 借助单细胞差异分析的思路,将多个比较组的数据放到一张图上以散点图的形式展示
2.1 首先是将差异表达分析的结果整理成如下格式
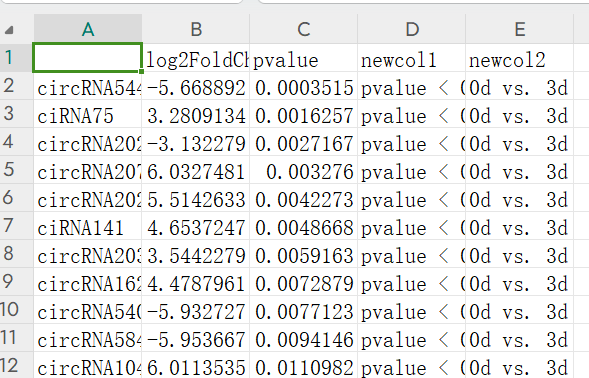
第一列:基因名;第二列:logfc;第三列:adjusted p value;第四列:给adjusted p value 一个分组;第五列:表示数据来自于哪组实验
2.2 代码运行
#读取数据
dat00<-read.csv("DEseq2-FC.csv")
head(dat00)
dat00=dat00[,-1] #因为行名的命名规则是不可有重复项,所以这里我们把第一列Name信息去了,使用序列号命名行名,不影响后期作图
#构造数据集用来添加中间部分的色块
dat<-data.frame(x=c("0d vs. 3d","0d vs. 6d","0d vs. 9d","3d vs. 6d","3d vs. 9d","6d vs. 9d"),
y=0,
label=c("0d vs. 3d","0d vs. 6d","0d vs. 9d","3d vs. 6d","3d vs. 9d","6d vs. 9d"))
#构造数据集用来添加背景的灰色柱子
datbar<-data.frame(x=c("0d vs. 3d","0d vs. 6d","0d vs. 9d","3d vs. 6d","3d vs. 9d","6d vs. 9d"),
y=c(20,22,24,10,10,10))
library(ggplot2)
library(ggnewscale)
library(tidyverse)
head(dat00)
p=ggplot()+
geom_col(data=datbar,aes(x=x,y=y),fill="grey",alpha=0.5)+
geom_col(data=datbar,aes(x=x,y=-y),fill="grey",alpha=0.5)+
geom_jitter(data=dat00 %>% filter(newcol1 == 'pvalue > 0.05'),
aes(x=newcol2,y=log2FoldChange,
color=newcol1))+
geom_jitter(data=dat00 %>% filter(newcol1 == 'pvalue < 0.05'),
aes(x=newcol2,y=log2FoldChange,
color=newcol1))+
scale_color_manual(name=NULL,
values = c("red","darkgrey"))+
ggnewscale::new_scale_fill()+
theme_minimal()+
theme(axis.line.y = element_line(),
axis.ticks.y = element_line(),
panel.grid = element_blank(),
legend.position = "top",
legend.justification = c(1,0),
legend.direction = "vertical",
axis.text.x = element_blank())+
labs(x=" ",y="average log2(FlodChang)")+
geom_tile(data=dat,
aes(x=x,y=y,fill=x),
height=3,color="black",
alpha=0.9,
show.legend = F)+
scale_fill_manual(values = c("#ECC947","#44a9a9","#4177aa","#12783c","#a94698","#E15659"))+
geom_text(data=dat,aes(x=x,y=y,label=label))
#输出图片
pdf(file="DEseq2-FC1.pdf", height=7, width=7)
print(p)
dev.off()
2.3 结果展示
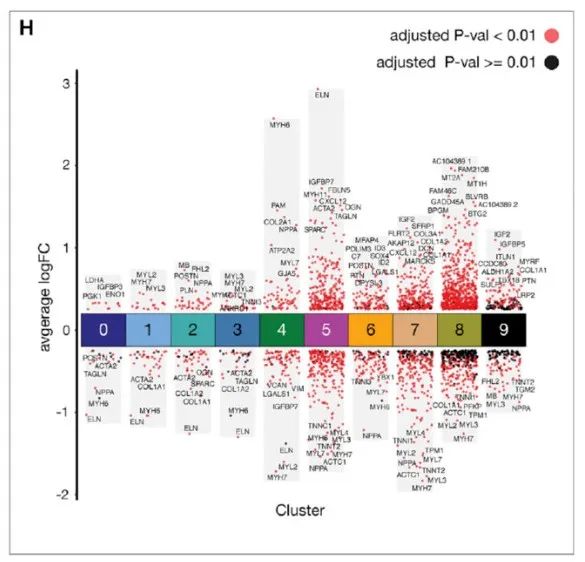
由于数据尚未发表,这里我们就不放结果的可视化效果图了,只要把表格信息整理好,稍微改一下代码就能做出效果图了。这里放一个来自A Spatiotemporal Organ-Wide Gene Expression and Cell Atlas of the Developing Human Heart文章的示例图供大家参考!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-05-20,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读