国内高速下载测序SRA数据
原创国内高速下载测序SRA数据
原创
欢迎大家关注全网生信学习者系列:
- WX公zhong号:生信学习者
- Xiao hong书:生信学习者
- 知hu:生信学习者
- CDSN:生信学习者2
介绍
在生物信息学研究中,公共测序数据资源的获取对于科研项目的进展至关重要。虽然NCBI的SRA(Sequence Read Archive)数据库提供了大量的测序数据,但由于网络访问速度的限制,特别是从国内访问时,下载速度可能受到严重影响。为了克服这一困难,欧洲生物信息学研究所(EBI)的ENA(European Nucleotide Archive)数据库及其提供的下载工具成为了一个可行的替代方案。EBI的ENA数据库与NCBI的SRA数据库类似,存储了大量的测序数据,并且提供了多种下载方式。其中,enaBrowserTools结合Aspera的方式因其高效和便捷性而受到推荐。这种下载方式不仅速度快,而且操作简单,只需提供数据的accession号(如SRR号)即可。
软件
- Aspera
High-performace transfer brower plugin,IBM旗下的高速下载插件。
# 下载
wget wget https://download.asperasoft.com/download/sw/connect/3.9.9/ibm-aspera-connect-3.9.9.177872-linux-g2.12-64.tar.gz
# 解压
tar -zxvf ibm-aspera-connect-3.9.9.177872-linux-g2.12-64.tar.gz
# 安装 软件安装路径是用户根目录的 .aspera/
./ibm-aspera-connect-3.9.9.177872-linux-g2.12-64.sh enaDataGet/enaGroupGet是enaBrowserTools的一个python脚本,enaBrowserTools是基于python3的与ENA web services接口的套件,可以方便访问ENA。安装方式简单,直接上github下载源代码,然后解压即可。
enaDataGet: all data for a given sequence, assembly, read or analysis accession or WGS set.
enaGroupGet: all data for a particular group(sequence, WGS, assembly, read or analysis) for a given sample or study accession.
# 下载&解压
wget https://github.com/enasequence/enaBrowserTools/archive/v1.6.tar.gz
tar -zxvf v1.6.tar.gz
# 配置别名 方便查看脚本位置信息
alias enaDataGet=/disk/share/toolkits/enaBrowserTools/python3-1.6/enaDataGet
alias enaGroupGet=/disk/share/toolkits/enaBrowserTools/python3-1.6/enaGroupGet配置
- 配置aspera:
# 配置许可(这一步需要账户有root权限,普通用户无法设置,也可以不需要设置)
sudo cp ~/.aspera/connect/etc/aspera-license /usr/local/bin/
# 添加环境变量
echo 'export PATH=~/.aspera/connect/bin:$PATH' >> ~/.bashrc
source ~/.bashrc
# 配置秘钥
mkdir /home/zouhua/.aspera/config/
# 复制到配置目录
cp ~/.aspera/connect/etc/asperaweb_id_dsa.openssh /home/zouhua/.aspera/config/- 配置enaDataGet
如果使用Aspera下载数据,则需要配置aspera_settings.ini文件
# step1 配置aspera_settings.ini
cd /disk/share/toolkits/enaBrowserTools-1.6 & vi aspera_settings.ini配置前
[aspera]
ASPERA_BIN = /path/to/ascp
ASPERA_PRIVATE_KEY = /path/to/aspera_dsa.openssh
ASPERA_OPTIONS =
ASPERA_SPEED = 100M配置后: 1. 指定ascp脚本;2.指定密钥;3.设置下载速度
[aspera]
ASPERA_BIN = /home/zouhua/.aspera/connect/bin/ascp
ASPERA_PRIVATE_KEY = /home/zouhua/.aspera/connect/etc/asperaweb_id_dsa.openssh
ASPERA_OPTIONS =
ASPERA_SPEED = 500M# step2 配置aspera
export ENA_ASPERA_INIFILE="/disk/share/toolkits/enaBrowserTools-1.6/aspera_settings.ini"查看帮助文档
- enaDataGet:重要参数 1. -f 指定数据类型;2. -d 指定本地下载目录;3. -a 指定是否使用aspera
usage: enaDataGet [-h] [-f {embl,fasta,submitted,fastq,sra}] [-d DEST] [-w]
[-m] [-i] [-a] [-as ASPERA_SETTINGS] [-v]
accession
Download data for a given accession
positional arguments:
accession Sequence, coding, assembly, run, experiment or
analysis accession or WGS prefix (LLLLVV) to download
optional arguments:
-h, --help show this help message and exit
-f {embl,fasta,submitted,fastq,sra}, --format {embl,fasta,submitted,fastq,sra}
File format required. Format requested must be
permitted for data type selected. sequence, assembly
and wgs accessions: embl(default) and fasta formats.
read group: submitted, fastq and sra formats. analysis
group: submitted only.
-d DEST, --dest DEST Destination directory (default is current running
directory)
-w, --wgs Download WGS set for each assembly if available
(default is false)
-e, --extract-wgs Extract WGS scaffolds for each assembly if available
(default is false)
-exp, --expanded Expand CON scaffolds when downloading embl format
(default is false)
-m, --meta Download read or analysis XML in addition to data
files (default is false)
-i, --index Download CRAM index files with submitted CRAM files,
if any (default is false). This flag is ignored for
fastq and sra format options.
-a, --aspera Use the aspera command line client to download,
instead of FTP.
-as ASPERA_SETTINGS, --aspera-settings ASPERA_SETTINGS
Use the provided settings file, will otherwise check
for environment variable or default settings file
location.
-v, --version show program's version number and exit
############################################################################
usage: enaGroupGet [-h] [-g {sequence,wgs,assembly,read,analysis}]
[-f {embl,fasta,submitted,fastq,sra}] [-d DEST] [-w] [-m]
[-i] [-a] [-as ASPERA_SETTINGS] [-t] [-v]
accession
Download data for a given study or sample, or (for sequence and assembly) taxon
positional arguments:
accession Study or sample accession or NCBI tax ID to fetch data
for
optional arguments:
-h, --help show this help message and exit
-g {sequence,wgs,assembly,read,analysis}, --group {sequence,wgs,assembly,read,analysis}
Data group to be downloaded for this
study/sample/taxon (default is read)
-f {embl,fasta,submitted,fastq,sra}, --format {embl,fasta,submitted,fastq,sra}
File format required. Format requested must be
permitted for data group selected. sequence, assembly
and wgs groups: embl and fasta formats. read group:
submitted, fastq and sra formats. analysis group:
submitted only.
-d DEST, --dest DEST Destination directory (default is current running
directory)
-w, --wgs Download WGS set for each assembly if available
(default is false)
-e, --extract-wgs Extract WGS scaffolds for each assembly if available
(default is false)
-exp, --expanded Expand CON scaffolds when downloading embl format
(default is false)
-m, --meta Download read or analysis XML in addition to data
files (default is false)
-i, --index Download CRAM index files with submitted CRAM files,
if any (default is false). This flag is ignored for
fastq and sra format options.
-a, --aspera Use the aspera command line client to download,
instead of FTP.
-as ASPERA_SETTINGS, --aspera-settings ASPERA_SETTINGS
Use the provided settings file, will otherwise check
for environment variable or default settings file
location.
-t, --subtree Include subordinate taxa (taxon subtree) when querying
with NCBI tax ID (default is false)
-v, --version show program's version number and exit使用
enaDataGet和enaGroupGet可配置aspera使用,参数为-a/--aspera,添加此参数则调用aspera。
- step1 先搜索accession在EBI的api接口;
- step2 本地创建下载日志文件目录 logs;
- step3 使用ascp软件下载accession;
/disk/share/toolkits/enaBrowserTools-1.6/python3/enaDataGet -f sra -a SRR212430 -d ./sra
# /disk/share/toolkits/enaBrowserTools-1.6/python3/enaDataGet -f sra SRR212430 -d ./sra下载成功
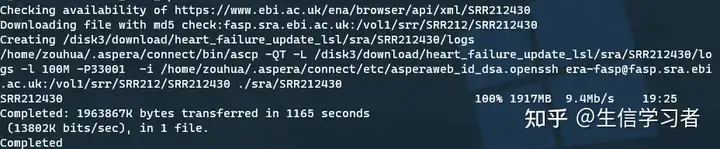
下载失败: 出现session stop即为失败
- 完全下载失败
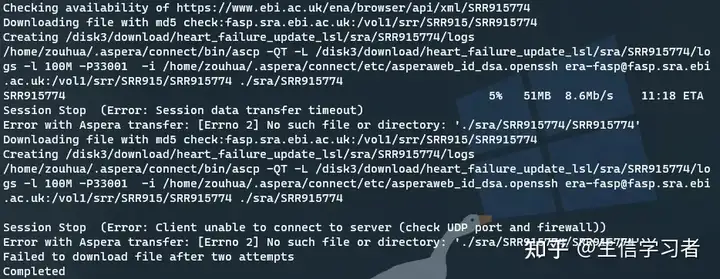
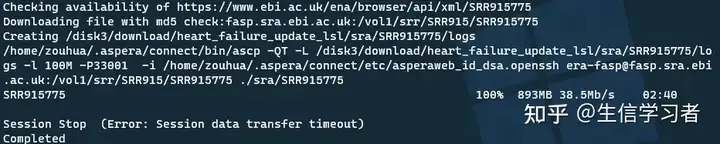
- 部分下载失败
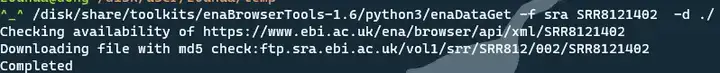
不使用aspera下载
问题
- 出现session stop信息,解决方案:
- ASPERA_SPEED 设置在100 - 400 M之间;
- root权限下开通 udp端口 iptables -I INPUT -p udp --dport 3301 -j ACCEPT iptables -I OUTPUT -p udp --dport 3301 -j ACCEPT
参考
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。