[数据分析]倾向得分匹配:在观察性研究中"创造"实验组
原创[数据分析]倾向得分匹配:在观察性研究中"创造"实验组
原创
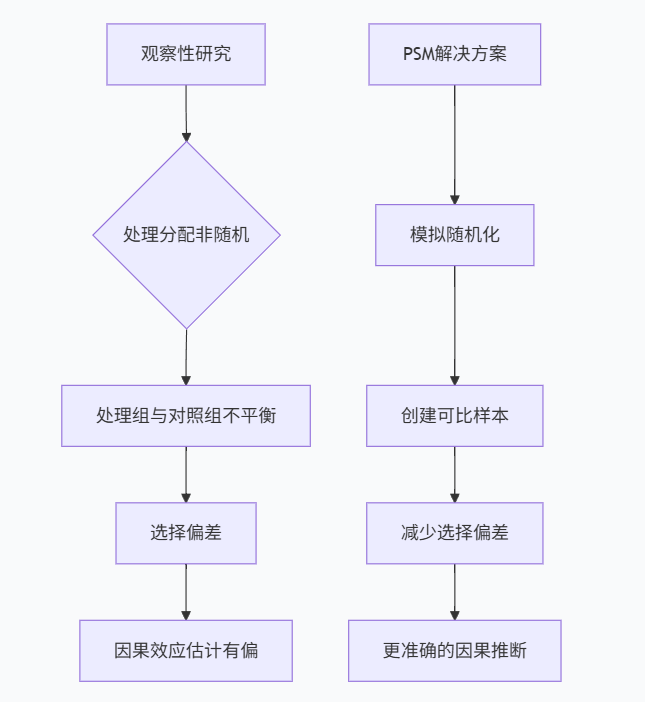
在观察性研究中,我们经常面临处理组和对照组基线特征不平衡的问题,这会导致选择偏差和混淆偏倚。倾向得分匹配(Propensity Score Matching, PSM)作为一种强大的因果推断方法,通过模拟随机实验的条件,帮助我们在非实验数据中估计处理效应。
I. 观察性研究中的挑战:为什么需要PSM?
在理想的世界中,我们总是可以通过随机对照试验(RCT)来评估处理效应,随机化能够确保处理组和对照组在所有观测和未观测特征上具有可比性。然而,在现实世界中,由于伦理、成本或可行性的限制,我们往往只能进行观察性研究。
观察性研究中的选择偏差问题
在观察性研究中,处理分配通常不是随机的,而是受到各种因素的影响。这导致处理组和对照组在基线特征上存在系统性差异,从而产生选择偏差。
问题类型 | 描述 | 示例 |
|---|---|---|
显性偏差 | 由观测到的协变量引起的偏差 | 年龄、性别、教育水平等 |
隐性偏差 | 由未观测到的协变量引起的偏差 | 动机、能力、遗传因素等 |
自选择偏差 | 个体自我选择进入处理组的倾向 | 患者选择更积极的治疗方案 |
传统方法的局限性
在PSM出现之前,研究者通常使用回归调整方法来控制混淆变量。然而,这种方法存在几个重要局限:
I. 模型依赖性强:结果对模型设定的敏感性高
II. 维度诅咒:当协变量较多时,难以找到足够匹配的个体
III. 外推问题:回归模型可能会在数据稀疏区域进行不可靠的外推
IV. 平衡性难以验证:无法直观检查协变量在处理组和对照组之间的平衡性
PSM的优势
倾向得分匹配通过将多维协变量信息压缩为一维的倾向得分,解决了上述问题:
- 降维处理:将多个协变量转换为一个得分,简化匹配过程
- 平衡性验证:可以直观检查匹配后样本的平衡性
- 减少模型依赖:对结果模型的依赖较小
- 透明性:匹配过程更加直观和透明
II. 倾向得分匹配的基本原理
倾向得分匹配的核心思想是通过模拟随机实验的条件,创建处理组和对照组的可比样本。本节将深入探讨PSM的理论基础和工作机制。
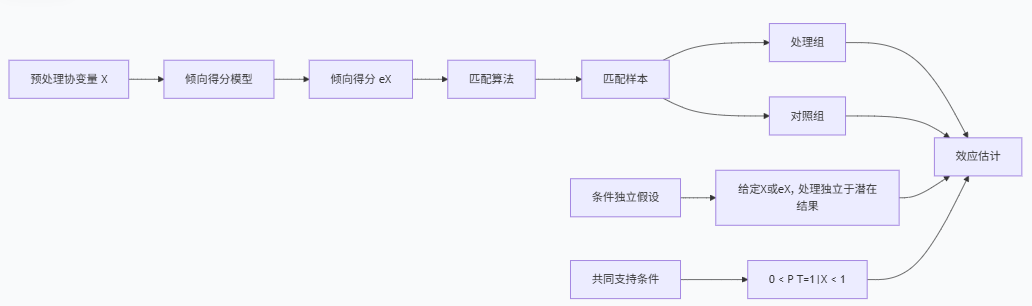
倾向得分的定义与性质
倾向得分由Rosenbaum和Rubin在1983年提出,定义为在给定一组协变量X的条件下,个体接受处理的条件概率:
e(X) = P(T=1|X)
其中,T是处理指示变量(1=处理组,0=对照组),X是预处理协变量向量。
倾向得分的关键性质:
- 平衡性性质:在给定倾向得分的条件下,处理组和对照组的协变量分布相似
T \perp X | e(X)
- 忽略性性质:如果条件独立假设成立,在给定倾向得分的条件下,处理分配与潜在结果独立
(Y(0), Y(1)) \perp T | e(X)
条件独立假设
PSM的有效性依赖于条件独立假设(CIA),也称为可忽略性假设。该假设要求,在控制了观测协变量后,处理分配与潜在结果独立。
数学表达:
(Y(0), Y(1)) \perp T | X
在实践中,我们使用倾向得分e(X)代替X,基于平衡性性质:
(Y(0), Y(1)) \perp T | e(X)
共同支持条件
另一个关键假设是共同支持条件,要求对于每个处理组个体,都存在具有相似倾向得分的对照组个体,反之亦然。
数学表达:
0 < P(T=1|X) < 1
这意味着没有个体肯定接受处理或肯定不接受处理,确保处理组和对照组有足够的重叠区域。
PSM的因果识别框架
在潜在结果框架下,个体i的处理效应定义为:
\tau_i = Y_i(1) - Y_i(0)
由于我们只能观察到其中一个潜在结果,PSM通过匹配来估计反事实结果:
对于处理组个体i,我们寻找对照组中具有相似倾向得分的个体j,然后用Y_j(0)作为Y_i(0)的估计。
平均处理效应(ATE)的估计为:
\hat{\tau}_{ATE} = \frac{1}{N} \sum_{i=1}^N [Y_i(1) - \hat{Y}_i(0)]
处理组平均处理效应(ATT)的估计为:
\hat{\tau}_{ATT} = \frac{1}{N_1} \sum_{i:T_i=1} [Y_i(1) - \hat{Y}_i(0)]
其中N是总样本数,N_1是处理组样本数,(\hat{Y}_i(0))是反事实结果的估计值。
III. PSM的实现步骤详解
成功的倾向得分匹配需要系统性地执行一系列步骤。本节将详细介绍PSM的完整流程,包括倾向得分估计、匹配方法选择、平衡性检验等关键环节。
PSM实施流程
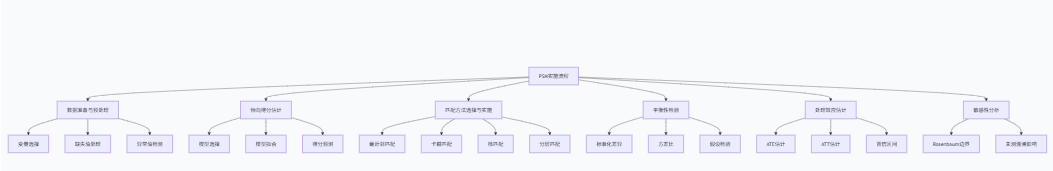
步骤1:数据准备与预处理
在开始PSM之前,需要进行充分的数据准备和预处理工作:
I. 变量选择:
- 包括所有与处理分配和结果相关的预处理变量
- 避免包括处理后的变量("坏控制"问题)
- 考虑包括工具变量以提高预测精度,但需谨慎
II. 缺失值处理:
- 使用多重插补等方法处理缺失数据
- 避免简单删除,可能导致选择偏差
III. 异常值检测:
- 识别和处理极端值,防止对倾向得分估计的过度影响
步骤2:倾向得分估计
倾向得分通常使用逻辑回归模型估计,但也可以使用其他机器学习方法:
import numpy as np
import pandas as pd
from sklearn.linear_model import LogisticRegression
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import StratifiedKFold
from sklearn.calibration import CalibratedClassifierCV
class PropensityScoreEstimator:
def __init__(self, method='logistic', **kwargs):
self.method = method
self.model = None
self.ps_scores = None
if method == 'logistic':
self.model = LogisticRegression(**kwargs)
elif method == 'random_forest':
self.model = RandomForestClassifier(**kwargs)
else:
raise ValueError("不支持的估计方法")
def estimate_ps(self, X, T, cv=False, n_splits=5):
"""估计倾向得分"""
if cv:
# 使用交叉验证提高估计稳健性
skf = StratifiedKFold(n_splits=n_splits)
ps_scores = np.zeros(len(T))
for train_idx, test_idx in skf.split(X, T):
X_train, X_test = X.iloc[train_idx], X.iloc[test_idx]
T_train = T.iloc[train_idx]
self.model.fit(X_train, T_train)
if hasattr(self.model, "predict_proba"):
ps_scores[test_idx] = self.model.predict_proba(X_test)[:, 1]
else:
# 对于没有predict_proba的模型,使用校准
calibrated_model = CalibratedClassifierCV(self.model, cv=3)
calibrated_model.fit(X_train, T_train)
ps_scores[test_idx] = calibrated_model.predict_proba(X_test)[:, 1]
self.ps_scores = ps_scores
else:
self.model.fit(X, T)
if hasattr(self.model, "predict_proba"):
self.ps_scores = self.model.predict_proba(X)[:, 1]
else:
calibrated_model = CalibratedClassifierCV(self.model, cv=5)
calibrated_model.fit(X, T)
self.ps_scores = calibrated_model.predict_proba(X)[:, 1]
return self.ps_scores
def check_overlap(self, T, bins=20):
"""检查共同支持区域"""
import matplotlib.pyplot as plt
ps_treated = self.ps_scores[T == 1]
ps_control = self.ps_scores[T == 0]
plt.figure(figsize=(10, 6))
plt.hist(ps_control, bins=bins, alpha=0.5, label='对照组', density=True)
plt.hist(ps_treated, bins=bins, alpha=0.5, label='处理组', density=True)
plt.xlabel('倾向得分')
plt.ylabel('密度')
plt.title('倾向得分分布 - 共同支持检查')
plt.legend()
plt.show()
# 计算共同支持区域
min_ps = max(np.min(ps_treated), np.min(ps_control))
max_ps = min(np.max(ps_treated), np.max(ps_control))
overlap_ratio = (len(ps_treated[(ps_treated >= min_ps) & (ps_treated <= max_ps)]) +
len(ps_control[(ps_control >= min_ps) & (ps_control <= max_ps)])) / len(T)
print(f"共同支持区域: [{min_ps:.3f}, {max_ps:.3f}]")
print(f"共同支持样本比例: {overlap_ratio:.3f}")
return min_ps, max_ps, overlap_ratio步骤3:匹配方法选择与实施
有多种匹配方法可用于PSM,每种方法各有优缺点:
匹配方法 | 描述 | 优点 | 缺点 |
|---|---|---|---|
最近邻匹配 | 为每个处理组个体寻找最相似的对照组个体 | 简单直观,易于实现 | 可能匹配质量不均 |
卡钳匹配 | 在最近邻匹配基础上添加得分差异限制 | 避免不良匹配,提高质量 | 可能丢失部分样本 |
核匹配 | 使用所有对照组个体,按相似度加权 | 减少方差,充分利用数据 | 计算复杂,需要选择带宽 |
分层匹配 | 按倾向得分分层,层内比较 | 简单,不需要个体匹配 | 层内可能仍有差异 |
精确匹配 | 要求协变量完全匹配 | 确保协变量平衡 | 可能找不到匹配,样本损失大 |
class PropensityScoreMatcher:
def __init__(self, method='nearest', **kwargs):
self.method = method
self.matched_data = None
self.matching_info = None
def nearest_neighbor_match(self, ps_scores, T, caliper=None, replace=False, ratio=1):
"""最近邻匹配"""
from sklearn.neighbors import NearestNeighbors
import numpy as np
treated_ps = ps_scores[T == 1].values.reshape(-1, 1)
control_ps = ps_scores[T == 0].values.reshape(-1, 1)
control_indices = np.where(T == 0)[0]
# 初始化最近邻模型
n_neighbors = ratio if replace else min(ratio, len(control_ps))
nn = NearestNeighbors(n_neighbors=n_neighbors, metric='euclidean')
nn.fit(control_ps)
# 寻找最近邻
distances, indices = nn.kneighbors(treated_ps)
# 应用卡钳值
if caliper is not None:
caliper_std = caliper * np.std(ps_scores)
valid_matches = distances < caliper_std
else:
valid_matches = np.ones_like(distances, dtype=bool)
# 构建匹配对
matched_pairs = []
for i, (t_idx, match_indices, match_distances, is_valid) in enumerate(zip(
np.where(T == 1)[0], indices, distances, valid_matches)):
for j, (c_idx, dist, valid) in enumerate(zip(match_indices, match_distances, is_valid)):
if valid:
matched_pairs.append({
'treated_index': t_idx,
'control_index': control_indices[c_idx],
'distance': dist,
'treated_ps': ps_scores.iloc[t_idx],
'control_ps': ps_scores.iloc[control_indices[c_idx]]
})
return pd.DataFrame(matched_pairs)
def caliper_match(self, ps_scores, T, caliper=0.2, ratio=1):
"""卡钳匹配"""
return self.nearest_neighbor_match(ps_scores, T, caliper=caliper, ratio=ratio)
def kernel_match(self, ps_scores, T, bandwidth=0.06):
"""核匹配"""
from scipy.stats import norm
import numpy as np
treated_ps = ps_scores[T == 1].values
control_ps = ps_scores[T == 0].values
control_indices = np.where(T == 0)[0]
treated_indices = np.where(T == 1)[0]
weights = []
for i, t_ps in enumerate(treated_ps):
# 计算核权重
kernel_weights = norm.pdf((control_ps - t_ps) / bandwidth)
kernel_weights = kernel_weights / np.sum(kernel_weights) # 标准化
weights.append({
'treated_index': treated_indices[i],
'control_weights': list(zip(control_indices, kernel_weights))
})
return weights
def match(self, data, ps_scores, T, method=None, **kwargs):
"""执行匹配"""
if method is None:
method = self.method
if method == 'nearest':
matched_pairs = self.nearest_neighbor_match(ps_scores, T, **kwargs)
elif method == 'caliper':
matched_pairs = self.caliper_match(ps_scores, T, **kwargs)
elif method == 'kernel':
return self.kernel_match(ps_scores, T, **kwargs)
else:
raise ValueError("不支持的匹配方法")
# 构建匹配后的数据集
treated_indices = matched_pairs['treated_index'].unique()
control_indices = matched_pairs['control_index'].unique()
matched_treated = data.iloc[treated_indices].copy()
matched_control = data.iloc[control_indices].copy()
matched_treated['matched_group'] = 'treated'
matched_control['matched_group'] = 'control'
self.matched_data = pd.concat([matched_treated, matched_control], axis=0)
self.matching_info = matched_pairs
return self.matched_data步骤4:平衡性检验
匹配后需要检验协变量在处理组和对照组之间是否达到平衡:
class BalanceChecker:
def __init__(self):
pass
def calculate_smd(self, variable, treated_data, control_data):
"""计算标准化均值差异"""
mean_treated = np.mean(treated_data[variable])
mean_control = np.mean(control_data[variable])
var_treated = np.var(treated_data[variable], ddof=1)
var_control = np.var(control_data[variable], ddof=1)
pooled_sd = np.sqrt((var_treated + var_control) / 2)
smd = (mean_treated - mean_control) / pooled_sd
return smd
def check_balance(self, data, covariates, treatment_var='treatment', matched_group_var='matched_group'):
"""检查匹配后平衡性"""
treated = data[data[matched_group_var] == 'treated']
control = data[data[matched_group_var] == 'control']
balance_results = []
for covariate in covariates:
# 标准化均值差异
smd = self.calculate_smd(covariate, treated, control)
# t检验
from scipy.stats import ttest_ind
t_stat, p_value = ttest_ind(treated[covariate], control[covariate])
# 方差比
var_ratio = np.var(treated[covariate], ddof=1) / np.var(control[covariate], ddof=1)
balance_results.append({
'covariate': covariate,
'smd': smd,
'smd_abs': abs(smd),
'p_value': p_value,
'var_ratio': var_ratio,
'balanced': abs(smd) < 0.1 and 0.5 < var_ratio < 2 # 平衡标准
})
return pd.DataFrame(balance_results)
def plot_balance(self, balance_before, balance_after, title='平衡性诊断图'):
"""绘制平衡性诊断图"""
import matplotlib.pyplot as plt
plt.figure(figsize=(10, 6))
# 绘制SMD绝对值
plt.subplot(1, 2, 1)
plt.scatter(balance_before['smd_abs'], range(len(balance_before)),
color='red', alpha=0.7, label='匹配前')
plt.scatter(balance_after['smd_abs'], range(len(balance_after)),
color='blue', alpha=0.7, label='匹配后')
plt.axvline(x=0.1, color='gray', linestyle='--', label='阈值 (0.1)')
plt.ylabel('协变量')
plt.xlabel('标准化均值差异绝对值')
plt.title('标准化均值差异')
plt.legend()
# 绘制方差比
plt.subplot(1, 2, 2)
plt.scatter(balance_before['var_ratio'], range(len(balance_before)),
color='red', alpha=0.7, label='匹配前')
plt.scatter(balance_after['var_ratio'], range(len(balance_after)),
color='blue', alpha=0.7, label='匹配后')
plt.axvline(x=0.5, color='gray', linestyle='--', label='下限 (0.5)')
plt.axvline(x=2, color='gray', linestyle='--', label='上限 (2)')
plt.xlabel('方差比')
plt.title('方差比')
plt.legend()
plt.tight_layout()
plt.suptitle(title, y=1.02)
plt.show()IV. 案例研究:教育对收入的影响
为了展示PSM的实际应用,我们将研究一个经典问题:大学教育对个人收入的影响。这是一个典型的观察性研究问题,因为个体是否上大学不是随机分配的,而是受到多种因素影响。
研究背景与数据生成
我们首先模拟一个包含5000个个体的数据集,包含以下变量:
I. 处理变量:大学教育(1=上大学,0=没上大学)
II. 结果变量:年收入(对数转换)
III. 协变量:
- 家庭收入
- 父母教育水平
- 认知能力测试分数
- 学习动机(未观测变量)
- 性别
- 种族
def generate_education_income_data(n=5000, seed=42):
"""生成教育-收入模拟数据"""
np.random.seed(seed)
# 生成基线特征
data = pd.DataFrame({
'family_income': np.random.lognormal(10.5, 0.8, n),
'parental_education': np.random.normal(14, 3, n),
'cognitive_ability': np.random.normal(100, 15, n),
'motivation': np.random.normal(0, 1, n), # 未观测变量
'gender': np.random.binomial(1, 0.5, n),
'race': np.random.choice([0, 1, 2], n, p=[0.6, 0.2, 0.2])
})
# 生成处理分配(大学教育)
# 处理概率受观测和未观测变量影响
treatment_propensity = (
0.4 * (data['family_income'] - np.mean(data['family_income'])) / np.std(data['family_income']) +
0.5 * (data['parental_education'] - 14) / 3 +
0.6 * (data['cognitive_ability'] - 100) / 15 +
0.7 * data['motivation'] + # 未观测变量
-0.2 * data['gender'] +
0.1 * (data['race'] == 1) + 0.3 * (data['race'] == 2) +
np.random.normal(0, 1, n)
)
data['college'] = (treatment_propensity > 0).astype(int)
# 生成结果(年收入,对数尺度)
# 真实处理效应为0.25(约28%的收入增加)
log_income = (
10.0 +
0.25 * data['college'] + # 真实处理效应
0.3 * (data['family_income'] - np.mean(data['family_income'])) / np.std(data['family_income']) +
0.4 * (data['parental_education'] - 14) / 3 +
0.5 * (data['cognitive_ability'] - 100) / 15 +
0.6 * data['motivation'] + # 未观测变量
-0.1 * data['gender'] +
0.05 * (data['race'] == 1) + 0.15 * (data['race'] == 2) +
np.random.normal(0, 0.5, n)
)
data['log_income'] = log_income
data['income'] = np.exp(log_income)
return data
# 生成数据
education_data = generate_education_income_data(n=5000)
# 查看数据摘要
print("数据摘要:")
print(education_data.describe())
print(f"\n大学比例: {education_data['college'].mean():.3f}")简单比较的问题
首先,我们进行简单的均值比较,看看大学毕业生和非大学毕业生的收入差异:
# 简单比较
simple_comparison = education_data.groupby('college')['income'].agg(['mean', 'std', 'count'])
simple_comparison['diff'] = simple_comparison['mean'] - simple_comparison.loc[0, 'mean']
simple_comparison['ratio'] = simple_comparison['mean'] / simple_comparison.loc[0, 'mean']
print("简单比较结果:")
print(simple_comparison.round(2))
# 计算简单比较的偏差
true_effect = 0.25 # 真实处理效应(对数尺度)
naive_effect_log = np.log(simple_comparison.loc[1, 'mean']) - np.log(simple_comparison.loc[0, 'mean'])
bias = naive_effect_log - true_effect
print(f"\n真实处理效应 (对数尺度): {true_effect:.3f}")
print(f"简单估计效应 (对数尺度): {naive_effect_log:.3f}")
print(f"估计偏差: {bias:.3f} ({bias/true_effect*100:.1f}%)")简单比较会高估大学教育的影响,因为上大学的人通常来自更高收入家庭、有更高认知能力和更强学习动机,这些因素本身也会影响收入。
应用PSM估计处理效应
现在,我们应用PSM来纠正选择偏差:
# 准备数据
X = education_data[['family_income', 'parental_education', 'cognitive_ability', 'gender', 'race']]
T = education_data['college']
y = education_data['log_income']
# 步骤1: 估计倾向得分
ps_estimator = PropensityScoreEstimator(method='logistic', max_iter=1000)
ps_scores = ps_estimator.estimate_ps(X, T, cv=True)
# 检查共同支持
min_ps, max_ps, overlap_ratio = ps_estimator.check_overlap(T)
# 步骤2: 执行匹配
matcher = PropensityScoreMatcher(method='caliper', caliper=0.2, ratio=1)
matched_data = matcher.match(education_data, pd.Series(ps_scores), T)
# 步骤3: 检查平衡性
balance_checker = BalanceChecker()
covariates = ['family_income', 'parental_education', 'cognitive_ability', 'gender', 'race']
# 匹配前平衡性
balance_before = balance_checker.check_balance(
education_data.assign(matched_group=np.where(T==1, 'treated', 'control')),
covariates
)
# 匹配后平衡性
balance_after = balance_checker.check_balance(matched_data, covariates)
print("匹配前后平衡性比较:")
print("匹配前SMD > 0.1的变量:", balance_before[balance_before['smd_abs'] > 0.1]['covariate'].tolist())
print("匹配后SMD > 0.1的变量:", balance_after[balance_after['smd_abs'] > 0.1]['covariate'].tolist())
# 绘制平衡性诊断图
balance_checker.plot_balance(balance_before, balance_after, '教育对收入影响研究的平衡性诊断')
# 步骤4: 估计处理效应
treated_income = matched_data[matched_data['matched_group'] == 'treated']['log_income'].mean()
control_income = matched_data[matched_data['matched_group'] == 'control']['log_income'].mean()
ate_estimate = treated_income - control_income
ate_percentage = (np.exp(ate_estimate) - 1) * 100
print(f"\nPSM估计结果:")
print(f"处理组平均收入 (对数): {treated_income:.3f}")
print(f"对照组平均收入 (对数): {control_income:.3f}")
print(f"ATE估计 (对数尺度): {ate_estimate:.3f}")
print(f"ATE估计 (百分比): {ate_percentage:.1f}%")
print(f"真实ATE (对数尺度): {true_effect:.3f}")
print(f"偏差: {ate_estimate - true_effect:.3f} ({(ate_estimate - true_effect)/true_effect*100:.1f}%)")结果解释与敏感性分析
最后,我们进行敏感性分析,评估结论对未测混淆的稳健性:
def rosenbaum_sensitivity_analysis(effect, se, gamma_range=np.arange(1, 2.6, 0.2)):
"""Rosenbaum敏感性分析"""
from scipy.stats import norm
import pandas as pd
results = []
for gamma in gamma_range:
# 计算隐藏偏倚下的p值边界
log_gamma = np.log(gamma)
delta = effect / se
p_upper = 2 * (1 - norm.cdf(abs(delta) - log_gamma/se))
p_lower = 2 * (1 - norm.cdf(abs(delta) + log_gamma/se))
results.append({
'gamma': gamma,
'p_lower': p_lower,
'p_upper': p_upper,
'significant_lower': p_lower < 0.05,
'significant_upper': p_upper < 0.05
})
return pd.DataFrame(results)
# 计算标准误
n_treated = len(matched_data[matched_data['matched_group'] == 'treated'])
n_control = len(matched_data[matched_data['matched_group'] == 'control'])
var_treated = matched_data[matched_data['matched_group'] == 'treated']['log_income'].var(ddof=1)
var_control = matched_data[matched_data['matched_group'] == 'control']['log_income'].var(ddof=1)
se_ate = np.sqrt(var_treated/n_treated + var_control/n_control)
# 进行敏感性分析
sensitivity_results = rosenbaum_sensitivity_analysis(ate_estimate, se_ate)
print("Rosenbaum敏感性分析结果:")
print(sensitivity_results.round(4))
# 找到结论变得不显著的临界Γ值
critical_gamma = sensitivity_results[sensitivity_results['significant_upper'] == False]['gamma'].min()
print(f"\n结论保持显著的最大隐藏偏倚: Γ = {critical_gamma:.2f}")
# 解释敏感性分析结果
if critical_gamma > 2.0:
print("结论对未测混淆较为稳健")
elif critical_gamma > 1.5:
print("结论对未测混淆中等敏感")
else:
print("结论对未测混淆高度敏感,需谨慎解释")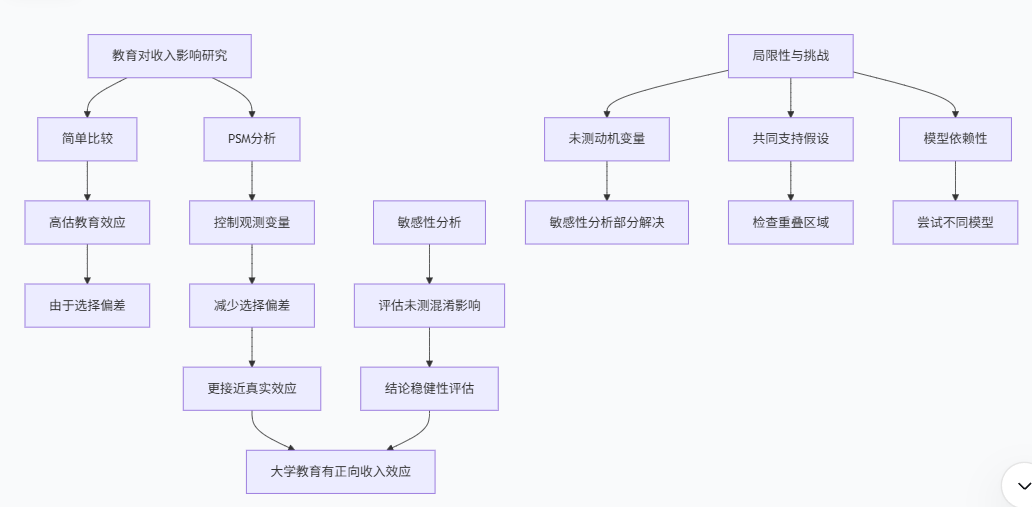
通过这个案例研究,我们展示了PSM如何帮助减少观察性研究中的选择偏差,提供更接近真实处理效应的估计。同时,敏感性分析帮助我们评估结论对未测混淆的稳健性。
V. Python代码实现与部署
在本节中,我们将构建一个完整的、可重用的PSM管道,并将其部署为Python包。这将使PSM分析更加标准化和可重复。
完整的PSM管道类
我们创建一个整合了所有步骤的PSM管道类:
class PSMipeline:
def __init__(self, treatment_var, outcome_var, covariates,
ps_method='logistic', matching_method='caliper', **kwargs):
self.treatment_var = treatment_var
self.outcome_var = outcome_var
self.covariates = covariates
self.ps_method = ps_method
self.matching_method = matching_method
self.matching_kwargs = kwargs
self.ps_estimator = None
self.ps_scores = None
self.matcher = None
self.matched_data = None
self.balance_checker = BalanceChecker()
def fit(self, data, estimate_ps=True):
"""拟合PSM模型"""
self.data = data.copy()
if estimate_ps:
# 估计倾向得分
self.ps_estimator = PropensityScoreEstimator(method=self.ps_method)
self.ps_scores = self.ps_estimator.estimate_ps(
self.data[self.covariates],
self.data[self.treatment_var],
cv=True
)
else:
# 使用预先计算的倾向得分
if not hasattr(self, 'ps_scores') or self.ps_scores is None:
raise ValueError("需要提供倾向得分或设置estimate_ps=True")
# 执行匹配
self.matcher = PropensityScoreMatcher(method=self.matching_method, **self.matching_kwargs)
self.matched_data = self.matcher.match(
self.data,
pd.Series(self.ps_scores),
self.data[self.treatment_var]
)
return self
def check_balance(self, plot=True):
"""检查平衡性"""
if self.matched_data is None:
raise ValueError("需要先拟合模型")
# 匹配前平衡性
balance_before = self.balance_checker.check_balance(
self.data.assign(matched_group=np.where(
self.data[self.treatment_var]==1, 'treated', 'control')),
self.covariates
)
# 匹配后平衡性
balance_after = self.balance_checker.check_balance(
self.matched_data,
self.covariates
)
if plot:
self.balance_checker.plot_balance(balance_before, balance_after)
return balance_before, balance_after
def estimate_effect(self, outcome_model='mean'):
"""估计处理效应"""
if self.matched_data is None:
raise ValueError("需要先拟合模型")
treated = self.matched_data[self.matched_data['matched_group'] == 'treated']
control = self.matched_data[self.matched_data['matched_group'] == 'control']
if outcome_model == 'mean':
# 简单均值比较
effect = treated[self.outcome_var].mean() - control[self.outcome_var].mean()
# 计算标准误和置信区间
n_treated = len(treated)
n_control = len(control)
var_treated = treated[self.outcome_var].var(ddof=1)
var_control = control[self.outcome_var].var(ddof=1)
se = np.sqrt(var_treated/n_treated + var_control/n_control)
ci_lower = effect - 1.96 * se
ci_upper = effect + 1.96 * se
elif outcome_model == 'regression':
# 回归调整,进一步提高精度
from sklearn.linear_model import LinearRegression
# 准备数据
X_matched = pd.get_dummies(self.matched_data[self.covariates], drop_first=True)
y_matched = self.matched_data[self.outcome_var]
t_matched = (self.matched_data['matched_group'] == 'treated').astype(int)
# 拟合模型
model = LinearRegression()
model.fit(pd.concat([X_matched, t_matched.rename('treatment')], axis=1), y_matched)
effect = model.coef_[-1]
se = np.sqrt(np.diag(model._intercept_stderr))[-1] # 简化处理
ci_lower = effect - 1.96 * se
ci_upper = effect + 1.96 * se
else:
raise ValueError("不支持的outcome_model")
return {
'effect': effect,
'se': se,
'ci_lower': ci_lower,
'ci_upper': ci_upper,
'n_treated': len(treated),
'n_control': len(control)
}
def sensitivity_analysis(self, effect_estimate, se, gamma_range=np.arange(1, 3.1, 0.2)):
"""敏感性分析"""
return rosenbaum_sensitivity_analysis(effect_estimate, se, gamma_range)
def run_full_analysis(self, data, plot=True):
"""运行完整分析"""
# 拟合模型
self.fit(data)
# 检查平衡性
balance_before, balance_after = self.check_balance(plot=plot)
# 估计处理效应
effect_result = self.estimate_effect()
# 敏感性分析
sensitivity_results = self.sensitivity_analysis(
effect_result['effect'],
effect_result['se']
)
return {
'balance_before': balance_before,
'balance_after': balance_after,
'effect_result': effect_result,
'sensitivity_results': sensitivity_results,
'matched_data': self.matched_data
}
# 使用完整管道示例
# 初始化管道
ps_pipeline = PSMipeline(
treatment_var='college',
outcome_var='log_income',
covariates=['family_income', 'parental_education', 'cognitive_ability', 'gender', 'race'],
ps_method='logistic',
matching_method='caliper',
caliper=0.2,
ratio=1
)
# 运行完整分析
results = ps_pipeline.run_full_analysis(education_data)
# 输出结果
print("PSM分析结果:")
print(f"处理效应估计: {results['effect_result']['effect']:.3f}")
print(f"95%置信区间: ({results['effect_result']['ci_lower']:.3f}, {results['effect_result']['ci_upper']:.3f})")
print(f"处理组样本量: {results['effect_result']['n_treated']}")
print(f"对照组样本量: {results['effect_result']['n_control']}")
# 检查平衡性改善
print("\n平衡性改善:")
print(f"匹配前SMD > 0.1的变量数: {sum(results['balance_before']['smd_abs'] > 0.1)}")
print(f"匹配后SMD > 0.1的变量数: {sum(results['balance_after']['smd_abs'] > 0.1)}")部署为Python包
我们将PSM实现打包为一个可安装的Python包,方便在其他项目中重用:
项目结构:
causalpsm/
│
├── __init__.py
├── core/
│ ├── __init__.py
│ ├── propensity.py # 倾向得分估计
│ ├── matching.py # 匹配算法
│ ├── balance.py # 平衡性检查
│ └── sensitivity.py # 敏感性分析
├── utils/
│ ├── __init__.py
│ └── visualization.py # 可视化工具
├── pipelines/
│ ├── __init__.py
│ └── psm_pipeline.py # 完整管道
└── tests/
├── __init__.py
└── test_psm.py # 单元测试setup.py配置:
from setuptools import setup, find_packages
setup(
name="causalpsm",
version="0.1.0",
description="A Python package for Propensity Score Matching analysis",
author="Your Name",
author_email="your.email@example.com",
packages=find_packages(),
install_requires=[
"numpy>=1.20.0",
"pandas>=1.3.0",
"scipy>=1.7.0",
"scikit-learn>=1.0.0",
"matplotlib>=3.5.0",
"seaborn>=0.11.0",
],
python_requires=">=3.8",
)使用示例:
from causalpsm import PSMipeline
from causalpsm.utils.visualization import plot_balance_diagnostics
# 初始化管道
psm = PSMipeline(
treatment_var='treatment',
outcome_var='outcome',
covariates=['age', 'gender', 'income', 'education'],
ps_method='logistic',
matching_method='caliper',
caliper=0.2
)
# 运行分析
results = psm.run_full_analysis(data)
# 生成诊断报告
plot_balance_diagnostics(
results['balance_before'],
results['balance_after'],
title='PSM平衡性诊断'
)自动化测试
为了确保代码质量,我们编写了全面的单元测试:
# tests/test_psm.py
import unittest
import numpy as np
import pandas as pd
from causalpsm.core.propensity import PropensityScoreEstimator
from causalpsm.core.matching import PropensityScoreMatcher
from causalpsm.core.balance import BalanceChecker
class TestPSM(unittest.TestCase):
def setUp(self):
"""设置测试数据"""
np.random.seed(42)
n = 1000
# 生成模拟数据
self.data = pd.DataFrame({
'x1': np.random.normal(0, 1, n),
'x2': np.random.normal(0, 1, n),
'x3': np.random.binomial(1, 0.5, n)
})
# 生成倾向得分和处理分配
true_ps = 1 / (1 + np.exp(-(0.5 + 0.8*self.data['x1'] + 0.6*self.data['x2'] + 0.4*self.data['x3'])))
self.data['treatment'] = np.random.binomial(1, true_ps)
self.data['outcome'] = 10 + 2*self.data['treatment'] + 1.5*self.data['x1'] + np.random.normal(0, 1, n)
self.covariates = ['x1', 'x2', 'x3']
def test_propensity_estimation(self):
"""测试倾向得分估计"""
estimator = PropensityScoreEstimator(method='logistic')
ps_scores = estimator.estimate_ps(
self.data[self.covariates],
self.data['treatment']
)
self.assertEqual(len(ps_scores), len(self.data))
self.assertTrue(all(0 <= p <= 1 for p in ps_scores))
def test_matching(self):
"""测试匹配过程"""
estimator = PropensityScoreEstimator(method='logistic')
ps_scores = estimator.estimate_ps(
self.data[self.covariates],
self.data['treatment']
)
matcher = PropensityScoreMatcher(method='nearest')
matched_data = matcher.match(
self.data,
pd.Series(ps_scores),
self.data['treatment']
)
# 检查匹配后样本量
treated_count = sum(matched_data['matched_group'] == 'treated')
control_count = sum(matched_data['matched_group'] == 'control')
self.assertGreater(treated_count, 0)
self.assertGreater(control_count, 0)
def test_balance_improvement(self):
"""测试平衡性改善"""
estimator = PropensityScoreEstimator(method='logistic')
ps_scores = estimator.estimate_ps(
self.data[self.covariates],
self.data['treatment']
)
matcher = PropensityScoreMatcher(method='nearest')
matched_data = matcher.match(
self.data,
pd.Series(ps_scores),
self.data['treatment']
)
# 检查平衡性
balance_checker = BalanceChecker()
balance_before = balance_checker.check_balance(
self.data.assign(matched_group=np.where(
self.data['treatment']==1, 'treated', 'control')),
self.covariates
)
balance_after = balance_checker.check_balance(matched_data, self.covariates)
# 匹配后平衡性应该改善
self.assertLess(
balance_after['smd_abs'].max(),
balance_before['smd_abs'].max() * 1.5 # 允许一定浮动
)
if __name__ == '__main__':
unittest.main()
VI. PSM的变体与进阶方法
基础的倾向得分匹配方法有许多扩展和变体,这些方法针对PSM的不同局限性提供了改进方案。本节将介绍几种重要的PSM变体和进阶方法。
1. 广义倾向得分
当处理变量不是二值而是多值或连续时,需要使用广义倾向得分(Generalized Propensity Score)。
定义:对于连续处理T,广义倾向得分是处理条件下的密度函数:
$$r(t, X) = f_{T|X}(t|X)$$
应用示例:
class GeneralizedPropensityScore:
def __init__(self, method='kernel'):
self.method = method
self.model = None
def estimate_gps(self, X, T):
"""估计广义倾向得分"""
if self.method == 'kernel':
from sklearn.neighbors import KernelDensity
# 对每个样本估计条件密度
gps_scores = np.zeros(len(T))
for i in range(len(T)):
# 留一法估计密度
kde = KernelDensity(kernel='gaussian', bandwidth=0.5)
kde.fit(T.drop(i).values.reshape(-1, 1))
gps_scores[i] = np.exp(kde.score([[T.iloc[i]]]))
return gps_scores
elif self.method == 'normal':
# 假设正态分布,使用线性回归估计条件分布
from sklearn.linear_model import LinearRegression
model = LinearRegression()
model.fit(X, T)
predictions = model.predict(X)
residuals = T - predictions
sigma = np.std(residuals)
gps_scores = 1/(sigma * np.sqrt(2 * np.pi)) * np.exp(-0.5 * (residuals/sigma)**2)
return gps_scores
else:
raise ValueError("不支持的GPS估计方法")
# 使用示例
# 生成连续处理数据
np.random.seed(42)
n = 1000
X = np.random.normal(0, 1, (n, 3))
T = 2 + 0.8*X[:,0] + 0.6*X[:,1] + np.random.normal(0, 1, n)
Y = 10 + 0.5*T + 1.2*X[:,0] + 0.8*X[:,1] + np.random.normal(0, 1, n)
# 估计广义倾向得分
gps_estimator = GeneralizedPropensityScore(method='normal')
gps_scores = gps_estimator.estimate_gps(pd.DataFrame(X), pd.Series(T))
print(f"广义倾向得分范围: [{gps_scores.min():.4f}, {gps_scores.max():.4f}]")2. 双重稳健估计
双重稳健估计(Doubly Robust Estimation)结合了倾向得分和结果模型的优势,只要其中一个模型正确设定,就能得到一致估计。
双重稳健估计量:
\hat{\tau}_{DR} = \frac{1}{N} \sum_{i=1}^N \left[ \frac{T_i(Y_i - \hat{\mu}_1(X_i))}{\hat{e}(X_i)} + \hat{\mu}_1(X_i) \right] - \left[ \frac{(1-T_i)(Y_i - \hat{\mu}_0(X_i))}{1-\hat{e}(X_i)} + \hat{\mu}_0(X_i) \right]
其中:
- (\hat{e}(X_i)) 是倾向得分
- (\hat{\mu}_1(X_i)) 是处理组结果模型预测
- (\hat{\mu}_0(X_i)) 是对照组结果模型预测
class DoublyRobustEstimator:
def __init__(self):
self.ps_model = None
self.outcome_model_treated = None
self.outcome_model_control = None
def fit(self, X, T, Y):
"""拟合双重稳健模型"""
from sklearn.linear_model import LinearRegression
# 拟合倾向得分模型
self.ps_model = LogisticRegression()
self.ps_model.fit(X, T)
ps_scores = self.ps_model.predict_proba(X)[:, 1]
# 拟合结果模型
self.outcome_model_treated = LinearRegression()
self.outcome_model_control = LinearRegression()
self.outcome_model_treated.fit(X[T == 1], Y[T == 1])
self.outcome_model_control.fit(X[T == 0], Y[T == 0])
return ps_scores
def estimate_effect(self, X, T, Y):
"""估计双重稳健处理效应"""
ps_scores = self.ps_model.predict_proba(X)[:, 1]
# 预测潜在结果
mu1 = self.outcome_model_treated.predict(X)
mu0 = self.outcome_model_control.predict(X)
# 计算双重稳健估计量
dr1 = (T * (Y - mu1)) / ps_scores + mu1
dr0 = ((1 - T) * (Y - mu0)) / (1 - ps_scores) + mu0
ate = np.mean(dr1) - np.mean(dr0)
return ate
# 使用示例
dr_estimator = DoublyRobustEstimator()
ps_scores = dr_estimator.fit(X, T, Y)
ate_dr = dr_estimator.estimate_effect(X, T, Y)
print(f"双重稳健ATE估计: {ate_dr:.3f}")3. 机器学习在PSM中的应用
机器学习方法可以用于改进倾向得分估计和结果预测:
class MLPropensityScore:
def __init__(self, ps_model=None, outcome_model=None):
self.ps_model = ps_model or RandomForestClassifier()
self.outcome_model = outcome_model or RandomForestRegressor()
self.ps_scores = None
def estimate_ps_ml(self, X, T, cv=False):
"""使用机器学习估计倾向得分"""
if cv:
from sklearn.model_selection import cross_val_predict
self.ps_scores = cross_val_predict(
self.ps_model, X, T,
method='predict_proba', cv=5
)[:, 1]
else:
self.ps_model.fit(X, T)
self.ps_scores = self.ps_model.predict_proba(X)[:, 1]
return self.ps_scores
def estimate_effect_ml(self, X, T, Y, method='aipw'):
"""使用机器学习方法估计处理效应"""
from sklearn.model_selection import cross_val_predict
# 估计倾向得分
ps_scores = self.estimate_ps_ml(X, T, cv=True)
if method == 'aipw':
# 增强逆概率加权(AIPW)
# 使用交叉拟合避免过拟合
from sklearn.model_selection import KFold
dr_estimates = []
kf = KFold(n_splits=5, shuffle=True, random_state=42)
for train_idx, test_idx in kf.split(X):
X_train, X_test = X.iloc[train_idx], X.iloc[test_idx]
T_train, T_test = T.iloc[train_idx], T.iloc[test_idx]
Y_train, Y_test = Y.iloc[train_idx], Y.iloc[test_idx]
# 训练结果模型
outcome_model_treated = clone(self.outcome_model)
outcome_model_control = clone(self.outcome_model)
outcome_model_treated.fit(X_train[T_train == 1], Y_train[T_train == 1])
outcome_model_control.fit(X_train[T_train == 0], Y_train[T_train == 0])
# 预测潜在结果
mu1_test = outcome_model_treated.predict(X_test)
mu0_test = outcome_model_control.predict(X_test)
# 计算AIPW估计量
dr1 = (T_test * (Y_test - mu1_test)) / ps_scores[test_idx] + mu1_test
dr0 = ((1 - T_test) * (Y_test - mu0_test)) / (1 - ps_scores[test_idx]) + mu0_test
dr_estimates.extend(dr1 - dr0)
ate = np.mean(dr_estimates)
elif method == 'tmle':
# 目标最大似然估计(TMLE)
# 实现略复杂,这里简化表示
ate = self._estimate_tmle(X, T, Y, ps_scores)
else:
raise ValueError("不支持的估计方法")
return ate
# 使用示例
ml_ps = MLPropensityScore(
ps_model=RandomForestClassifier(n_estimators=100),
outcome_model=RandomForestRegressor(n_estimators=100)
)
ate_ml = ml_ps.estimate_effect_ml(X, T, Y, method='aipw')
print(f"机器学习ATE估计: {ate_ml:.3f}")4. 分层倾向得分匹配
当样本量较大时,可以使用分层倾向得分匹配,先按倾向得分分层,再在层内进行匹配:
class StratifiedPSM:
def __init__(self, n_strata=5):
self.n_strata = n_strata
self.strata_boundaries = None
def create_strata(self, ps_scores):
"""创建倾向得分分层"""
self.strata_boundaries = np.quantile(ps_scores, np.linspace(0, 1, self.n_strata + 1))
strata = np.digitize(ps_scores, self.strata_boundaries[1:-1])
return strata
def stratified_match(self, data, ps_scores, T, covariates):
"""分层匹配"""
strata = self.create_strata(ps_scores)
data['stratum'] = strata
matched_data = []
for stratum in range(1, self.n_strata + 1):
stratum_data = data[data['stratum'] == stratum]
stratum_treated = stratum_data[T == 1]
stratum_control = stratum_data[T == 0]
if len(stratum_treated) > 0 and len(stratum_control) > 0:
# 在层内进行匹配
matcher = PropensityScoreMatcher(method='nearest')
stratum_ps = ps_scores[stratum_data.index]
stratum_matched = matcher.match(
stratum_data, pd.Series(stratum_ps), stratum_data[T.name]
)
matched_data.append(stratum_matched)
return pd.concat(matched_data, axis=0)
# 使用示例
stratified_matcher = StratifiedPSM(n_strata=5)
stratified_matched = stratified_matcher.stratified_match(
education_data, ps_scores, education_data['college'], covariates
)
print(f"分层匹配后样本量: {len(stratified_matched)}")
这些进阶方法扩展了PSM的应用范围,提高了估计的稳健性和效率,使研究者能够处理更复杂的因果推断问题。
VII. PSM的局限性及应对策略
尽管倾向得分匹配是强大的因果推断工具,但它也有重要的局限性。了解这些局限性并采取适当的应对策略对于正确应用PSM至关重要。
主要局限性及应对策略
局限性 | 描述 | 应对策略 |
|---|---|---|
未测混淆 | 无法控制未观测的混淆变量 | 进行敏感性分析,评估未测混淆的影响 |
共同支持假设 violation | 处理组和对照组倾向得分范围不重叠 | 检查重叠区域,限制分析到共同支持区域 |
模型误设 | 倾向得分模型或结果模型设定错误 | 尝试多种模型设定,使用双重稳健估计 |
匹配质量差 | 匹配后协变量仍不平衡 | 检查平衡性,尝试不同匹配方法,调整卡钳值 |
方差增加 | 匹配过程可能减少样本量,增加方差 | 使用有放回匹配,核匹配或加权方法 |
外推问题 | 匹配样本可能不能代表总体 | 明确目标总体,评估样本代表性 |
未测混淆的敏感性分析
未测混淆是PSM最严重的局限性之一。我们之前介绍了Rosenbaum边界方法,以下是更详细的实现:
class SensitivityAnalysisAdvanced:
def __init__(self):
pass
def rosenbaum_bounds(self, effect, se, gamma_range=np.arange(1, 3.1, 0.1)):
"""计算Rosenbaum边界"""
from scipy.stats import norm
import pandas as pd
results = []
for gamma in gamma_range:
log_gamma = np.log(gamma)
delta = effect / se
# 计算p值边界
p_upper = 2 * (1 - norm.cdf(abs(delta) - log_gamma/se))
p_lower = 2 * (1 - norm.cdf(abs(delta) + log_gamma/se))
# 计算置信区间边界
ci_upper = effect + log_gamma
ci_lower = effect - log_gamma
results.append({
'gamma': gamma,
'p_lower': p_lower,
'p_upper': p_upper,
'ci_lower': ci_lower,
'ci_upper': ci_upper,
'significant_lower': p_lower < 0.05,
'significant_upper': p_upper < 0.05
})
return pd.DataFrame(results)
def plot_sensitivity(self, sensitivity_results, true_effect=None):
"""绘制敏感性分析图"""
import matplotlib.pyplot as plt
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(15, 6))
# p值图
ax1.plot(sensitivity_results['gamma'], sensitivity_results['p_lower'],
label='p值下限', marker='o')
ax1.plot(sensitivity_results['gamma'], sensitivity_results['p_upper'],
label='p值上限', marker='o')
ax1.axhline(y=0.05, color='r', linestyle='--', label='显著性阈值 (0.05)')
ax1.set_xlabel('隐藏偏倚强度 (Γ)')
ax1.set_ylabel('p值')
ax1.set_title('Rosenbaum敏感性分析 - p值')
ax1.legend()
ax1.grid(True)
# 效应值图
ax2.plot(sensitivity_results['gamma'], sensitivity_results['ci_lower'],
label='CI下限', marker='o')
ax2.plot(sensitivity_results['gamma'], sensitivity_results['ci_upper'],
label='CI上限', marker='o')
if true_effect is not None:
ax2.axhline(y=true_effect, color='g', linestyle='--', label='真实效应')
ax2.set_xlabel('隐藏偏倚强度 (Γ)')
ax2.set_ylabel('处理效应')
ax2.set_title('Rosenbaum敏感性分析 - 效应值')
ax2.legend()
ax2.grid(True)
plt.tight_layout()
plt.show()
return fig
def assess_sensitivity(self, effect, se, alpha=0.05):
"""评估敏感性"""
# 找到结论变得不显著的临界Γ值
gamma_values = np.arange(1, 5, 0.01)
critical_gamma = None
for gamma in gamma_values:
log_gamma = np.log(gamma)
p_upper = 2 * (1 - norm.cdf(abs(effect/se) - log_gamma/se))
if p_upper >= alpha:
critical_gamma = gamma
break
# 解释结果
if critical_gamma is None:
robustness = "非常稳健"
interpretation = "即使存在很强的未测混淆,结论仍然显著"
elif critical_gamma >= 2.0:
robustness = "较为稳健"
interpretation = f"结论能够抵抗中等强度的未测混淆 (Γ ≥ {critical_gamma:.2f})"
elif critical_gamma >= 1.5:
robustness = "中等敏感"
interpretation = f"结论对未测混淆较为敏感 (Γ = {critical_gamma:.2f})"
else:
robustness = "高度敏感"
interpretation = f"结论对未测混淆高度敏感 (Γ = {critical_gamma:.2f})"
return {
'critical_gamma': critical_gamma,
'robustness': robustness,
'interpretation': interpretation
}
# 使用示例
sensitivity_advanced = SensitivityAnalysisAdvanced()
sensitivity_results = sensitivity_advanced.rosenbaum_bounds(ate_estimate, se_ate)
# 绘制敏感性分析图
sensitivity_advanced.plot_sensitivity(sensitivity_results, true_effect=0.25)
# 评估敏感性
sensitivity_assessment = sensitivity_advanced.assess_sensitivity(ate_estimate, se_ate)
print(f"敏感性评估: {sensitivity_assessment['robustness']}")
print(f"解释: {sensitivity_assessment['interpretation']}")共同支持问题的诊断与处理
共同支持假设 violation 会导致外推问题和估计偏差。以下是诊断和处理方法:
class CommonSupportDiagnosis:
def __init__(self):
pass
def diagnose_common_support(self, ps_scores, T, plot=True):
"""诊断共同支持问题"""
ps_treated = ps_scores[T == 1]
ps_control = ps_scores[T == 0]
# 计算重叠统计量
min_ps = max(np.min(ps_treated), np.min(ps_control))
max_ps = min(np.max(ps_treated), np.max(ps_control))
overlap_ratio = (len(ps_treated[(ps_treated >= min_ps) & (ps_treated <= max_ps)]) +
len(ps_control[(ps_control >= min_ps) & (ps_control <= max_ps)])) / len(T)
# 计算非重叠样本比例
non_overlap_treated = len(ps_treated[ps_treated < min_ps]) + len(ps_treated[ps_treated > max_ps])
non_overlap_control = len(ps_control[ps_control < min_ps]) + len(ps_control[ps_control > max_ps])
non_overlap_ratio = (non_overlap_treated + non_overlap_control) / len(T)
if plot:
import matplotlib.pyplot as plt
plt.figure(figsize=(10, 6))
plt.hist(ps_control, bins=20, alpha=0.5, label='对照组', density=True)
plt.hist(ps_treated, bins=20, alpha=0.5, label='处理组', density=True)
plt.axvline(x=min_ps, color='red', linestyle='--', label='共同支持下限')
plt.axvline(x=max_ps, color='red', linestyle='--', label='共同支持上限')
plt.xlabel('倾向得分')
plt.ylabel('密度')
plt.title('共同支持诊断')
plt.legend()
plt.show()
return {
'min_ps': min_ps,
'max_ps': max_ps,
'overlap_ratio': overlap_ratio,
'non_overlap_ratio': non_overlap_ratio,
'non_overlap_treated': non_overlap_treated,
'non_overlap_control': non_overlap_control
}
def handle_common_support(self, data, ps_scores, T, method='trimming'):
"""处理共同支持问题"""
diagnosis = self.diagnose_common_support(ps_scores, T, plot=False)
min_ps, max_ps = diagnosis['min_ps'], diagnosis['max_ps']
if method == 'trimming':
# 修剪法:删除非重叠样本
in_support = (ps_scores >= min_ps) & (ps_scores <= max_ps)
return data[in_support], ps_scores[in_support], T[in_support]
elif method == 'weighting':
# 加权法:使用重叠权重
weights = np.zeros(len(T))
weights[T == 1] = np.minimum(ps_scores[T == 1], 1 - ps_scores[T == 1]) / ps_scores[T == 1]
weights[T == 0] = np.minimum(ps_scores[T == 0], 1 - ps_scores[T == 0]) / (1 - ps_scores[T == 0])
return data, ps_scores, T, weights
else:
raise ValueError("不支持的处理方法")
# 使用示例
support_diagnosis = CommonSupportDiagnosis()
diagnosis_result = support_diagnosis.diagnose_common_support(ps_scores, T)
print("共同支持诊断结果:")
print(f"共同支持区域: [{diagnosis_result['min_ps']:.3f}, {diagnosis_result['max_ps']:.3f}]")
print(f"重叠样本比例: {diagnosis_result['overlap_ratio']:.3f}")
print(f"非重叠样本比例: {diagnosis_result['non_overlap_ratio']:.3f}")
# 处理共同支持问题
if diagnosis_result['non_overlap_ratio'] > 0.1:
print("\n检测到显著共同支持问题,进行处理...")
data_trimmed, ps_trimmed, T_trimmed = support_diagnosis.handle_common_support(
education_data, ps_scores, T, method='trimming'
)
print(f"修剪后样本量: {len(data_trimmed)}")模型不确定性的Bootstrap评估
为了评估PSM估计对模型设定的敏感性,可以使用Bootstrap方法:
class BootstrapPSM:
def __init__(self, n_bootstrap=100):
self.n_bootstrap = n_bootstrap
self.bootstrap_estimates = []
def bootstrap_analysis(self, data, treatment_var, outcome_var, covariates):
"""Bootstrap分析"""
from sklearn.utils import resample
for i in range(self.n_bootstrap):
# 有放回抽样
bootstrap_sample = resample(data, replace=True, random_state=i)
try:
# 估计倾向得分
ps_estimator = PropensityScoreEstimator(method='logistic')
ps_scores = ps_estimator.estimate_ps(
bootstrap_sample[covariates],
bootstrap_sample[treatment_var]
)
# 执行匹配
matcher = PropensityScoreMatcher(method='caliper', caliper=0.2)
matched_data = matcher.match(
bootstrap_sample, pd.Series(ps_scores), bootstrap_sample[treatment_var]
)
# 估计处理效应
treated_mean = matched_data[matched_data['matched_group'] == 'treated'][outcome_var].mean()
control_mean = matched_data[matched_data['matched_group'] == 'control'][outcome_var].mean()
effect = treated_mean - control_mean
self.bootstrap_estimates.append(effect)
except Exception as e:
print(f"Bootstrap迭代 {i} 失败: {str(e)}")
continue
return self.bootstrap_estimates
def summarize_bootstrap(self):
"""总结Bootstrap结果"""
if not self.bootstrap_estimates:
raise ValueError("需要先运行Bootstrap分析")
estimates = np.array(self.bootstrap_estimates)
mean_effect = np.mean(estimates)
se_effect = np.std(estimates, ddof=1)
ci_lower = np.percentile(estimates, 2.5)
ci_upper = np.percentile(estimates, 97.5)
return {
'mean_effect': mean_effect,
'se_effect': se_effect,
'ci_lower': ci_lower,
'ci_upper': ci_upper,
'n_successful': len(estimates)
}
# 使用示例
bootstrap_psm = BootstrapPSM(n_bootstrap=200)
bootstrap_estimates = bootstrap_psm.bootstrap_analysis(
education_data, 'college', 'log_income', covariates
)
bootstrap_summary = bootstrap_psm.summarize_bootstrap()
print("Bootstrap分析结果:")
print(f"平均效应: {bootstrap_summary['mean_effect']:.3f}")
print(f"标准误: {bootstrap_summary['se_effect']:.3f}")
print(f"95%置信区间: ({bootstrap_summary['ci_lower']:.3f}, {bootstrap_summary['ci_upper']:.3f})")
print(f"成功迭代次数: {bootstrap_summary['n_successful']}/{200}")
通过识别PSM的局限性并采取适当的应对策略,研究人员可以提高因果推断的可靠性和有效性,从而在观察性研究中获得更可信的结论。
VIII. 结论与最佳实践
PSM的核心价值与适用场景
倾向得分匹配在观察性研究中具有不可替代的价值:
I. 模拟随机化:在无法进行随机实验时,PSM提供了近似随机化的解决方案
II. 减少选择偏差:通过平衡协变量分布,减少处理组和对照组的系统性差异
III. 提高透明度:匹配过程直观透明,便于理解和验证
IV. 灵活性:可以与其他因果推断方法结合使用,形成更强大的分析框架
适用场景:
- 评估政策干预效果
- 研究医疗处理效果
- 分析教育项目影响
- 评估市场营销活动效果
- 任何存在选择偏差的观察性研究
完整的最佳实践指南
为了确保PSM分析的质量和可靠性,我们建议遵循以下最佳实践:
1. 研究设计阶段
实践 | 说明 | 检查点 |
|---|---|---|
明确因果问题 | 清晰定义处理变量、结果变量和目标参数(ATE/ATT) | 是否明确定义了反事实问题? |
变量选择 | 包括所有与处理分配和结果相关的预处理变量 | 是否避免了"坏控制"(处理后的变量)? |
样本大小规划 | 确保有足够的样本量,特别是处理组样本 | 处理组样本量是否足够? |
2. 数据预处理阶段
实践 | 说明 | 检查点 |
|---|---|---|
探索性数据分析 | 全面了解数据特征和分布 | 是否识别了异常值和缺失模式? |
缺失值处理 | 使用适当方法处理缺失数据 | 缺失数据是否影响样本代表性? |
变量转换 | 必要时进行变量转换(如对数转换) | 变量分布是否适合模型假设? |
3. 倾向得分估计阶段
实践 | 说明 | 检查点 |
|---|---|---|
模型选择 | 尝试多种模型(逻辑回归、随机森林等) | 模型预测准确性如何? |
模型诊断 | 检查模型校准和拟合优度 | 倾向得分分布是否合理? |
共同支持检查 | 评估处理组和得对照组的得分重叠 | 重叠区域是否足够? |
4. 匹配阶段
实践 | 说明 | 检查点 |
|---|---|---|
匹配方法选择 | 尝试不同匹配方法(最近邻、卡钳、核匹配等) | 匹配方法是否适合数据特征? |
卡钳值选择 | 根据倾向得分标准差选择适当卡钳值 | 卡钳值是否平衡了匹配质量和样本量? |
匹配比例 | 考虑使用1:1或多重匹配 | 匹配比例是否适当? |
5. 后匹配分析阶段
实践 | 说明 | 检查点 |
|---|---|---|
平衡性检验 | 全面检查匹配后协变量平衡 | SMD是否小于0.1?方差比是否接近1? |
处理效应估计 | 使用适当方法估计处理效应 | 是否考虑了匹配设计? |
敏感性分析 | 评估未测混淆的影响 | 结论对未测混淆的稳健性如何? |
6. 结果报告与解释阶段
实践 | 说明 | 检查点 |
|---|---|---|
透明报告 | 详细报告所有分析选择和参数设定 | 是否足够详细以便复现? |
局限性讨论 | 坦诚讨论PSM的局限性和假设 | 是否讨论了未测混淆等问题? |
谨慎解释 | 避免过度宣称因果结论 | 结论是否考虑了不确定性? |
PSM与其他方法的结合
PSM可以与其他因果推断方法结合使用,形成更强大的分析框架:
I. PSM + 双重稳健估计:结合倾向得分和结果模型的优势
II. PSM + 差分法:在面板数据中同时控制时间不变和时变混淆
III. PSM + 工具变量:处理未测混淆问题
IV. PSM + 机器学习:利用机器学习方法处理高维数据和复杂关系
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

