百度团队打造 基于HelixFold3 的端到端AI反向虚拟筛选技术 助力 小分子蛋白靶标识别
百度团队打造 基于HelixFold3 的端到端AI反向虚拟筛选技术 助力 小分子蛋白靶标识别

DrugIntel
发布于 2026-02-04 14:23:13
发布于 2026-02-04 14:23:13

在药物研发领域,小分子蛋白靶标识别(反向筛选)是解析药物作用机制、推动老药新用、预测脱靶效应的核心计算技术。然而,传统分步式反向筛选流程存在误差累积、结构建模精度不足等痛点,严重制约了靶标发现的效率与准确性。
近期,百度PaddleHelix团队在预印版的研究论文《End-to-End Reverse Screening Identifies Protein Targets of Small Molecules Using HelixFold3》,提出了一种基于HelixFold3的端到端反向筛选策略,从根本上缓解了传统方法的技术瓶颈,为药物研发提供了高效工具。
一、传统反向筛选的技术痛点
反向筛选的核心目标是从海量蛋白库中,精准定位与小分子配体具有高亲和力的靶标蛋白。传统基于结构的反向筛选采用分步式流程,主要包含四个核心环节:
- 靶标蛋白结构获取:依赖实验解析结构或同源建模,静态结构难以模拟配体结合后的构象变化
- 结合口袋识别:依赖P2Rank、fpocket等工具,易筛选出无生物学意义的表面空腔
- 分子对接:AutoDock Vina等工具的对接精度受限于初始结构与口袋定义,难以捕捉诱导契合效应
- 亲和力打分排序:基于物理模型的打分函数,对构象误差高度敏感,易导致假阳性/假阴性结果
上述环节彼此独立,误差会逐级传递放大,最终导致靶标识别成功率偏低。此外,传统方法无法同时优化蛋白折叠与配体结合过程,难以精准表征蛋白-配体复合物的动态相互作用。
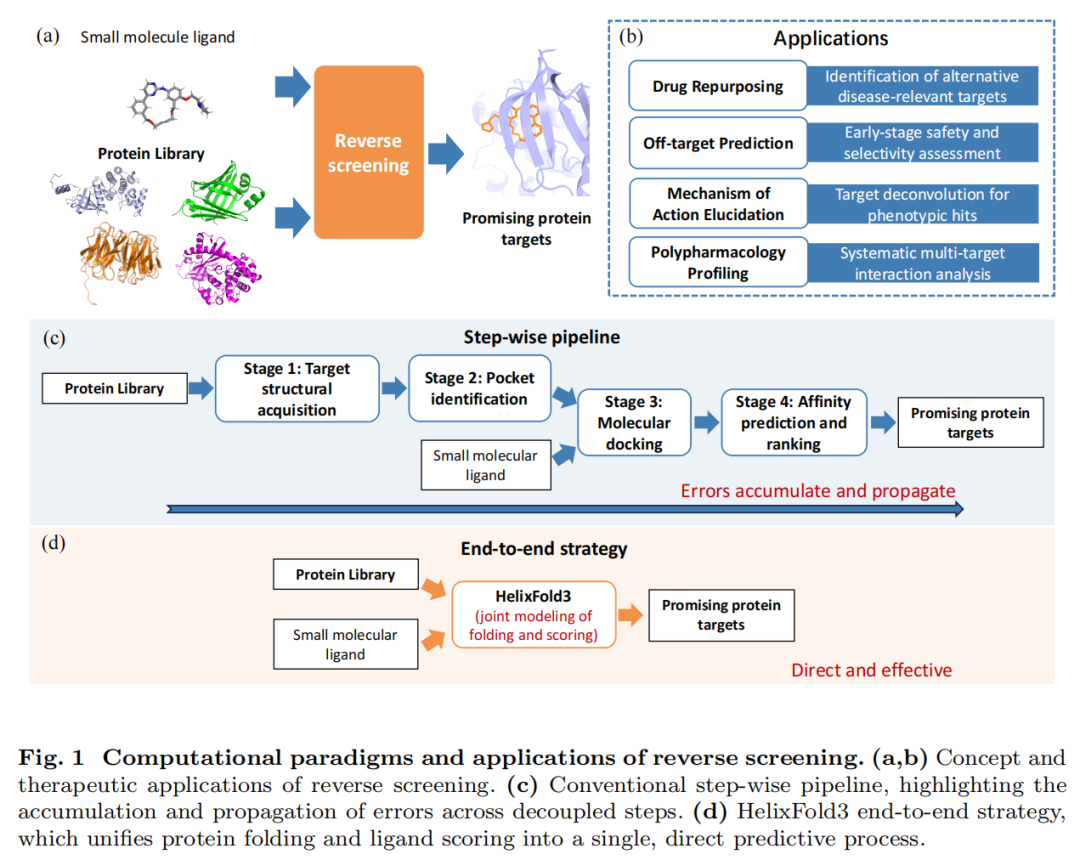
二、HelixFold3端到端策略的核心创新
该研究的核心突破在于构建了一体化的蛋白-配体复合物预测与筛选框架,将蛋白折叠、配体对接、亲和力评估三个关键步骤整合为一个联合优化过程,从根本上消除了分步流程的误差传递问题。
1. 技术原理:一体化建模与精准打分
HelixFold3是一款媲美AlphaFold3的高精度生物分子结构预测模型,其核心优势在于:
- 极简输入需求:仅需提供蛋白氨基酸序列和小分子SMILES表达式,无需预先获取蛋白三维结构或手动定义结合口袋
- 联合建模能力:同步完成蛋白折叠构象预测与配体-蛋白对接过程,可精准捕捉结合诱导的蛋白构象变化
- 内置置信度评分:输出两种核心评估指标——界面模板建模分数(ipTM)和配体局部距离差异分数(pLDDT),用于量化复合物结构的可靠性,并实现靶标蛋白的精准排序
2. 性能验证:全面超越传统分步方法
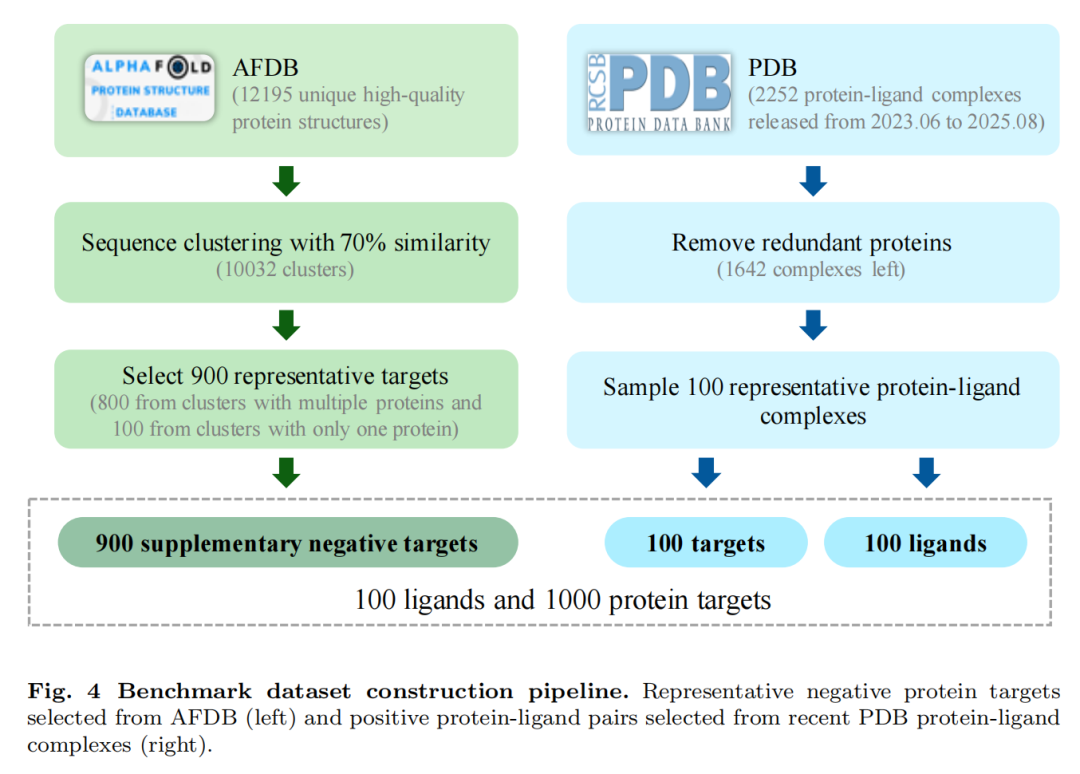
研究团队构建了包含100个高置信度蛋白-配体复合物,900个阴性蛋白的基准数据集,将HelixFold3端到端策略与三种传统分步流程(标准流程、基于真实蛋白结构的流程、基于真实结构+真实口袋的流程)进行了系统性对比,核心结果如下:
- 靶标识别效率大幅提升:端到端策略的Top-1%靶标识别成功率达38.0%,Top-10%成功率达71.0%,较传统分步方法提升数倍,即便是使用真实晶体结构与口袋坐标的分步流程,也无法企及这一性能
- 结构预测精度显著优化:在蛋白链水平,预测结构与真实结构的RMSD<0.5Å的比例达81%;在结合口袋区域,该比例达58%,远高于传统方法预测的未结合态蛋白结构
- 结合位点定位精准度高:48%的预测结合位点与真实位点的距离≤2Å,而传统口袋识别工具的这一比例仅为27%,未结合态蛋白结构的预测则完全无法定位有效结合位点
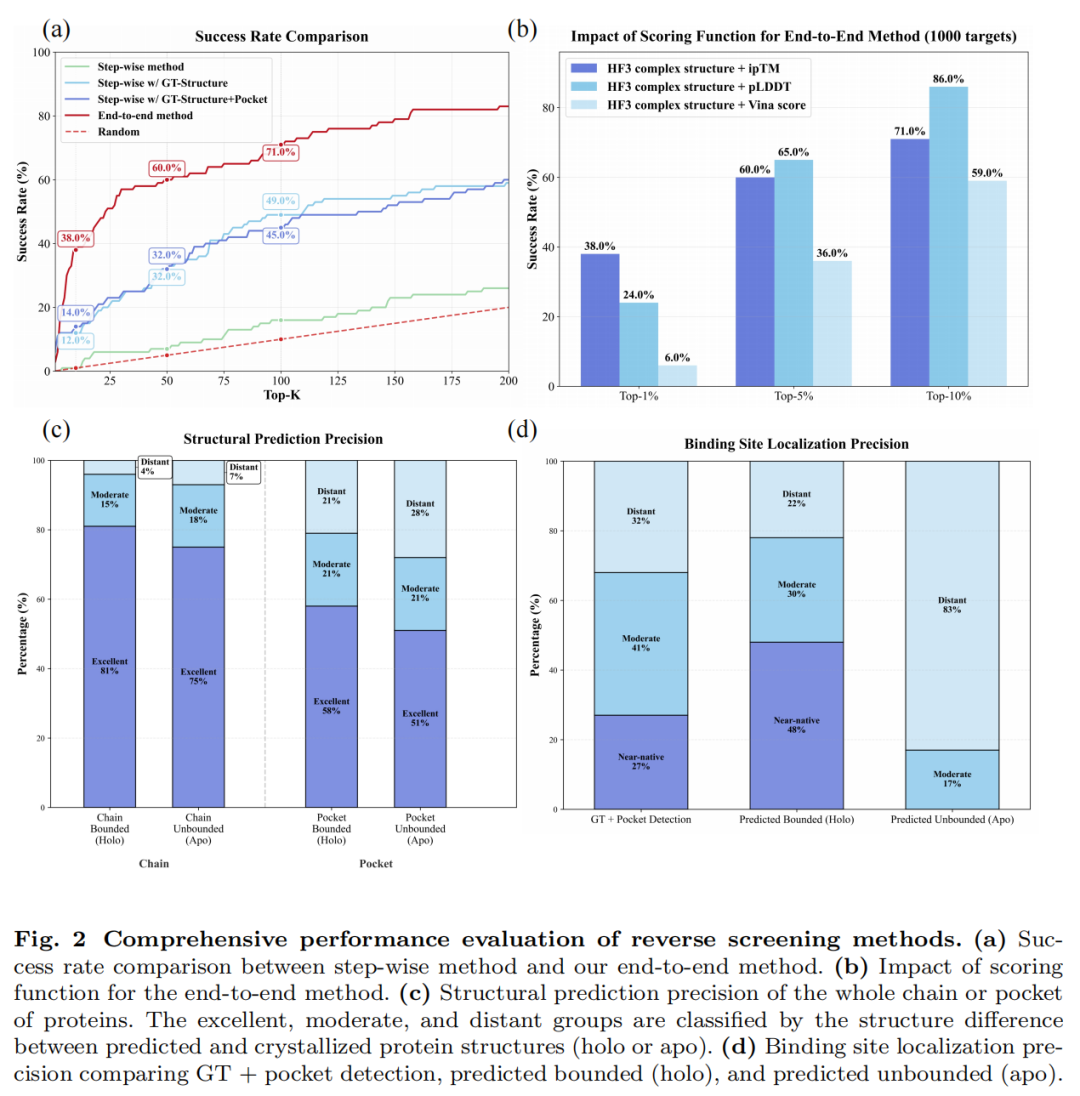
3. 打分函数性能对比:内置指标完胜传统工具
研究进一步对比了不同打分函数对筛选结果的影响,发现:
- HelixFold3内置的ipTM和pLDDT指标,远优于基于物理的AutoDock Vina打分函数
- ipTM在高优先级靶标筛选中表现最优,Top-1%成功率达38%;pLDDT在广谱筛选中更具优势,Top-10%成功率达86%
- 这一结果证明,基于深度学习的置信度评分,更适合表征蛋白-配体复合物的真实结合强度
三、实战应用:老药新用与脱靶效应预测
为验证技术的实际应用价值,研究团队针对两款临床经典药物开展了回顾性靶标筛选验证,充分展现了该策略在药物研发中的核心应用场景。
1. 伊马替尼:老药新用的靶标精准定位
伊马替尼是治疗慢性粒细胞白血病(CML)的一线药物,后被成功重定位用于胃肠间质瘤(GIST)的治疗。研究团队构建了包含176个候选蛋白的靶标库,其中包含10个已知结合靶标。
- 端到端策略成功将CML的核心靶标ABL1、GIST的关键靶标KIT/PDGFRA全部纳入Top-40排名
- pLDDT评分将ABL1列为第1位,ipTM评分将PDGFRA列为第3位,两种评分的Top-10靶标中均包含5个已知结合靶标,命中率达50%
- 验证了该技术在老药新用靶标发现中的核心价值
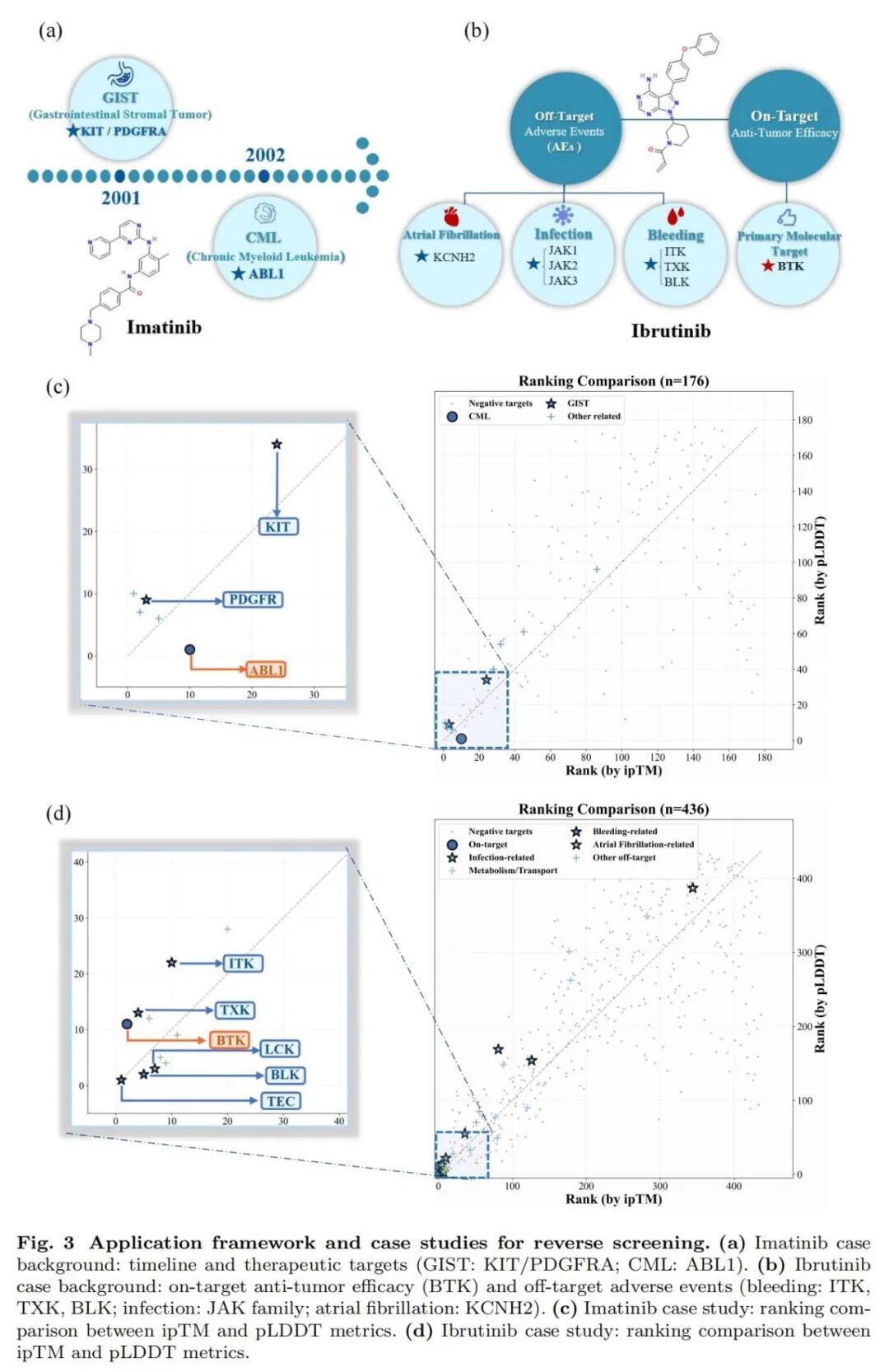
2. 伊布替尼:脱靶效应的精准预测
伊布替尼是治疗B细胞恶性肿瘤的BTK抑制剂,其临床副作用(出血、感染、房颤)与脱靶激酶结合密切相关。研究团队构建了436个候选蛋白的靶标库,包含26个已知结合靶标。
- 精准识别核心靶标BTK,ipTM排名第2,pLDDT排名第11
- 对出血相关脱靶激酶(TEC、TXK、BLK、ITK)的Top-10识别成功率达100%
- 对感染相关靶标(LCK、ITK)也实现高效筛选,但对跨膜离子通道靶标(如房颤相关的hERG通道)的预测能力较弱,这也指出了该技术当前的局限性
四、总结与展望
基于HelixFold3的端到端反向筛选技术,通过一体化的蛋白-配体复合物建模,打破了传统分步流程的技术瓶颈,实现了靶标识别效率与精度的双重飞跃。该技术不仅为药物重定位、脱靶效应预测提供了高效工具,也为解析小分子的多靶标作用机制、推动多靶点药物设计奠定了坚实基础。
当前,该技术在跨膜蛋白靶标预测方面仍存在不足,未来可通过扩展跨膜蛋白训练数据、优化模型对膜环境的表征能力进一步提升性能。随着模型的持续迭代,端到端反向筛选有望成为药物研发领域的标准工具,加速新药从实验室走向临床的进程。
参考文献:Xu S, Ye X, Zhu M, et al. End-to-End Reverse Screening Identifies Protein Targets of Small Molecules Using HelixFold3[J]. arXiv preprint arXiv:2601.13693, 2026.
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-02-02,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

