没有将“DefaultAssay”应用于类“列表”对象的适用方法
没有将“DefaultAssay”应用于类“列表”对象的适用方法
提问于 2022-04-20 11:37:46
我用"seurat“来处理老师给我的一些数据。我首先提取了B细胞的部分,并将其定义为pbmc。然后我试着分析里面的线粒体。但是,我得到了以下错误,我不知道如何修复它。
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^mt-")
Error in UseMethod(generic = "DefaultAssay", object = object) :
no applicable method for 'DefaultAssay' applied to an object of class "list"下面是我的操作所涉及的代码:
install.packages("Seurat")
install.packages("dplyr")
install.packages("patchwork")
library(dplyr)
library(Seurat)
library(patchwork)
library(ggplot2)
setwd('E:/YL')
rm(list=ls())
pbmc.data <- readRDS("E:/YL/BB.rds")
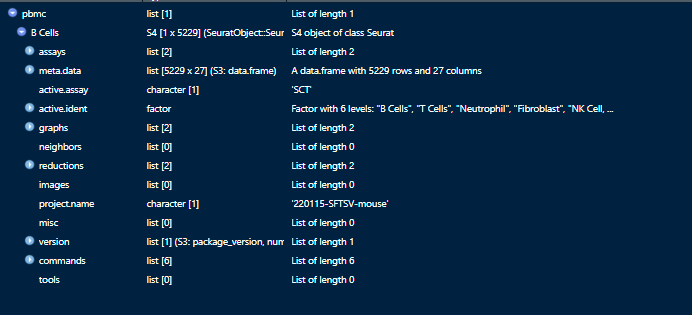
pbmc <- pbmc.data['B Cells']
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^mt-")这是我正在处理的数据的图片。

回答 1
Stack Overflow用户
发布于 2022-09-24 14:49:50
帮助您解决这个问题可能为时已晚,但是PBMC对象是list类型,而列表中的第一个元素(B单元格)是函数正在寻找的实际seurat对象。您需要从list对象中提取B单元对象并使用它。
类似于: b_cells <- pbmc11] b_cells["percent.mt"] <- PercentageFeatureSet(b_cells,pattern = "^mt-")
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/71938964
复制相关文章
相似问题

