克尼梅氨基酸筛选文库的构建
克尼梅氨基酸筛选文库的构建
提问于 2022-06-06 14:19:48
我有一个任务是建立四肽筛选文库氨基酸使用Knime。我以前从来没用过克尼莫。我需要创建一个包含所有20个氨基酸的工作流,将其与另外20个氨基酸相乘,然后将结果与另一个20个相乘,然后重复得到四肽的最终结果。有人能建议我如何输入氨基酸吗?非常感谢!
回答 1
Stack Overflow用户
发布于 2022-09-14 17:45:43
使用Table Creator节点输入氨基酸单字母代码,每个表一个.现在,使用一个Cross Joiner节点将表交叉连接到它自己--您现在应该有一个表,其行如下:
A|A
A|C
etc.现在,将此表放入第二个Cross Joiner节点的两个输入中,这将为您提供一个相当长的表,开始如下所示:
A|A|A|A
A|A|A|C
A|C|A|A
A|C|A|C
etc.现在使用一个Column Aggregator节点,选择所有列作为聚合列,聚合方法作为Concatenate,并将分隔符更改为空字符串:
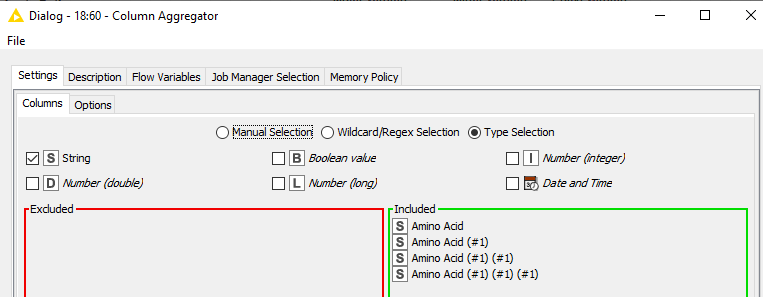
以及:
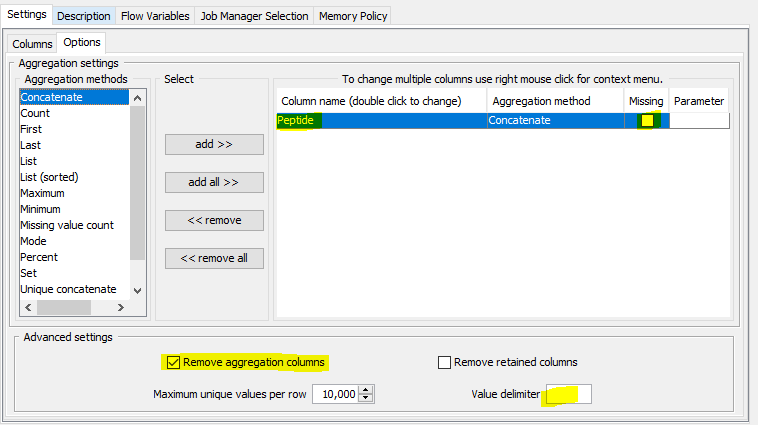
这会给你一张只有一列“肽”的桌子:
AAAA
AAAC
ACAA
ACAC
etc.如果您希望输出作为一个化学结构,那么从v1.36.0开始,Vernalis社区贡献包含一个节点Speedy Sequence to SMILES,该节点将将序列转换为一个微笑字符串(请确保选择输入列是蛋白质的选项!)
整个工作流程如下所示:
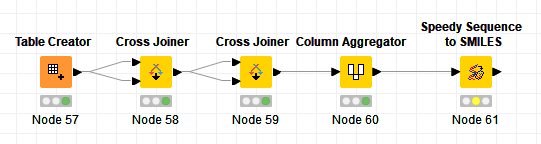
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/72519070
复制相关文章
相似问题

