Gseapy:如何获得用于每个途径的基因列表
Gseapy:如何获得用于每个途径的基因列表
提问于 2022-06-29 13:58:02
我正在用gseapy富集酶对一系列基因进行浓缩分析。我使用以下代码:
enr_res = gseapy.enrichr(gene_list = glist[:5000],
organism = 'Mouse',
gene_sets = ['GO_Biological_Process_2021'],
description = 'pathway',
#cutoff = 0.5
)结果如下:
enr_res.results.head(10)
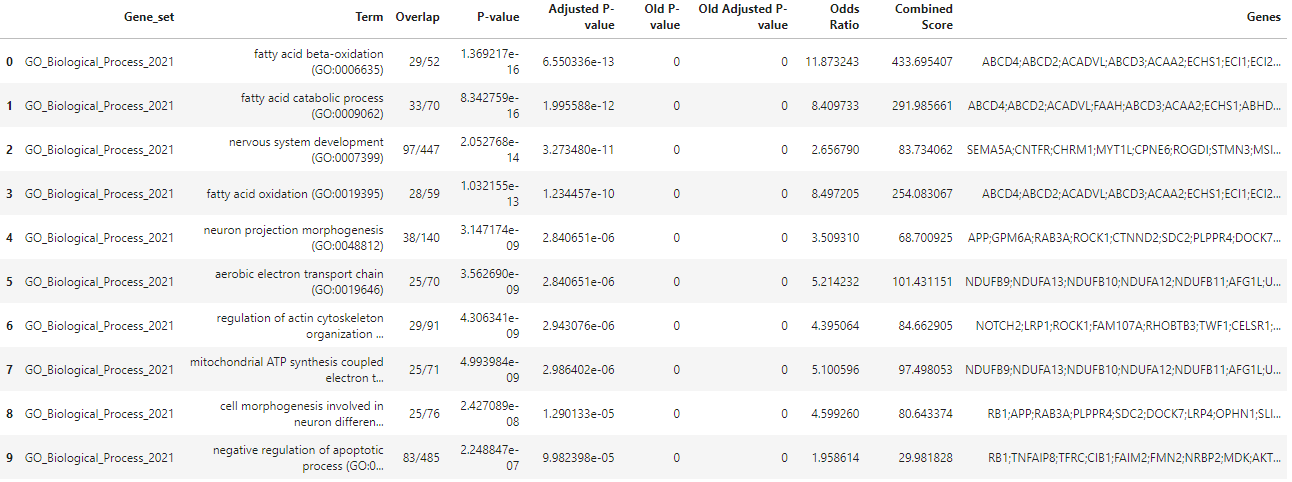
我的问题是,我如何获得完整的基因(在图片中非常正确的列)用于各个路径?
如果我尝试下面的代码,它只会给我已经显示的基因。我添加了一些更正,以有一个列表,然后我可以进一步使用的分析。
x = 'fatty acid beta-oxidation (GO:0006635)'
g_list = enr_res.results[enr_res.results.Term == x]['Genes'].to_string()
deliminator = ';'
g_list = [section + deliminator for section in g_list.split(deliminator) if section]
g_list = [s.replace(';', '') for s in g_list]
g_list = [s.replace(' ', '') for s in g_list]
g_list = [s.replace('.', '') for s in g_list]
first_gene = g_list[0:1]
first_gene = [sub[1 : ] for sub in first_gene]
g_list[0:1] = first_gene
for i in range(len(g_list)):
g_list[i] = g_list[i].lower()
for i in range(len(g_list)):
g_list[i] = g_list[i].capitalize()
g_list我认为我的方法可能是错误的,我得到所有的基因,我只是得到显示的基因。有人知道怎么才能得到所有的基因吗?
回答 1
Stack Overflow用户
回答已采纳
发布于 2022-07-07 07:53:28
pd.set_option('display.max_colwidth', 3000)这增加了显示字符的数量,并以某种方式解决了我的问题。:)
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/72802665
复制相关文章
相似问题

