是否有一个R函数将ICD10码的数据转换成它们各自的子章节
是否有一个R函数将ICD10码的数据转换成它们各自的子章节
提问于 2022-08-07 10:01:02
我有一个ICD10代码的数据,我需要转换到它们各自的子章节。这些代码的子章节是使用每个代码的前3个字符来识别的,即M1711的子章节是M17。
有没有一种有效的方法从这些代码映射到它们的子章节?
下面是我正在使用的代码的示例数据集:
df <- data.frame(codes = c("Z23","M1711","E0500","Z00129","G4452"))据我所知,JackO.Wasey有一个很好的包ICD,它可以转换为共混数据集,还有子章节数据集:
install.packages("devtools")
devtools::install_github("jackwasey/icd")
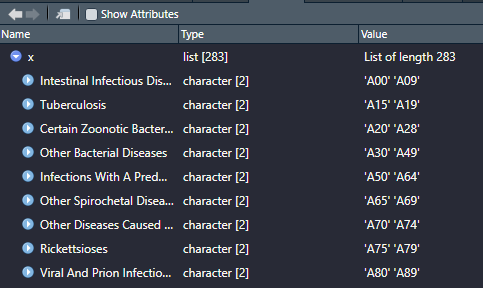
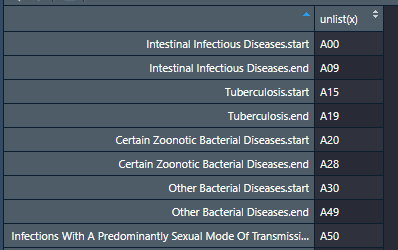
sub_chap <- icd::icd10_sub_chapters但正如您在下面所看到的,数据是在一个范围内的值,并没有以正确的格式‘加入’到。
当我取消列出子章节时,我在原始数据文件中的值之间缺少了值。
sub_chap_df = as.data.frame(unlist(sub_chap))
是否有一种有效的方法可以将我的ICD10代码转换成它们各自的子章节?
回答 1
Stack Overflow用户
回答已采纳
发布于 2022-08-07 13:08:14
您可以使用tidyr::complete()和tidyr::full_seq()来填充全部代码。您还需要将代码中的字母和数字部分分开使用full_seq(),然后将它们重新组合在一起。
注意:我没有安装icd软件包,所以我做了一些快速备份数据。
library(tidyverse)
# example data
sub_chap <- list(
cat1 = c(start = "A01", end = "A09"),
cat2 = c(start = "A15", end = "A19")
)
subchap_lookup <- tibble(
subchapter = names(sub_chap),
codes = sub_chap
) %>%
unnest_longer(codes, indices_include = FALSE) %>%
separate(codes, into = c("letter", "number"), sep = 1, convert = TRUE) %>%
group_by(subchapter, letter) %>%
complete(number = full_seq(number, 1)) %>%
ungroup() %>%
mutate(
codes = str_c(letter, str_pad(number, 2, pad = "0")),
.keep = "unused"
)输出:
# A tibble: 14 × 2
subchapter codes
<chr> <chr>
1 cat1 A01
2 cat1 A02
3 cat1 A03
4 cat1 A04
5 cat1 A05
6 cat1 A06
7 cat1 A07
8 cat1 A08
9 cat1 A09
10 cat2 A15
11 cat2 A16
12 cat2 A17
13 cat2 A18
14 cat2 A19 然后,您可以进行一个简单的左联接:
left_join(df, subchap_lookup)页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/73266528
复制相关文章
相似问题

