如何用等高线和平面图可视化GAM结果(使用ggplot2)
我想用ggplot2用gam结果绘制等高线图。以下是我想要的详细说明:
#packages
library(mgcv)
library(ggplot2)
library(tidyr)
#prepare data
df <- data.frame(x = iris$Sepal.Width,
y = iris$Sepal.Length,
z = iris$Petal.Length)
#fit gam
gam_fit <- gam(z ~
s(x) +
s(y),
data=df,na.action = "na.fail")为了根据gam_fit预测z值,我从https://drmowinckels.io/blog/2019-11-16-plotting-gamm-interactions-with-ggplot2/找到了一种方法
#predict z values
df_pred <- expand_grid(
x = seq(from=min(df$x),
to=max(df$x),
length.out = 100),
y = seq(from=min(df$y),
to=max(df$y),
length.out = 100)
)
df_pred <- predict(gam_fit, newdata = df_pred,
se.fit = TRUE) %>%
as_tibble() %>%
cbind(df_pred)
gg <- ggplot() +
geom_tile(data=df_pred, aes(x=x, y=y, fill = fit)) +
geom_point(data=df,aes(x=x, y=y))+
scale_fill_distiller(palette = "YlGnBu")+
geom_contour(data=df_pred, aes(x=x, y=y, z = fit), colour = "white")
print(gg)这给了我一个下面的情节
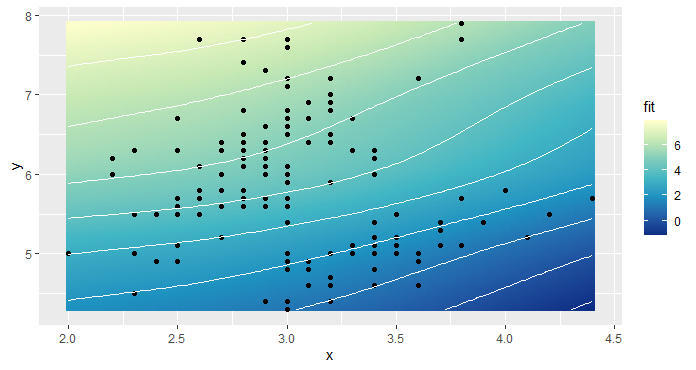
我的目标是去除瓷砖和轮廓,在那里没有测量的x-y点。例如,在图的右上角和左上角周围没有测量的点.
我想知道mgcViz是否能做到这一点,但它需要将x&y作为交互项,如下所示(我也不知道如何在下图中添加测量点):
library(mgcViz)
gamm_fit2 <- gam(z ~
s(x,y),
data=df,na.action = "na.fail") #,REML=TRUE
b <- getViz(gamm_fit2)
plot(sm(b, 1))
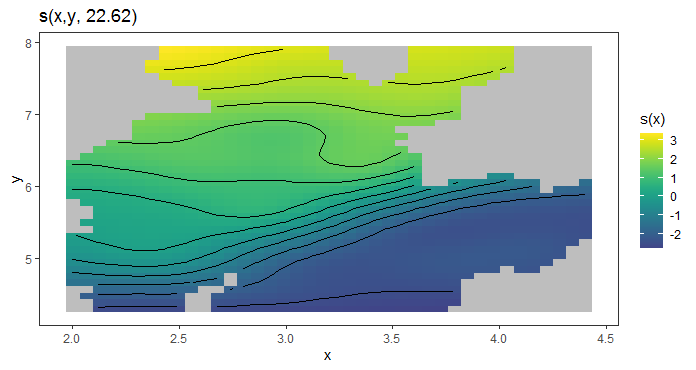
我认为df_pred可能不是实现我的目标的最佳格式,但我不知道如何做到这一点。如果您能给我任何ggplot2的解决方案,我将不胜感激。
回答 1
Stack Overflow用户
发布于 2022-09-16 08:39:56
要获得更类似于mgcv::plot.gam()和mgcViz如何生成类似这样的图的东西,您需要识别出与数据支持太远的对协变量。我们可能更倾向于这样做,比如,把预测剪裁到观测的凸壳上,原因是数据以外的一些轻微的外极化可能并不太违反样条只适用于数据范围的惩罚这一事实。从一个更实际的角度来看,这是在这个例子中使用的Anderson的Iris数据中所显示的,在协变量空间的一些区域,我们必须对这些区域进行插值,如果不是更远的话,数据的支持比我们可以推断的点更远。
mgcv具有一个名为exclude.too.far()的函数,因此如果您想要完全控制,可以重用@jared_mamrot的优秀答案(修改过一点)中的代码。
library("dplyr")
library("tidyr")
library("ggplot2")
library("mgcv")
# prepare data
df <- with(iris, data.frame(x = Sepal.Width,
y = Sepal.Length,
z = Petal.Length))
#fit gam
gam_fit <- gam(z ~ s(x) + s(y), data = df, method = "REML")
df_new <- with(df, expand_grid(x = seq(from = min(x), to = max(x),
length.out = 100),
y = seq(from = min(y), to = max(y),
length.out = 100)))
df_pred <- predict(gam_fit, newdata = df_new)
df_pred <- tibble(fitted = df_pred) |>
bind_cols(df_new)现在我们可以找出我们在网格中预测的哪些行表示协变量对,它们离原始数据的支持太远了。exclude.too.far()将预测网格中的协变量对转换为单位平方,0,0表示坐标(min(x),min(y)),1,1表示坐标(max(x),max(y))。它也将比原始协变量数据转换为这个单位平方。然后计算网格中的每个点(单位方格上)和观测数据中的每一行(投影到单位方)之间的欧几里德距离。
任何位于预测网格中节点的> dist的观察都会被识别为被排除在离数据支持太远的地方。dist是控制我们所说的“太远”的论点。dist是以单位平方来指定的,因此单位平方上的任意两点的最大值是
r$> dist(data.frame(x = c(0,1), y = c(0,1)))
1
2 1.414214plot.gam中的缺省值和mgcvViz中的IIRC都是dist = 0.1。如果我们这样做是为了我们的例子
drop <- exclude.too.far(df_pred$x, df_pred$y, df$x, df$y, dist = 0.1)drop现在是长度nrow(df_pred)的逻辑向量,TRUE表示应该排除观察对。
使用drop,我们可以将fitted设置为NA,用于我们想要排除的点:
df_pred <- df_pred |>
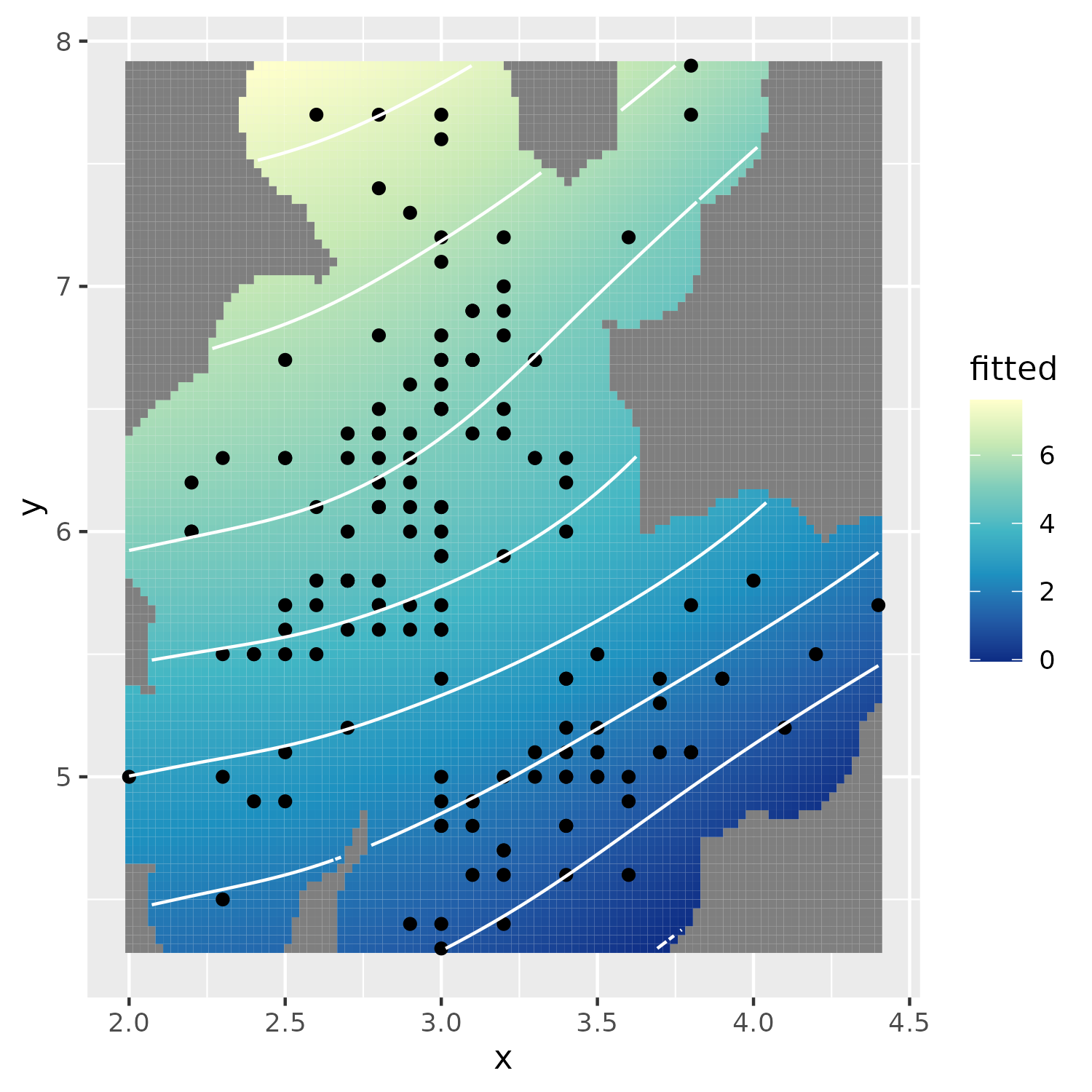
mutate(fitted = if_else(drop, NA_real_, fitted))现在我们可以阴谋:
df_pred |>
ggplot(aes(x = x, y = y, fill = fitted)) +
geom_tile() +
geom_point(data = df, aes(x = x, y = y, fill = NULL)) +
scale_fill_distiller(palette = "YlGnBu") +
geom_contour(aes(z = fitted, fill = NULL), colour = "white")生产
使用我的惠给金包(IMHO),您可以更容易地做到这一点,但总体思路是相同的。
# remotes::install_github("gavinsimpson/gratia") # need's dev version
library("gratia")
# prepare data
df <- with(iris, data.frame(x = Sepal.Width,
y = Sepal.Length,
z = Petal.Length))
# fit model
gam_fit <- gam(z ~ s(x) + s(y), data = df, method = "REML")
# prepare a data slice through the covariate space
ds <- data_slice(gam_fit, x = evenly(x, n = 100), y = evenly(y, n = 100))
# predict
fv <- fitted_values(gam_fit, data = ds)
# exclude points that are too far
drop <- too_far(ds$x, ds$y, df$x, df$y, dist = 0.1)
fv <- fv |>
mutate(fitted = if_else(drop, NA_real_, fitted))
# then plot
fv |>
ggplot(aes(x = x, y = y, fill = fitted)) +
geom_tile() +
geom_point(data = df, aes(x = x, y = y, fill = NULL)) +
scale_fill_distiller(palette = "YlGnBu") +
geom_contour(aes(z = fitted, fill = NULL), colour = "white")https://stackoverflow.com/questions/73738521
复制相似问题

