在python的元组列表中找到连续系列
我正在努力解决以下问题:我想编写一些小代码来去除同位素质量标准数据。
为此,我比较,如果两个信号之间的差等于一个质子的质量除以电荷态。到目前为止还很简单。
我现在挣扎着,找到了不止两座山峰。
我将问题分解为有一个元组列表,一个系列是n个元组,其中前一个元组的最后一个数等于当前元组的第一个元组。
由此:
(1,2),(2,3),(4,5),(7,9),(8,10),(9,11)
对此:
(1,2,3),(4,5),(7,9,11),(8,10)
简单的顺序将失败,因为可能有跳跃(7->9)和中间信号(8,10)。
以下是一些测试数据:
import numpy as np
proton = 1.0078250319
data = [
(632.3197631835938, 2244.3374), #0
(632.830322265625, 2938.797), #1
(634.3308715820312, 1567.1385), #2
(639.3309326171875, 80601.41), #3
(640.3339233398438, 23759.367), #4
(641.3328247070312, 4771.9946), #5
(641.8309326171875, 2735.902), #6
(642.3365478515625, 4600.567), #7
(642.84033203125, 1311.657), #8
(650.34521484375, 11952.237), #9
(650.5, 1), #10
(650.84228515625, 10757.939), #11
(651.350341796875, 6324.9023), #12
(651.8455200195312, 1398.8452), #13
(654.296875, 1695.3457)] #14
mz, i = zip(*data)
mz = np.array(mz)
i = np.array(i)
arr = np.triu(mz - mz[:, np.newaxis])
charge = 2实际上,在第一步,我只是对mz值感兴趣。我从所有值中减去所有值,并隔离上三角形。
为了计算,如果两个信号实际上在正确的质量范围内,那么我将使用以下代码:
>>> pairs = tuple(zip(*np.where(abs(arr - (proton / charge)) < 0.01)))
((0, 1), (5, 6), (6, 7), (7, 8), (9, 11), (11, 12), (12, 13))现在,对应的信号可以用眼睛看清楚:
峰值1: 0至1
高峰期2: 5至8
高峰3: 9到13,没有10。
所以原则上,我想把每个元组的第二个值与其他任何一个元组的第一个元组进行比较,以确定结果。
我试过的是,平放列表,删除重复,并在这个1D列表中找到相应的计数。但这是失败的,因为在5-9的峰值被发现。
我想要一个矢量化的解,因为这个计算是为100-500个信号在30000+光谱中的多个电荷态。
我很肯定,这以前曾有人问过,但未能找到合适的解决办法。
最终,这些序列被用来检查相应峰的强度,并将它们加和,并使用最大初始值在此指定去同位素峰。
泰克斯基督教
ps。此外,如果有一些建议,已经存在的代码,我很高兴学习。我对矢量化计算非常陌生,通常会编写大量的for循环,这些循环需要很长时间才能完成。
回答 4
Stack Overflow用户
发布于 2022-11-22 15:31:16
尝试:
d = {}
pairs = [(1,2), (2,3), (4,5), (7,9), (8,10), (9,11)]
for t in pairs:
if t[0] in d:
d[t[0]].append(t[1])
d[t[1]] = d.pop(t[0])
else:
d[t[1]] = list(t)
signals = tuple(tuple(v) for v in d.values())虽然解不是矢量化的,但它将是O(n)时间。请注意,此解决方案只有在对进行排序时才有效。
Stack Overflow用户
发布于 2022-11-22 15:43:27
在图论中,您的问题将是“如何在图中找到所有不连通子图?”。
那么,为什么不使用网络分析库(如networkx )呢?
import networkx as nx
# Your tuples become the edges of the graph.
edge = [(1,2), (2,3), (4,5), (7,9), (8,10), (9,11)]
# We create the graph
G = nx.Graph()
G.add_edges_from(edge)
# Use connected_components to detect the subgraphs.
d = list(nx.connected_components(G)) 我们得到了如下预期:
[{1, 2, 3}, {4, 5}, {7, 9, 11}, {8, 10}]有四个子图:
Stack Overflow用户
发布于 2022-11-23 10:20:54
谢谢你的回复。
我非常喜欢networkx方法,但有趣的是,这比O(n)循环解决方案慢。我用我的原始数据和随机训练集对两者进行了基准测试。在这两种情况下,结果是相等的(这是一个先决条件),但是O(n)解的速度要快4到10倍。
这是基准
networkx
size: 10
22.9 µs ± 1.92 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 100
165 µs ± 4.59 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 1000
2.08 ms ± 52.4 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 10000
23.1 ms ± 499 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 100000
350 ms ± 6.12 ms per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 1000000
4.82 s ± 120 ms per loop (mean ± std. dev. of 2 runs, 10 loops each)
loop
size: 10
3.31 µs ± 418 ns per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 100
35.3 µs ± 5.68 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 1000
350 µs ± 22.2 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 10000
3.95 ms ± 38.5 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 100000
71.6 ms ± 278 µs per loop (mean ± std. dev. of 2 runs, 10 loops each)
size: 1000000
1.11 s ± 30.8 ms per loop (mean ± std. dev. of 2 runs, 10 loops each)
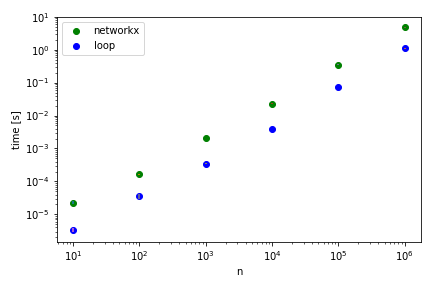
我认为在非常大的样本大小下,NetworkX可能表现得更好,但在我的场景中,小样本(< 1000个初始信号),但通常情况下,循环解决方案获胜。
这里是要复制的代码。
import numpy as np
import networkx as nx
def p1(edge):
# from https://stackoverflow.com/a/74535322/14571316
G = nx.Graph()
G.add_edges_from(edge)
# Use connected_components to detect the subgraphs.
d = list(nx.connected_components(G))
return d
def p2(pairs):
# from https://stackoverflow.com/a/74535164/14571316
d = {}
for t in pairs:
if t[0] in d:
d[t[0]].append(t[1])
d[t[1]] = d.pop(t[0])
else:
d[t[1]] = list(t)
signals = tuple(tuple(v) for v in d.values())
return signals
print("networkx")
for size in [10, 100, 1000, 10000, 100000, 1000000]:
print(f'size: {size}')
l1 = np.random.randint(0, high=int(size/2), size=size)
l2 = np.random.randint(0, high=int(size/2), size=size)
pairs = tuple(zip(l1, l2))
%timeit -n10 -r2 p1(pairs)
print("loop")
for size in [10, 100, 1000, 10000, 100000, 1000000]:
print(f'size: {size}')
l1 = np.random.randint(0, high=int(size/2), size=size)
l2 = np.random.randint(0, high=int(size/2), size=size)
pairs = tuple(zip(l1, l2))
%timeit -n10 -r2 p2(pairs)https://stackoverflow.com/questions/74534622
复制相似问题

