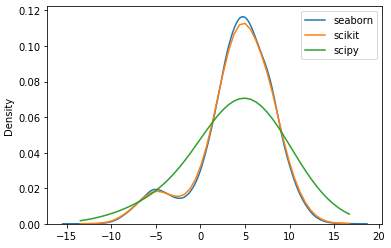
利用gaussian_kde和sklearn的KernelDensity进行核密度估计得到了不同的结果。
我从两个叠加的正态分布中创建了一些数据,然后应用sklearn.neighbors.KernelDensity和scipy.stats.gaussian_kde来估计密度函数。但是,使用相同的带宽(1.0)和相同的内核,两种方法产生不同的结果。有人能解释一下为什么吗?谢谢你帮忙。
下面可以找到重现问题的代码:
import matplotlib.pyplot as plt
import numpy as np
from scipy.stats import gaussian_kde
import seaborn as sns
from sklearn.neighbors import KernelDensity
n = 10000
dist_frac = 0.1
x1 = np.random.normal(-5,2,int(n*dist_frac))
x2 = np.random.normal(5,3,int(n*(1-dist_frac)))
x = np.concatenate((x1,x2))
np.random.shuffle(x)
eval_points = np.linspace(np.min(x), np.max(x))
kde_sk = KernelDensity(bandwidth=1.0, kernel='gaussian')
kde_sk.fit(x.reshape([-1,1]))
y_sk = np.exp(kde_sk.score_samples(eval_points.reshape(-1,1)))
kde_sp = gaussian_kde(x, bw_method=1.0)
y_sp = kde_sp.pdf(eval_points)
sns.kdeplot(x)
plt.plot(eval_points, y_sk)
plt.plot(eval_points, y_sp)
plt.legend(['seaborn','scikit','scipy'])
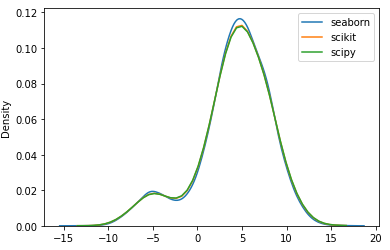
如果将枕骨带改为0.25,两种方法的结果大致相同。
回答 3
Stack Overflow用户
发布于 2021-07-16 11:08:09
在scipy.stats.gaussian_kde和sklearn.neighbors.KernelDensity中,带宽的含义是不同的。Scipy.stats.gaussian_kde使用带宽因子https://docs.scipy.org/doc/scipy/reference/generated/scipy.stats.gaussian_kde.html。对于一维核密度估计,应用了以下公式:
sklearn.neighbors.KernelDensity的带宽= scipy.stats.gaussian_kde *标准差的带宽因子
对于您的估计,这可能意味着您的标准偏差等于4。
有关更多信息,我想参考Getting bandwidth used by SciPy's gaussian_kde function。
Stack Overflow用户
发布于 2022-11-07 15:28:48
老实说,我不知道为什么,但使用the超参数bw_method='scott'使其工作与海运完全一样。
所以,这似乎都是关于超参数的。我们可以通过深入了解它们来找出原因,但与此同时,我们只需使用“scott”或“silverman”,而不是使用随机标量。
import matplotlib.pyplot as plt
import numpy as np
from scipy.stats import gaussian_kde
import seaborn as sns
from sklearn.neighbors import KernelDensity
n = 10000
dist_frac = 0.1
x1 = np.random.normal(-5,2,int(n*dist_frac))
x2 = np.random.normal(5,3,int(n*(1-dist_frac)))
x = np.concatenate((x1,x2))
np.random.shuffle(x)
eval_points = np.linspace(np.min(x), np.max(x))
kde_sk = KernelDensity(bandwidth=1, kernel='gaussian')
kde_sk.fit(x.reshape([-1,1]))
y_sk = np.exp(kde_sk.score_samples(eval_points.reshape(-1,1)))
kde_sp = gaussian_kde(x, bw_method='scott') ### I MEAN HERE! ###
y_sp = kde_sp.pdf(eval_points)
sns.kdeplot(x)
plt.plot(eval_points, y_sk)
plt.plot(eval_points, y_sp)
plt.legend(['seaborn','scikit','scipy'])Stack Overflow用户
发布于 2022-01-18 19:52:35
增加“随机正常”的大小。你的数据点太少了。尝试使用n=500000并检查结果。
https://stackoverflow.com/questions/68396403
复制相似问题

