R- stat_compare_means回归差值与Kruskal-Wallis检验
R- stat_compare_means回归差值与Kruskal-Wallis检验
提问于 2019-12-26 22:17:04
我想使用包ggplot中的R函数stat_compare_means,将Kruskal测试的p值绘制到我的ggpubr。
但是,如果只运行函数,则绘制的值与值不同:
kruskal.test(value ~ type, data = Profile_melt)
我绘制p值的代码是:
ggplot(Profile_melt, aes(type, value)) +
geom_boxplot(aes(fill = factor(type), alpha = 0.5),
outlier.shape = NA, show.legend = FALSE) +
geom_jitter(width = 0.2, size = 2, show.legend = FALSE,
aes(colour = factor(type)), alpha = 0.5) +
theme_bw() +
facet_grid(Case ~ Marker, scales = 'free') +
stat_compare_means(comparison = list(c("Real", "Binomial")),method = 'kruskal.test')+
background_grid(major = 'y', minor = "none") + # add thin horizontal lines
xlab('Category') +
ylab('Cell counts (Frequencies)')+
theme(axis.text = element_text(size = 15),
axis.title = element_text(size = 20),
legend.text = element_text(size = 38),
legend.title = element_text(size = 30),
strip.background = element_rect(colour="black", fill="white"),
strip.text = element_text(margin = margin(10, 10, 10, 10), size = 25)) +
panel_border()这是我的数据样本数据
回答 2
Stack Overflow用户
回答已采纳
发布于 2019-12-26 23:29:53
有许多代码行可能与这个问题无关。也许,你的问题是:
为什么
kruskal.test(value ~ type, data = Profile_melt)
#Kruskal-Wallis chi-squared = 4.9673, df = 1, p-value = 0.02583生成不同的p值。
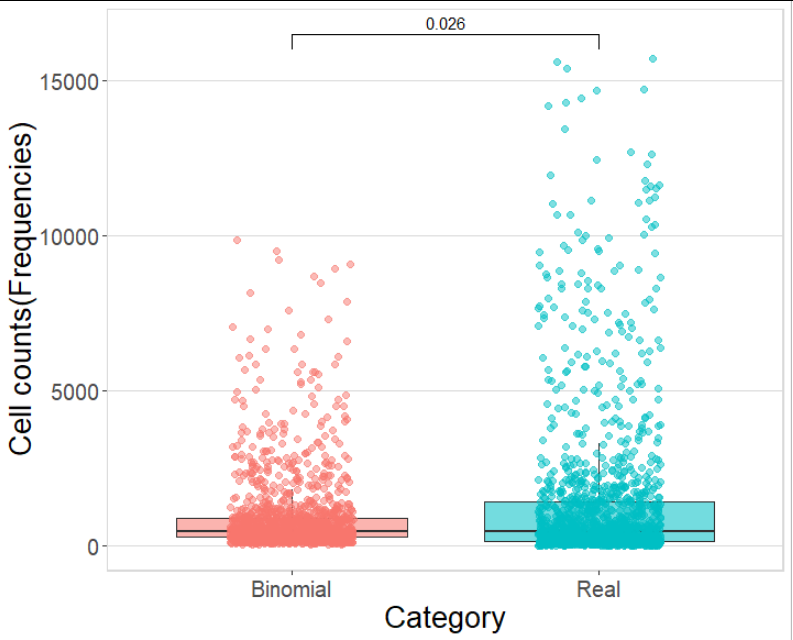
ggboxplot(Profile_melt, x="type", y = "value") +
stat_compare_means(comparison = list(c("Real", "Binomial")), method = 'kruskal.test')
# p-value = 0.49您可以通过检查原始代码来找出原因。ggpubr的开发人员可能会更好地解释这一点,如果它是一个问题,可能会在那里修复它。若要获得正确和一致的p值,请删除comparison = list(c("Real", "Binomial"))
ggboxplot(Profile_melt, x="type", y = "value") +
stat_compare_means(method = 'kruskal.test')或
编辑
ggboxplot(Profile_melt, x="type", y = "value") +
stat_compare_means(comparison = list(c("Real", "Binomial")))对于其他代码,图如下所示:
Stack Overflow用户
发布于 2019-12-27 09:17:40
来自ggpubr的stat_compare_means调用compare_means,默认情况下使用wilcox.test。因此,正如@芝强王所指出的,如果删除该方法或比较,它将进入与最初得到的p值类似的缺省值,因为wilcoxon和kruskal对于2个样本非常相似:
kruskal.test(value ~ type, data = Profile_melt)
#Kruskal-Wallis chi-squared = 4.9673, df = 1, p-value = 0.02583
wilcox.test(value ~ type, data = Profile_melt)
#W = 1034939, p-value = 0.02583现在,对于您拥有的数据,您很可能希望每个单独的大小写和标记都有一个p值,而不是使用kruskal.test(value ~ type, data = Profile_melt)进行pan比较。为所有方面打印相同的p值是没有意义的。
我们首先检查所需的p值:
compare_means(value ~ type, Profile_melt, group.by = c("Case","Marker"),
method="kruskal")
# A tibble: 30 x 8
Case Marker .y. p p.adj p.format p.signif method
<fct> <fct> <chr> <dbl> <dbl> <chr> <chr> <chr>
1 Case 1A CD3 value 0.000470 0.0085 0.00047 *** Kruskal-Wallis
2 Case 1A CD4 value 0.00000915 0.00022 9.2e-06 **** Kruskal-Wallis
3 Case 1A CD8 value 0.00695 0.09 0.00695 ** Kruskal-Wallis
4 Case 1A CD20 value 0.707 1 0.70724 ns Kruskal-Wallis
5 Case 1A FoxP3 value 0.00102 0.014 0.00102 ** Kruskal-Wallis
6 Case 1B CD3 value 0.0000415 0.00091 4.1e-05 **** Kruskal-Wallis它类似于:
Profile_melt %>%
group_by(Case,Marker) %>%
summarize(k_p=kruskal.test(value ~ type)$p.value)
# A tibble: 30 x 3
# Groups: Case [6]
Case Marker k_p
<fct> <fct> <dbl>
1 Case 1A CD3 0.000470
2 Case 1A CD4 0.00000915
3 Case 1A CD8 0.00695
4 Case 1A CD20 0.707
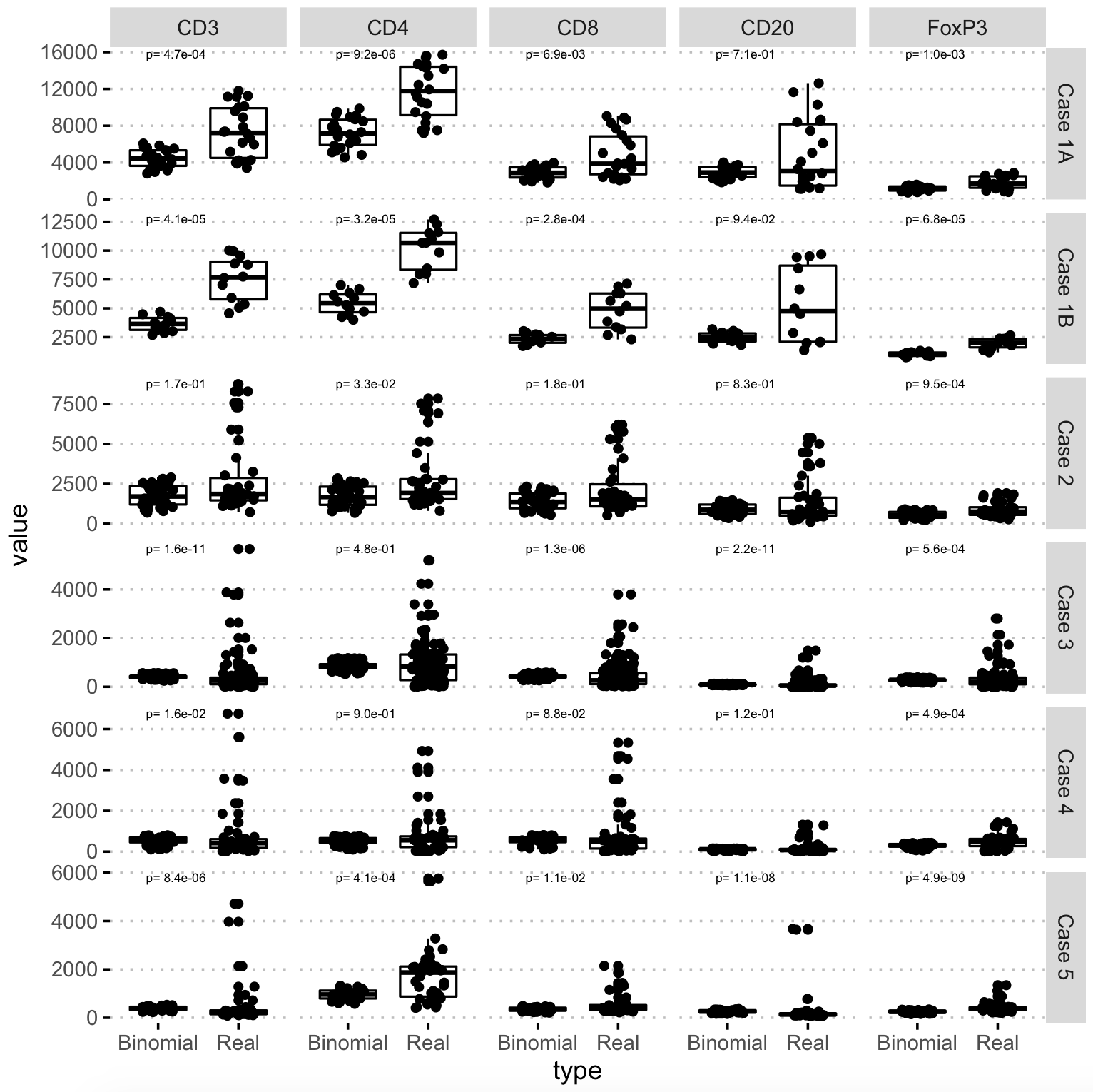
5 Case 1A FoxP3 0.00102 我们可以绘制,必须更容易地使用ggpubr包中的ggpubr绘图:
p = ggboxplot(Profile_melt,x="type",y="value",add="jitter",
facet.by=c("Case","Marker"),scales="free_y",ggtheme=theme_pubclean())
p+stat_compare_means(
aes(label =paste("p=",scientific(as.numeric(..p.format..)))),
method="kruskal",size=2)
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/59494025
复制相关文章
相似问题

