为什么我的ROC图和AUC值看起来很好,而我来自随机森林的混淆矩阵显示该模型不能很好地预测疾病?
我正在使用R中的包randomForest创建一个模型,将病例分为疾病(1)或无疾病(0):
classify_BV_100t <- randomForest(bv.disease~., data=RF_input_BV_clean, ntree = 100, localImp = TRUE)
print(classify_BV_100t)
Call:
randomForest(formula = bv.disease ~ ., data = RF_input_BV_clean, ntree = 100, localImp = TRUE)
Type of random forest: classification
Number of trees: 100
No. of variables tried at each split: 53
OOB estimate of error rate: 8.04%
Confusion matrix:
0 1 class.error
0 510 7 0.01353965
1 39 16 0.70909091我的混淆矩阵表明,该模型对0(无疾病)进行了很好的分类,但对1(疾病)的分类效果很差。
但是当我绘制ROC图的时候,它给人的印象是这个模型很好。
以下是我绘制中华民国的两种不同方式:
- (使用https://stats.stackexchange.com/questions/188616/how-can-we-calculate-roc-auc-for-classification-algorithm-such-as-random-forest) 库(PROC) rf.roc<-roc(RF_input_BV_clean$bv.disease,classify_BV_100t$votes,2)图(rf.roc) auc(rf.roc)
- (使用如何在R中使用插入符号计算ROC和ROC下的AUC?) 库(ROCR)预测<- as.vector(classify_BV_100t$votes,2) <-预测(预测,RF_input_BV_clean$bv.disease) perf_AUC <- performance( pred," AUC ") #计算AUC值<- perf_AUC@y.values[1] perf_ROC <- performance(pred,"tpr","fpr") #绘制实际的ROC曲线图(perf_ROC,main="ROC图“)文本(0.5,0.5,粘贴(”AUC= ",format(AUC,digits=5 ),(scientific=FALSE))
这些是中华民国第一和第二章的情节:
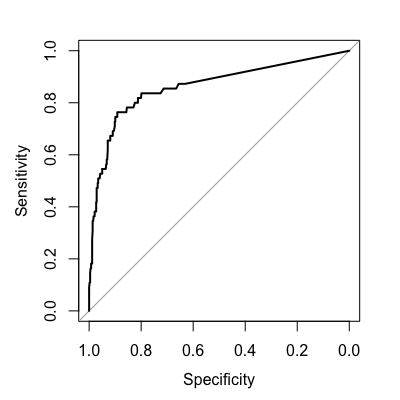
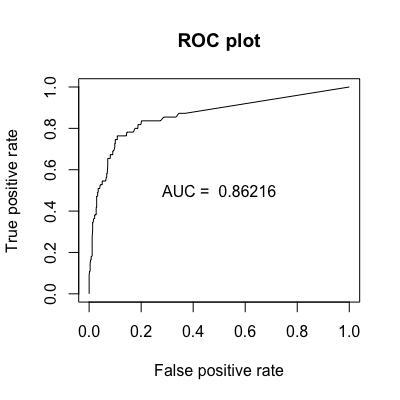
这两种方法给我的AUC为0.8621593。
有没有人知道为什么随机森林混淆矩阵的结果似乎与中华民国不相符?
回答 2
Stack Overflow用户
发布于 2019-11-20 16:53:10
我不认为你的图谋有什么问题,你对这一差异的评估是正确的。
高的AUC值是一个很高的真实负率的产物。我国考虑的是敏感性,主要是衡量真实的正值和特异性,衡量的是真正的负值。因为您的特异性非常高,该度量有效地携带了较低的模型灵敏度值,这使您的AUC相对较高。是的,这是一个很高的AUC,但正如你所提到的,这个模型只擅长预测负面影响。
我建议计算额外的指标(敏感性,特异性,真阳性率,假阳性率.)并在评估模型时评估所有这些指标的组合。AUC是一个质量度量,但有了额外的度量,它就意味着更多。
Stack Overflow用户
发布于 2019-11-20 18:07:05
为了增加@DanCarver的答案,您还可以更改预测结果的截止概率,如0或1。默认情况下,对于一个两类问题,randomForest中的概率阈值都是0.5 .但是,如果假阴性(对0的错误预测)比假阳性(以及对1的错误预测)代价更高,您可以使用较低的截止概率来预测类1。
下面是一个使用BreastCancer数据的示例:
library(randomForest)
library(mlbench)
data(BreastCancer)
library(caret)
# Limit data frame to complete cases
d = BreastCancer[complete.cases(BreastCancer),]
# Run random forest model
set.seed(10)
m1 = randomForest(Class ~ Bare.nuclei + Marg.adhesion, data=d)
m1
# Generate data frame of predictions
pred = data.frame(predict(m1, type="prob"),
actual=d$Class,
thresh0.5=predict(m1))
# Add prediction if we set probability threshold of 0.3 (instead of 0.5)
# for classifying a prediction as "malignant"
pred$thresh0.3 = factor(ifelse(pred$malignant > 0.3, "malignant", "benign"))
# Look at confusion matrix for each probability threshold
confusionMatrix(pred$thresh0.5, pred$actual)
confusionMatrix(pred$thresh0.3, pred$actual)下面是confusionMatrix函数输出的一部分。注意,在阈值较低的情况下,我们捕获更多的真阳性(220而不是214),但代价是获得更多的假阳性(28而不是20)。如果假阴性比假阳性代价更高的话,这可能是一个很好的权衡。这篇文章讨论了优化randomForest模型以优化概率阈值的问题。
阈值概率0.5预测恶性
Reference Prediction benign malignant benign 424 25 malignant 20 214
阈值预测恶性的概率0.3
Reference Prediction benign malignant benign 416 19 malignant 28 220
https://stackoverflow.com/questions/58959104
复制相似问题
