如何对齐峰值标签?
我试图用R中的峰值来绘制数据,图形本身进展良好,但我遇到了标记相关峰的问题。我目前的标签制度(详见下文)奇怪地将峰值标签移向一边,导致直线相交。是否有一种方法使标签与峰本身对齐,或以其他方式将它们以美观的方式排列?
下面的代码使用这些数据再现我的问题。
library(ggplot2)
library(ggpmisc)
library(ggrepel)
x=read.csv("data.csv")
colnames(x)=c("wv", "abs")
ggplot(x, aes(x=wv, y=abs)) + geom_line() + xlab(bquote('Wavenumbers ('~cm^-1*')')) + ylab("Absorbance (A.U.)") + scale_x_reverse(limits=c(2275,1975), expand=c(0,0)) + ylim(-0.01,0.29) + stat_peaks(colour = "black", span = 11, geom ="text_repel", direction = "y", angle = 90, ignore_threshold = 0.09, size = 3, x.label.fmt = "%.2f", vjust = 1, hjust = 0, segment.color = "red") + ggtitle("FTIR - Carbon Monoxide, Fundamentals")
回答 3
Stack Overflow用户
发布于 2021-05-05 02:10:14
下面是一种使用ggplot2::stage以及内置的segment和text几何学的方法:
library(ggplot2)
library(ggpmisc)
id <- "1S345TaPqANriDPLN6H_PyunuM1QHi485"
x <- read.csv(sprintf("https://docs.google.com/uc?id=%s&export=download", id))
colnames(x)=c("wv", "abs")
ggplot(x, aes(x=wv, y=abs)) + geom_line() +
xlab(bquote('Wavenumbers ('~cm^-1*')')) + ylab("Absorbance (A.U.)") + ggtitle("FTIR - Carbon Monoxide, Fundamentals") + ylim(-0.01,0.29) +
stat_peaks(mapping = aes(x = stage(wv, after_scale = x + 0.25)),
geom ="text", colour = "black", span = 11, angle = 90,
ignore_threshold = 0.09, size = 2.5, x.label.fmt = "%.2f",
vjust = 0.5, hjust = -1) +
stat_peaks(mapping = aes(xend = stage(wv, after_scale = x + 0.25),
y = stage(abs, after_stat = y + 0.005),
yend = after_stat(y + 0.02)),
geom ="segment", lwd = 0.5, colour = "red",
span = 11, ignore_threshold = 0.09) +
scale_x_reverse(expand=c(0,0), limits = c(2275,1975))
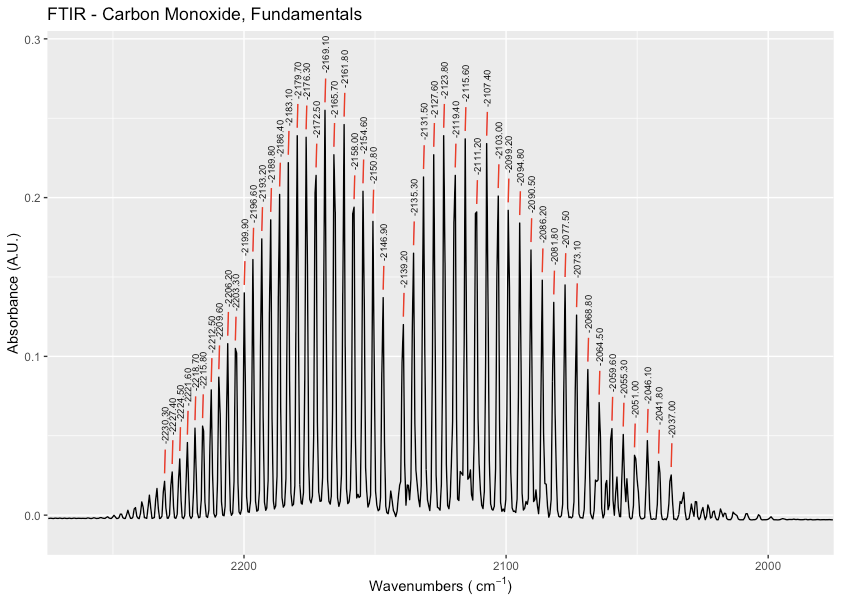
从help(stat_peaks)中我们可以看到,x和y统计量是为每个峰值计算的。通常,我们可以使用after_stat访问这些统计数据,但是由于您也转换了x轴,所以需要在缩放之后实际访问该统计数据。
在stat_peaks的第一层中,我们使用stage(wv, after_scale = x + 0.25)向x坐标添加一点提示。在stat_peaks的第二次调用中,我们将几何设置为segment。段需要一个xend和yend。我们可以使用stage和after_stat再次添加一个提示和行长度。
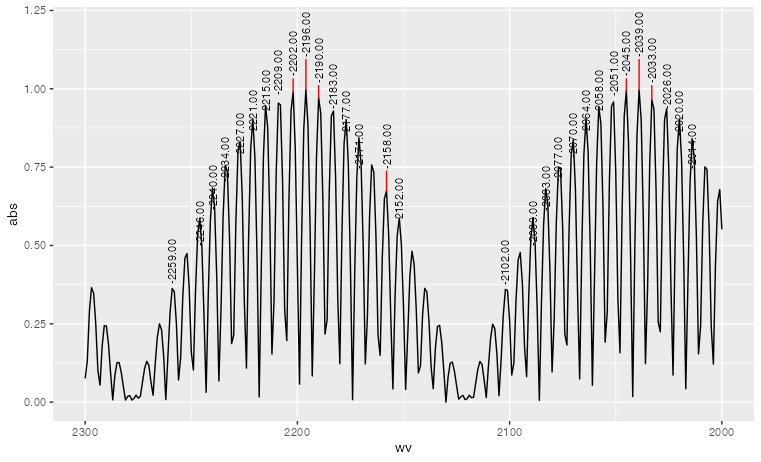
Stack Overflow用户
发布于 2021-05-04 23:20:24
hjust = 0.5应该更好地工作。使用hjust = 0可以使标签稍微向右对齐,文本的顶部边缘与每个峰值的中间对齐。
这里有一个可重复的例子,它不依赖于外部数据,这些数据可能在该链接中不可用。(关于OP数据集的应用程序,请参见底部。)
library(ggpmisc)
library(ggrepel)
library(ggplot2)
x <- data.frame(wv = 2300:2000)
x$abs = abs(cos(x$wv/50) * sin(x$wv/2))
ggplot(x, aes(wv, abs)) +
geom_line() +
stat_peaks(colour = "black", span = 11,
geom ="text_repel", direction = "y",
angle = 90, ignore_threshold = 0.09,
size = 3, x.label.fmt = "%.2f",
vjust = 1, hjust = 0.5, segment.color = "red") +
scale_x_reverse(limits = c(2300,2000))
下面是加载原始数据:
library(readr)
x <- read_csv("~/Downloads/CO-FTIR Spectrum-1800 mTorr-2021.csv")
colnames(x)=c("wv", "abs")这里添加了这些参数:
box.padding = 0.0, nudge_y = 0.02,
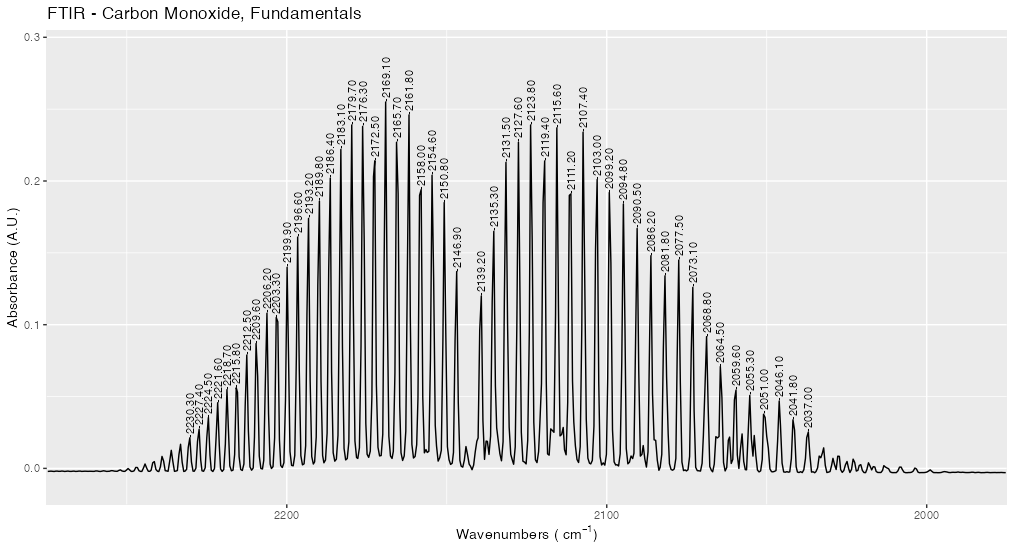
Stack Overflow用户
发布于 2021-05-05 00:29:56
在不构造辅助数据集的情况下,我无法找到这样做的方法。然而,一旦构建了实际的绘图,就会变得相当容易:
守则如下:
library(dplyr)
library(ggplot2)
library(ggpmisc)
library(ggrepel)
library(pracma)
#####################################
# Constants
#####################################
wvfilter = c(1975, 2275)
data_filename = "data.csv"
#####################################
# Read data
#####################################
dat = read.csv(data_filename)
colnames(dat)=c("wv", "abs")
#####################################
# Identify peaks
#####################################
# Extract peaks
peaks = data.frame(findpeaks(dat$abs, threshold=0, minpeakheight=0.03))
# Give data frame reasonable names
colnames(peaks) = c("height", "x_index", "peak_begin_index", "peak_end_index")
# Convert from index to wavelength
peaks$wv = dat$wv[peaks$x_index]
# Set the y position of the labels
peaks$nudge = 0.3-peaks$height
# Filter by wavelength
peaks = peaks %>% filter(wvfilter[1] <= wv & wv <= wvfilter[2])
#####################################
# Plot data
#####################################
ggplot(x, aes(x=wv, y=abs)) +
geom_line() +
geom_text_repel(
data=peaks,
mapping=aes(x=wv, y=height, label=height),
force=0,
nudge_y=peaks$nudge,
direction="x",
angle=90,
segment.color="red"
) +
xlab(bquote('Wavenumbers ('~cm^-1*')')) +
ylab("Absorbance (A.U.)") +
scale_x_reverse(limits=rev(wvfilter), expand=c(0,0)) +
ylim(-0.01,0.29) +
ggtitle("FTIR - Carbon Monoxide, Fundamentals")这给出了情节:
一些注意事项:
- 在图像中,我对标签使用了错误的文本。用
label=wv而不是label=height修复这个问题。 - 需要使用传递给
peaks的相同值对scale_x_reverse(limits=数据集进行筛选。为了简化这个问题,我引入了一个全局变量来设置x轴限制。 - 使用
force参数可以对标签进行不同程度的聚类。force=0似乎对您的数据最有效。 - 您需要手动设置
peaks$nudge值。除了猜测和检查之外,我不知道有什么好办法。
https://stackoverflow.com/questions/67393067
复制相似问题

