如何在matplotlib中用密度来绘制散点图?
我想做一个散点图,每个点都用附近点的空间密度来着色。
我遇到了一个非常类似的问题,它展示了一个使用R的例子:
在python中使用matplotlib实现类似事情的最佳方法是什么?
回答 4
Stack Overflow用户
发布于 2013-11-20 21:39:37
除了@askewchan建议的hist2d或hexbin之外,您还可以使用与链接到的问题中接受的答案相同的方法。
如果你想这么做:
import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import gaussian_kde
# Generate fake data
x = np.random.normal(size=1000)
y = x * 3 + np.random.normal(size=1000)
# Calculate the point density
xy = np.vstack([x,y])
z = gaussian_kde(xy)(xy)
fig, ax = plt.subplots()
ax.scatter(x, y, c=z, s=100)
plt.show()
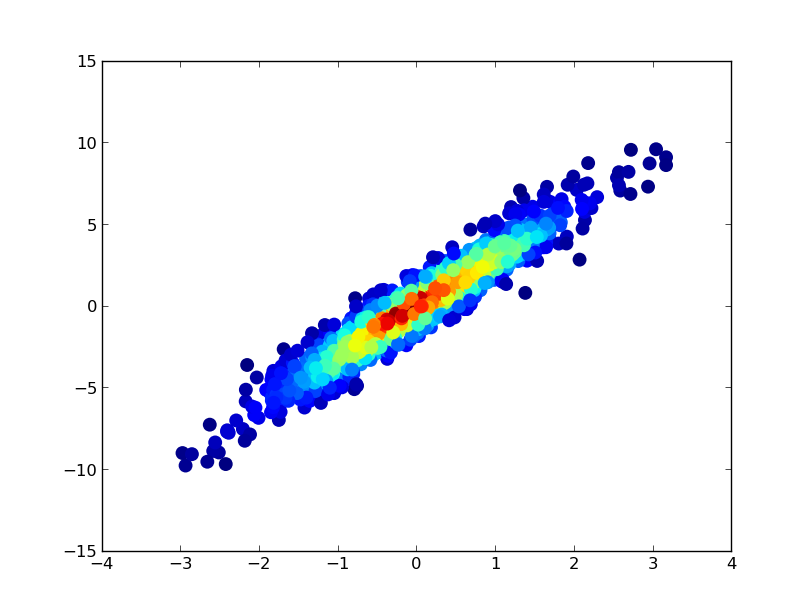
如果您希望按密度顺序绘制点,使最密集的点始终位于顶部(类似于链接的示例),只需按z值对它们进行排序。我还将在这里使用一个较小的标记大小,因为它看起来更好一些:
import numpy as np
import matplotlib.pyplot as plt
from scipy.stats import gaussian_kde
# Generate fake data
x = np.random.normal(size=1000)
y = x * 3 + np.random.normal(size=1000)
# Calculate the point density
xy = np.vstack([x,y])
z = gaussian_kde(xy)(xy)
# Sort the points by density, so that the densest points are plotted last
idx = z.argsort()
x, y, z = x[idx], y[idx], z[idx]
fig, ax = plt.subplots()
ax.scatter(x, y, c=z, s=50)
plt.show()
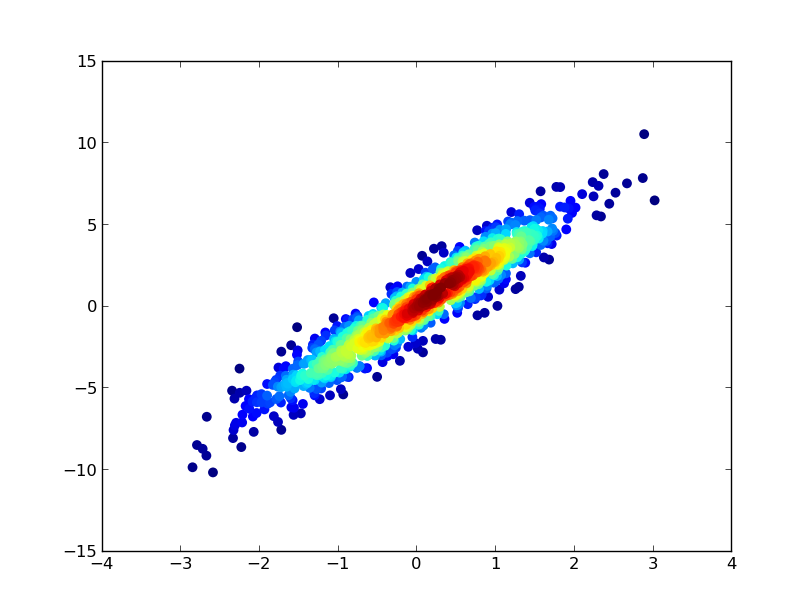
Stack Overflow用户
发布于 2018-12-20 09:28:24
此外,如果点数使KDE计算太慢,则可以在np.histogram2d更新中对注释进行内插:如果希望显示色条,请使用plt.scatter()而不是ax.scatter(),后面跟着plt.colorbar()
import numpy as np
import matplotlib.pyplot as plt
from matplotlib import cm
from matplotlib.colors import Normalize
from scipy.interpolate import interpn
def density_scatter( x , y, ax = None, sort = True, bins = 20, **kwargs ) :
"""
Scatter plot colored by 2d histogram
"""
if ax is None :
fig , ax = plt.subplots()
data , x_e, y_e = np.histogram2d( x, y, bins = bins, density = True )
z = interpn( ( 0.5*(x_e[1:] + x_e[:-1]) , 0.5*(y_e[1:]+y_e[:-1]) ) , data , np.vstack([x,y]).T , method = "splinef2d", bounds_error = False)
#To be sure to plot all data
z[np.where(np.isnan(z))] = 0.0
# Sort the points by density, so that the densest points are plotted last
if sort :
idx = z.argsort()
x, y, z = x[idx], y[idx], z[idx]
ax.scatter( x, y, c=z, **kwargs )
norm = Normalize(vmin = np.min(z), vmax = np.max(z))
cbar = fig.colorbar(cm.ScalarMappable(norm = norm), ax=ax)
cbar.ax.set_ylabel('Density')
return ax
if "__main__" == __name__ :
x = np.random.normal(size=100000)
y = x * 3 + np.random.normal(size=100000)
density_scatter( x, y, bins = [30,30] )
Stack Overflow用户
发布于 2020-09-28 15:37:18
绘图>100 K数据点?
使用接受答案,使用kde()需要很长时间。在我的机器上,100 K行花费了大约11分钟。在这里,我将添加两个替代方法(mpl-散射密度和数据哈德),并将给定的答案与相同的数据集进行比较。
在下面,我使用了一个100 k行的测试数据集:
import matplotlib.pyplot as plt
import numpy as np
# Fake data for testing
x = np.random.normal(size=100000)
y = x * 3 + np.random.normal(size=100000)输出与计算时间比较
下面是不同方法的比较。
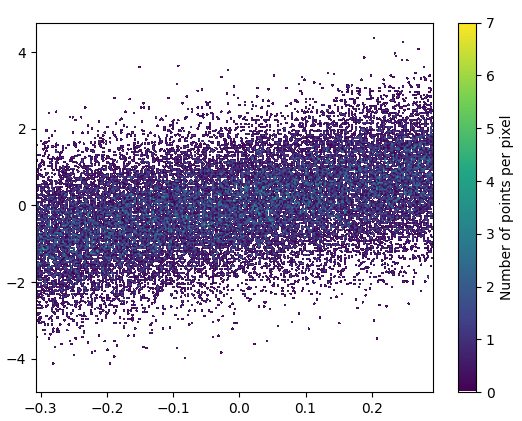
1: mpl-scatter-density
安装
pip install mpl-scatter-density示例代码
import mpl_scatter_density # adds projection='scatter_density'
from matplotlib.colors import LinearSegmentedColormap
# "Viridis-like" colormap with white background
white_viridis = LinearSegmentedColormap.from_list('white_viridis', [
(0, '#ffffff'),
(1e-20, '#440053'),
(0.2, '#404388'),
(0.4, '#2a788e'),
(0.6, '#21a784'),
(0.8, '#78d151'),
(1, '#fde624'),
], N=256)
def using_mpl_scatter_density(fig, x, y):
ax = fig.add_subplot(1, 1, 1, projection='scatter_density')
density = ax.scatter_density(x, y, cmap=white_viridis)
fig.colorbar(density, label='Number of points per pixel')
fig = plt.figure()
using_mpl_scatter_density(fig, x, y)
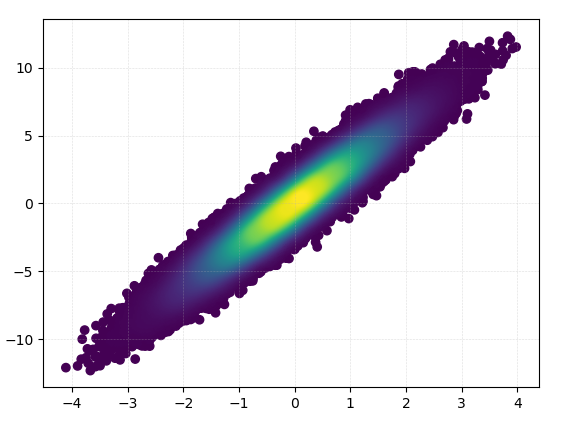
plt.show()画这个花了0.05秒:
而且变焦效果也很好:
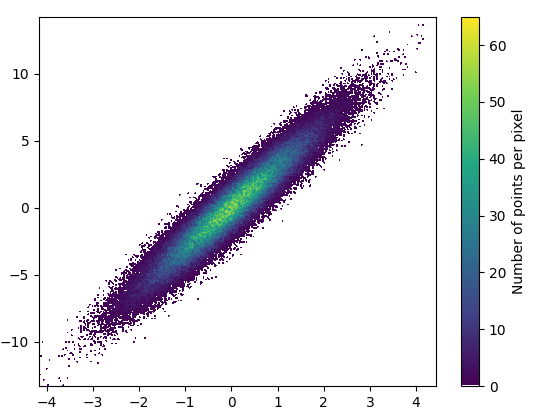
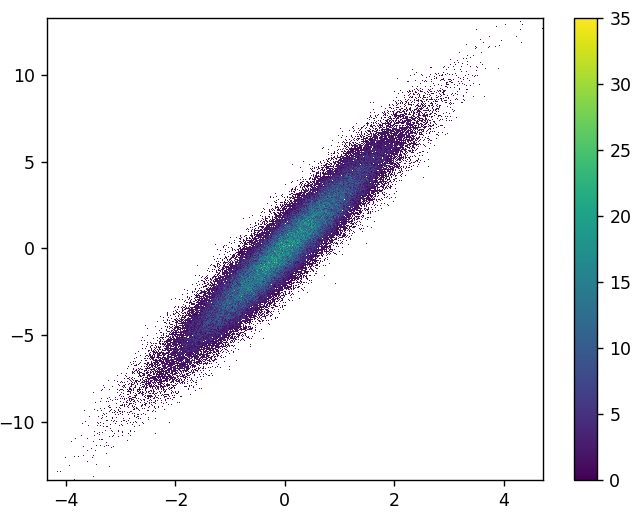
2: datashader
Installation
pip install datashader代码(双面显示的源代码和参数列表):
import datashader as ds
from datashader.mpl_ext import dsshow
import pandas as pd
def using_datashader(ax, x, y):
df = pd.DataFrame(dict(x=x, y=y))
dsartist = dsshow(
df,
ds.Point("x", "y"),
ds.count(),
vmin=0,
vmax=35,
norm="linear",
aspect="auto",
ax=ax,
)
plt.colorbar(dsartist)
fig, ax = plt.subplots()
using_datashader(ax, x, y)
plt.show()- 花了0.83秒才画出这样的图:
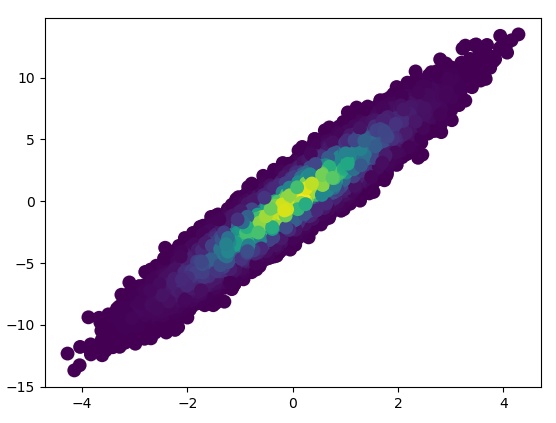
3: scatter_with_gaussian_kde
def scatter_with_gaussian_kde(ax, x, y):
# https://stackoverflow.com/a/20107592/3015186
# Answer by Joel Kington
xy = np.vstack([x, y])
z = gaussian_kde(xy)(xy)
ax.scatter(x, y, c=z, s=100, edgecolor='')- 花了11分钟才画出这幅图:
4: using_hist2d
import matplotlib.pyplot as plt
def using_hist2d(ax, x, y, bins=(50, 50)):
# https://stackoverflow.com/a/20105673/3015186
# Answer by askewchan
ax.hist2d(x, y, bins, cmap=plt.cm.jet)- 花了0.021秒绘制这个bins=(50,50):
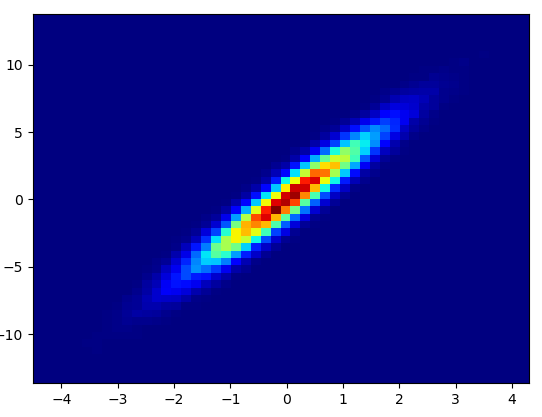
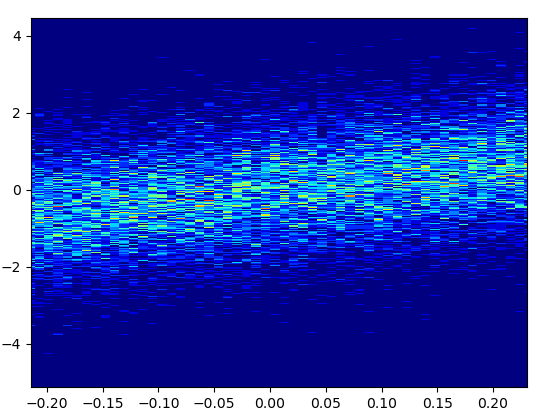
- 花了0.173秒绘制这个bins=(1000,1000):
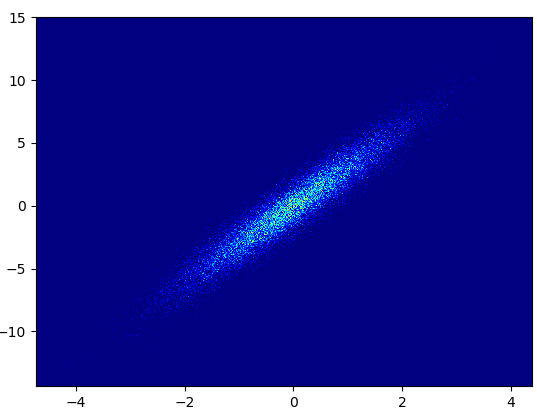
- 缺点:放大后的数据看起来不如mpl中的好--散射密度或数据增强器。另外,你必须自己决定垃圾箱的数量。
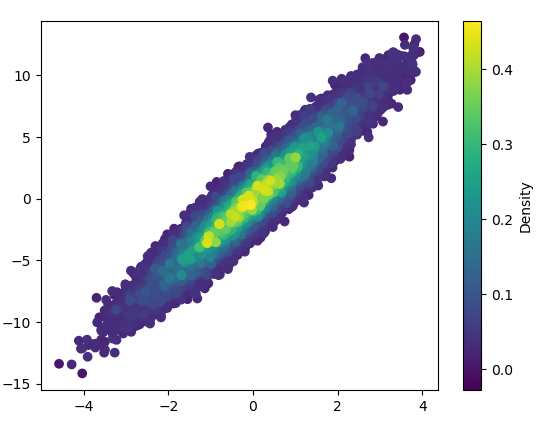
5: density_scatter
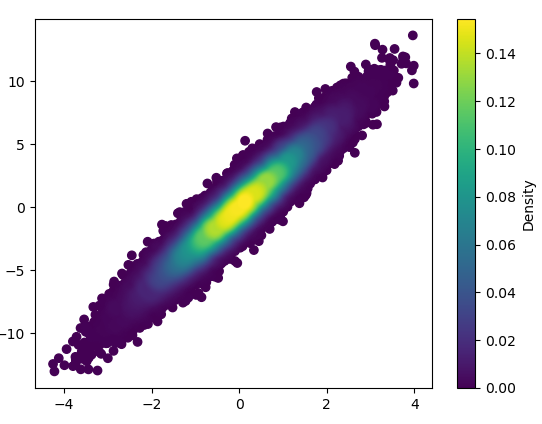
- 用bins=(1000,1000)绘制这幅图花费了0.368秒:
https://stackoverflow.com/questions/20105364
复制相似问题

