为什么ggplot2饼图面混淆了面标记
为什么ggplot2饼图面混淆了面标记
提问于 2014-03-07 07:53:51
我有两种类型的数据,如下所示:类型1 (http://dpaste.com/1697615/plain/)
Cluster-6 abTcells 1456.74119
Cluster-6 Macrophages 5656.38478
Cluster-6 Monocytes 4415.69078
Cluster-6 StemCells 1752.11026
Cluster-6 Bcells 1869.37056
Cluster-6 gdTCells 1511.35291
Cluster-6 NKCells 1412.61504
Cluster-6 DendriticCells 3326.87741
Cluster-6 StromalCells 2008.20603
Cluster-6 Neutrophils 12867.50224
Cluster-3 abTcells 471.67118
Cluster-3 Macrophages 1000.98164
Cluster-3 Monocytes 712.92273
Cluster-3 StemCells 557.88648
Cluster-3 Bcells 599.94109
Cluster-3 gdTCells 492.61994
Cluster-3 NKCells 524.42522
Cluster-3 DendriticCells 647.28811
Cluster-3 StromalCells 876.27875
Cluster-3 Neutrophils 1025.24105第二种类型(http://dpaste.com/1697602/plain/)。这些值与上文第1类型中的群集-6相同:
abTcells 1456.74119
Macrophages 5656.38478
Monocytes 4415.69078
StemCells 1752.11026
Bcells 1869.37056
gdTCells 1511.35291
NKCells 1412.61504
DendriticCells 3326.87741
StromalCells 2008.20603
Neutrophils 12867.50224但是,为什么在使用此代码处理第1类型数据时:
library(ggplot2);
library(RColorBrewer);
filcol <- brewer.pal(10, "Set3")
dat <- read.table("http://dpaste.com/1697615/plain/")
ggplot(dat,aes(x=factor(1),y=dat$V3,fill=dat$V2))+
facet_wrap(~V1)+
xlab("") +
ylab("") +
geom_bar(width=1,stat="identity",position = "fill") +
scale_fill_manual(values = filcol,guide = guide_legend(title = "")) +
coord_polar(theta="y")+
theme(strip.text.x = element_text(size = 8, colour = "black", angle = 0))现成数据:
> dput(dat)
structure(list(V1 = structure(c(2L, 2L, 2L, 2L, 2L, 2L, 2L, 2L,
2L, 2L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L, 1L), .Label = c("Cluster-3",
"Cluster-6"), class = "factor"), V2 = structure(c(1L, 5L, 6L,
9L, 2L, 4L, 8L, 3L, 10L, 7L, 1L, 5L, 6L, 9L, 2L, 4L, 8L, 3L,
10L, 7L), .Label = c("abTcells", "Bcells", "DendriticCells",
"gdTCells", "Macrophages", "Monocytes", "Neutrophils", "NKCells",
"StemCells", "StromalCells"), class = "factor"), V3 = c(1456.74119,
5656.38478, 4415.69078, 1752.11026, 1869.37056, 1511.35291, 1412.61504,
3326.87741, 2008.20603, 12867.50224, 471.67118, 1000.98164, 712.92273,
557.88648, 599.94109, 492.61994, 524.42522, 647.28811, 876.27875,
1025.24105)), .Names = c("V1", "V2", "V3"), class = "data.frame", row.names = c(NA,
-20L))产生了以下数字:
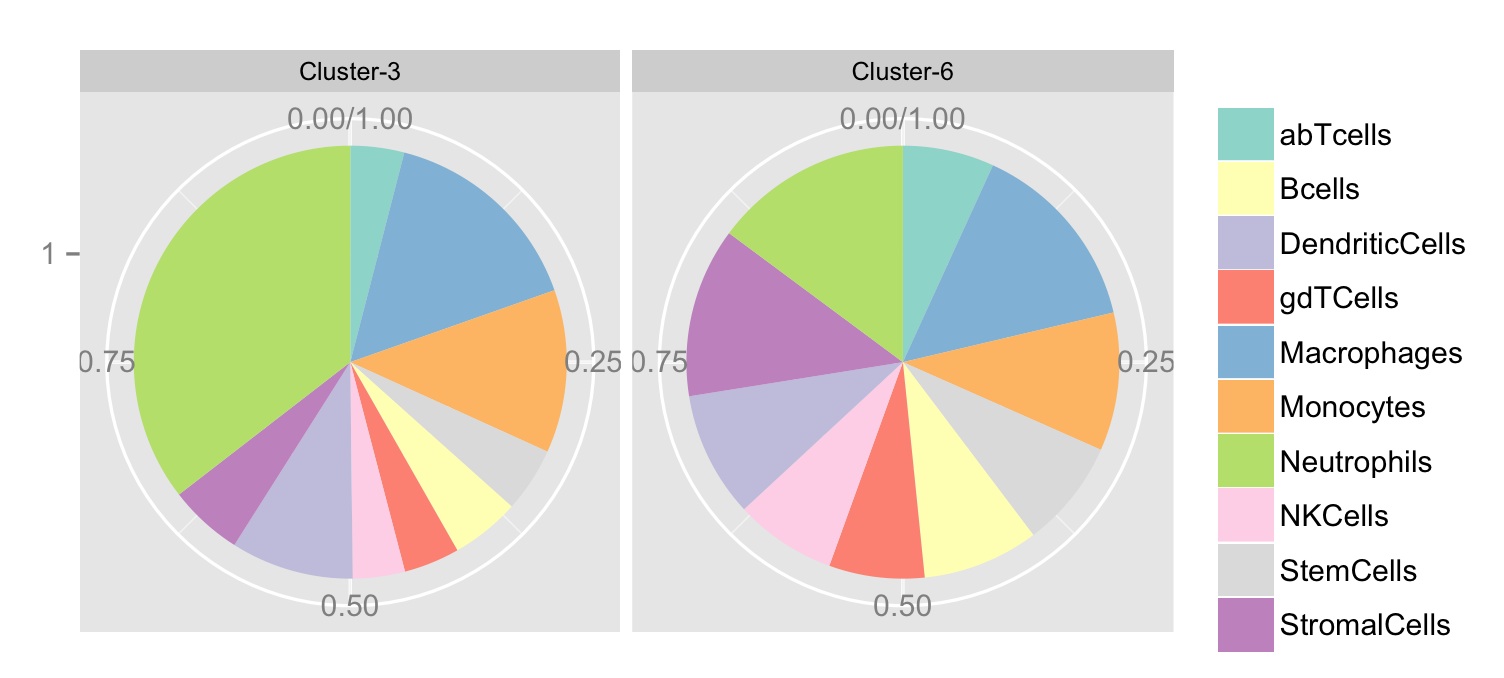
注意,小面标签不正确,群集-3应该是群集-6,其中中性粒细胞所占的比例更大。
我如何解决这个问题?
在处理类型2的数据时,根本没有问题。
library(ggplot2)
df <- read.table("http://dpaste.com/1697602/plain/");
library(RColorBrewer);
filcol <- brewer.pal(10, "Set3")
ggplot(df,aes(x=factor(1),y=V2,fill=V1))+
geom_bar(width=1,stat="identity")+coord_polar(theta="y")+
theme(axis.title = element_blank())+
scale_fill_manual(values = filcol,guide = guide_legend(title = "")) +
theme(strip.text.x = element_text(size = 8, colour = "black", angle = 0))现成数据:
> dput(df)
structure(list(V1 = structure(c(1L, 5L, 6L, 9L, 2L, 4L, 8L, 3L,
10L, 7L), .Label = c("abTcells", "Bcells", "DendriticCells",
"gdTCells", "Macrophages", "Monocytes", "Neutrophils", "NKCells",
"StemCells", "StromalCells"), class = "factor"), V2 = c(1456.74119,
5656.38478, 4415.69078, 1752.11026, 1869.37056, 1511.35291, 1412.61504,
3326.87741, 2008.20603, 12867.50224)), .Names = c("V1", "V2"), class = "data.frame", row.names = c(NA,
-10L))
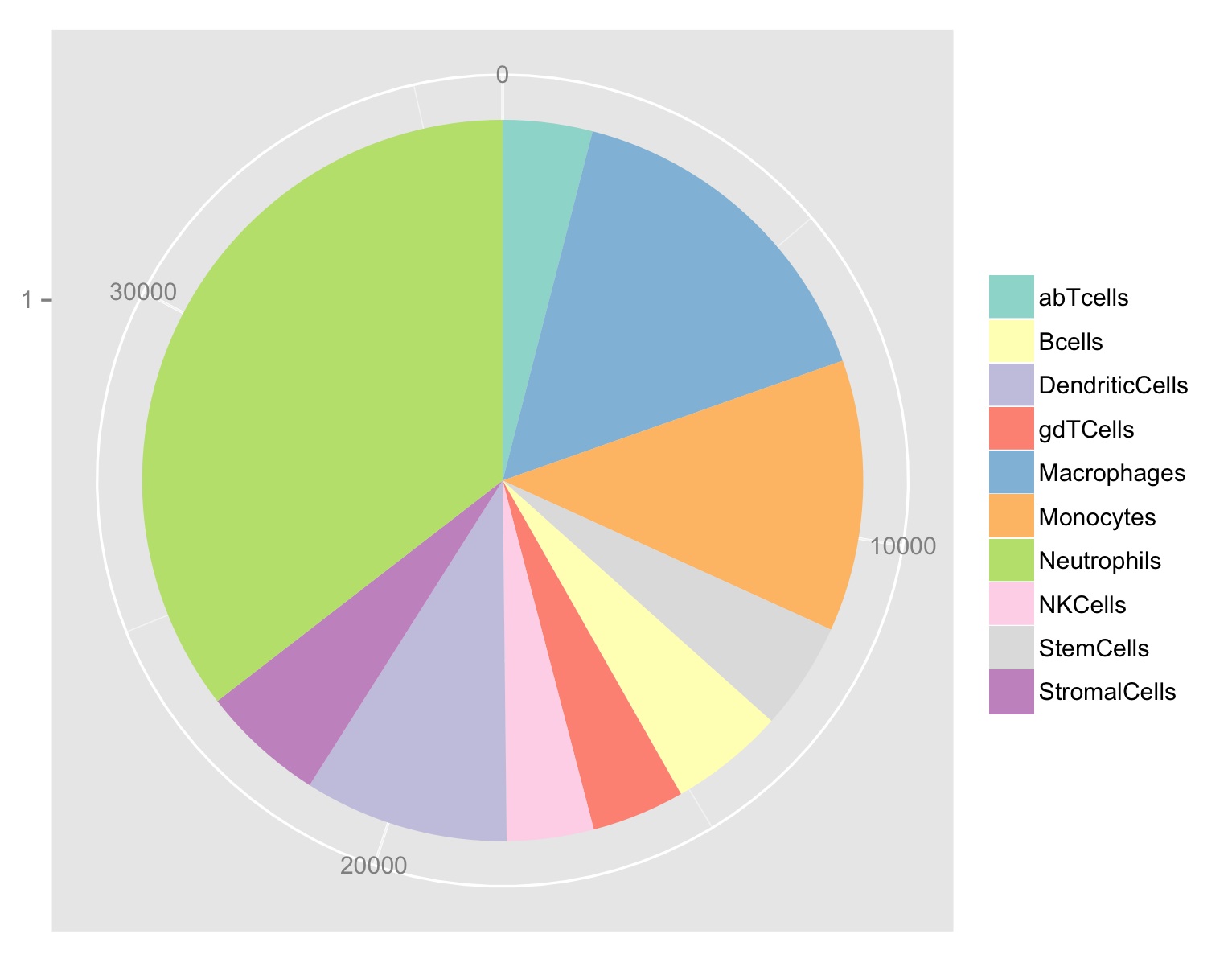
回答 1
Stack Overflow用户
回答已采纳
发布于 2014-03-07 08:26:30
这是因为您在aes(...)中使用了数据帧名。这解决了问题。
ggplot(dat,aes(x=factor(1),y=V3,fill=V2))+
facet_wrap(~V1)+
xlab("") +
ylab("") +
geom_bar(width=1,stat="identity",position = "fill") +
scale_fill_manual(values = filcol,guide = guide_legend(title = "")) +
coord_polar(theta="y")+
theme(strip.text.x = element_text(size = 8, colour = "black", angle = 0))
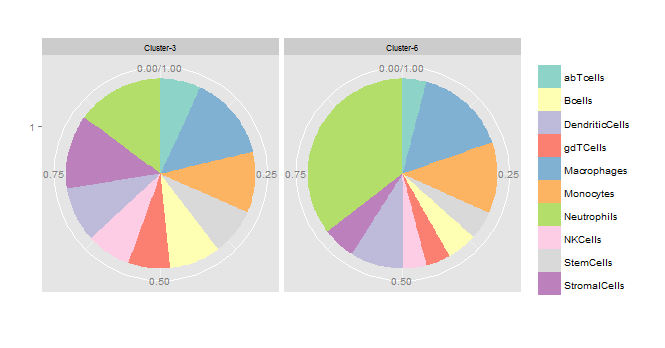
在定义方面时,在默认数据集的上下文中引用V1,ggplot按级别进行字母排序(因此“集群-3”优先)。在对aes(...)的调用中,直接引用dat$V3,因此ggplot从默认dataset的上下文中移到原始dataframe。在那里,群集-6是第一。
作为一般性评论,不应该在用aes(...)定义的数据集上下文之外引用data=...中的数据。所以:
ggplot(data=dat, aes(y=V3...)) # good
ggplot(data=dat, aes(y=dat$V3...)) # bad您的问题是第二个选项为什么不好的一个完美例子。
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/22244500
复制相关文章
相似问题

