如何在R中可视化一个大型网络?
网络可视化已成为科学实践中的普遍现象。但是随着网络规模的扩大,普通的可视化变得不那么有用了。有太多的节点/顶点和链接/边。通常,可视化的努力最终会产生“发球”。
为克服这一问题,提出了一些新的办法,例如:
- 边缘捆绑:
- http://vis.stanford.edu/papers/divided-edge-bundling或
- https://gephi.org/tag/edge-bundling/
- 层次边缘捆绑:
- http://graphics.cs.illinois.edu/sites/graphics.dev.engr.illinois.edu/files/edgebundles.pdf
- 组属性布局:
- 3/用户手册
- 如何在照片中进行分组布局?
我相信还有更多的办法。因此,我的问题是:如何克服发球问题,即如何使用R?可视化大型网络。
下面是一些模拟示例性网络的代码:
# Load packages
lapply(c("devtools", "sna", "intergraph", "igraph", "network"), install.packages)
library(devtools)
devtools::install_github(repo="ggally", username="ggobi")
lapply(c("sna", "intergraph", "GGally", "igraph", "network"),
require, character.only=T)
# Set up data
set.seed(123)
g <- barabasi.game(1000)
# Plot data
g.plot <- ggnet(g, mode = "fruchtermanreingold")
g.plot

这个问题与可视化对GraphViz来说太大的无向图吗?有关。但是,这里我搜索的不是一般的软件建议,而是的具体示例(使用上面提供的数据),这些技术通过使用R (可与本线程中的示例R:点太多的散点图相比较)帮助实现大型网络的良好可视化。
回答 5
Stack Overflow用户
发布于 2014-03-19 05:44:19
另一种显示大型网络的方法是使用BioFabric (www.BioFabric.org),它使用水平线而不是点来表示节点。然后使用垂直线段显示边缘。该技术的快速D3演示见:http://www.biofabric.org/gallery/pages/SuperQuickBioFabric.html。
BioFabric是一个Java应用程序,但是一个简单的R版本可以在:https://github.com/wjrl/RBioFabric上获得。
下面是R代码的一个片段:
# You need 'devtools':
install.packages("devtools")
library(devtools)
# you need igraph:
install.packages("igraph")
library(igraph)
# install and load 'RBioFabric' from GitHub
install_github('RBioFabric', username='wjrl')
library(RBioFabric)
#
# This is the example provided in the question:
#
set.seed(123)
bfGraph = barabasi.game(1000)
# This example has 1000 nodes, just like the provided example, but it
# adds 6 edges in each step, making for an interesting shape; play
# around with different values.
# bfGraph = barabasi.game(1000, m=6, directed=FALSE)
# Plot it up! For best results, make the PDF in the same
# aspect ratio as the network, though a little extra height
# covers the top labels. Given the size of the network,
# a PDF width of 100 gives us good resolution.
height <- vcount(bfGraph)
width <- ecount(bfGraph)
aspect <- height / width;
plotWidth <- 100.0
plotHeight <- plotWidth * (aspect * 1.2)
pdf("myBioFabricOutput.pdf", width=plotWidth, height=plotHeight)
bioFabric(bfGraph)
dev.off()下面是发问者提供的数据的BioFabric版本,尽管以m>1的值创建的网络更有趣。嵌入细节显示了网络左上角的特写;节点BF4是网络中的最高度节点,默认布局是从该节点开始对网络进行宽度优先搜索(忽略边缘方向),相邻节点按降低节点度的顺序遍历。请注意,我们可以立即看到,例如,大约60%的节点bf4的邻居为1级,我们还可以从严格的45度下边缘看出,这个1000节点的网络有999个边,因此是一棵树。
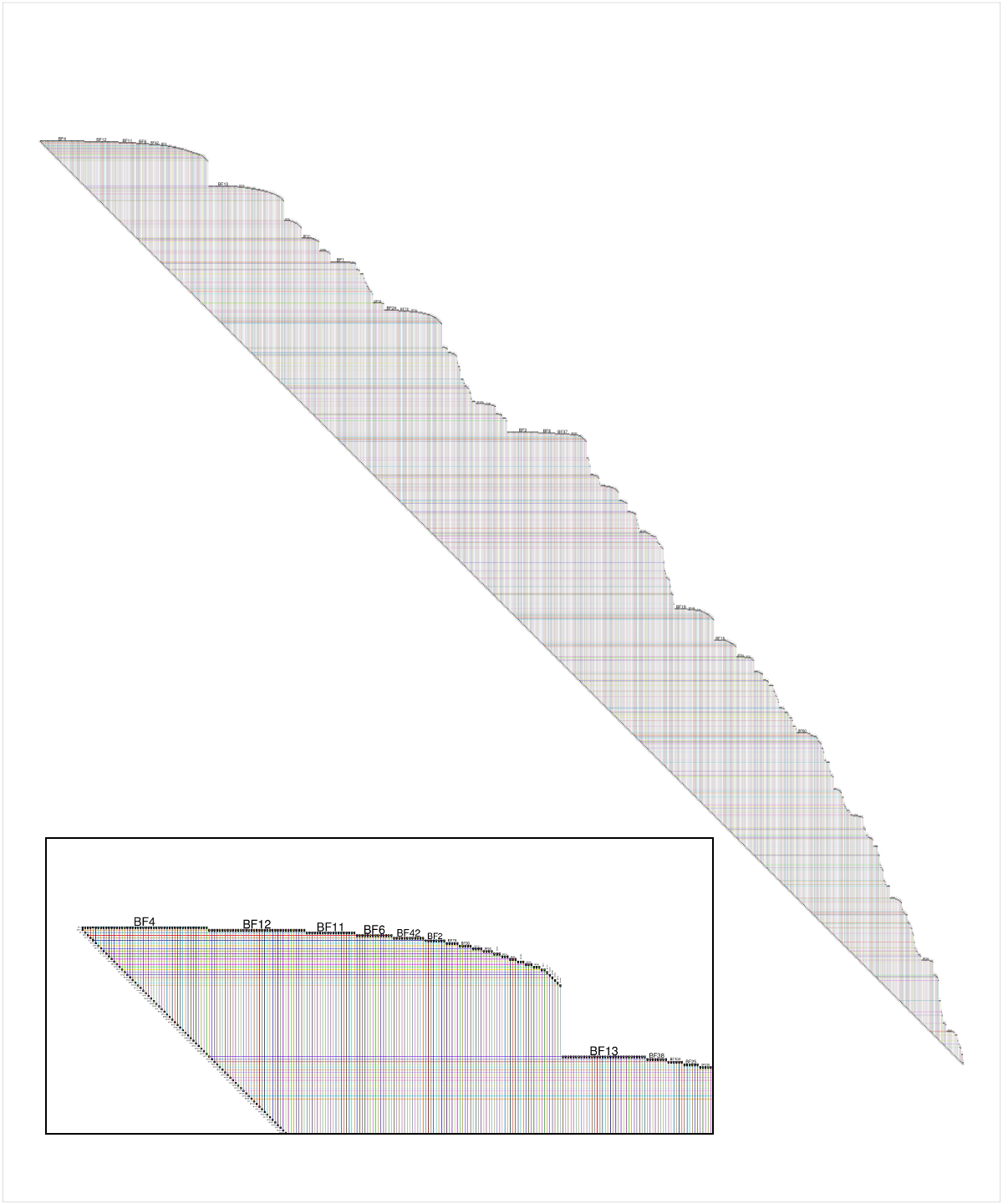
完全公开: BioFabric是我写的一个工具。
Stack Overflow用户
发布于 2016-01-25 20:33:01
我最近一直在处理这个问题。因此,我想出了另一个解决方案。按社区/集群折叠图表。这一做法类似于上文任择议定书概述的第三种选择。作为警告,这种方法在无向图中效果最好。例如:
library(igraph)
set.seed(123)
g <- barabasi.game(1000) %>%
as.undirected()
#Choose your favorite algorithm to find communities. The algorithm below is great for large networks but only works with undirected graphs
c_g <- fastgreedy.community(g)
#Collapse the graph by communities. This insight is due to this post http://stackoverflow.com/questions/35000554/collapsing-graph-by-clusters-in-igraph/35000823#35000823
res_g <- simplify(contract(g, membership(c_g))) 这个过程的结果是下图,其中顶点的名称代表社区成员。
plot(g, margin = -.5)
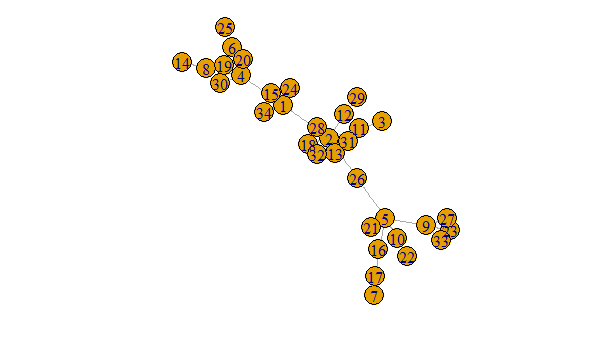
上面的情况显然比这个可怕的烂摊子要好得多。
plot(r_g, margin = -.5)

要将社区链接到原始顶点,您需要类似于以下内容
mem <- data.frame(vertices = 1:vcount(g), memeber = as.numeric(membership(c_g)))海事组织,这是一个很好的方法,有两个原因。首先,它可以在理论上处理任意大小的图形。发现社区的过程可以在折叠图上不断重复。第二,采用交互式方法将产生非常可读的结果。例如,可以想象用户可以单击折叠图中的一个顶点来扩展该社区,显示其所有原始顶点。
Stack Overflow用户
发布于 2019-08-05 12:30:51
我环顾四周,没有找到好的解决办法。我的方法一直是删除节点和发挥边缘的透明度。与其说这是一种技术解决方案,不如说是一种设计解决方案,但我已经能够在我的笔记本电脑上绘制多达5万条边界的类似地理信息的网络,而没有太多复杂的情况。
以你为例:
plot(simplify(g), vertex.size= 0.01,edge.arrow.size=0.001,vertex.label.cex = 0.75,vertex.label.color = "black" ,vertex.frame.color = adjustcolor("white", alpha.f = 0),vertex.color = adjustcolor("white", alpha.f = 0),edge.color=adjustcolor(1, alpha.f = 0.15),display.isolates=FALSE,vertex.label=ifelse(page_rank(g)$vector > 0.1 , "important nodes", NA))

twitter的例子提到了拥有30,000条边的网络:

https://stackoverflow.com/questions/22453273
复制相似问题

