R:使用grep类函数提取表中的cerain位。
R:使用grep类函数提取表中的cerain位。
提问于 2014-04-15 09:25:29
感谢所有回复的人。让我重新编辑我的上一篇文章,以明确我需要什么。我确实有一个如下结构的输入数据:
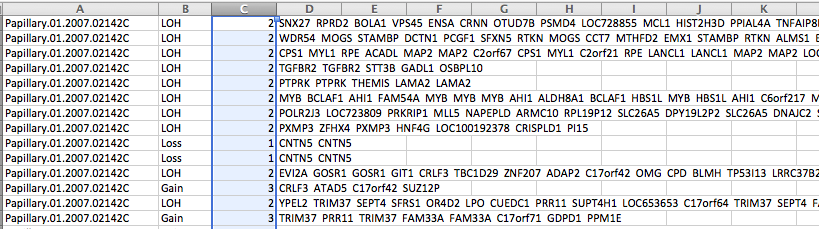
第一列有一个样本名,第二和第三列是事件的描述,最后一列有一组由“”分隔的基因。当然,这是来自XLS的截图,但是我没有问题把它们导入R。
我想在R中创建一个循环,遍历表中的每一行和每一列,并搜索作为向量提供的基因名。(例如:基因<- c("APC","TP53"))。如果它找到匹配项,那么它将把这一行复制到新的矩阵中。然而,我确实想知道它找到的载体上的哪个基因,因此所有D列的剩余基因都必须省略。
我应该能够创建循环,但在执行grep函数时失败了,并且不太确定如何从行中省略其余的基因。
理想的输出应该如下所示:
01.2007.02142C LOH 2 BOLA1
非常感谢你的想法。
回答 1
Stack Overflow用户
回答已采纳
发布于 2014-04-15 09:53:41
这个怎么样:
# define data, could be read from file with dat <- read.table(file, sep="\t")
dat <- as.data.frame(matrix(
c("Papillary.01.2007.02142C", "LOH", "2", "SNX27 RPD2 BOLA1",
"Papillary.01.2007.02142C", "LOH", "2", "APC MOGS STAMBP",
"Papillary.01.2007.02142C", "LOH", "2", "TGFBR2",
"Papillary.01.2007.02142C", "LOH", "2", "TP52 CNTN5 EV12A",
"Papillary.01.2007.02142C", "LOH", "2", "APC TRIM37 PRR11 FAM33A"),
ncol=4, byrow=T
))
# gene vector
vec <- c("APC", "TP53", "TP52")
# apply function over the gene vector, to return a data frame with
# matching rows. Also include column containing query gene
out <- lapply(vec, function(gene) {
df <- dat[grep(gene, dat[, 4]), ]
df$match <- rep(gene, nrow(df))
df
})
# combine results and subset to get desired output
out <- do.call(rbind, out)
out[, c(1:3, 5)]输出:
V1 V2 V3 match
2 Papillary.01.2007.02142C LOH 2 APC
5 Papillary.01.2007.02142C LOH 2 APC
4 Papillary.01.2007.02142C LOH 2 TP52请注意,如果两个不同的基因匹配同一行,则行将在输出中重复。
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/23079595
复制相关文章
相似问题

