如何从GMM迭代中给出集群的标签?
如何从GMM迭代中给出集群的标签?
提问于 2014-10-16 12:58:58
我从理解高斯混合模型的概念上读到了GMM的概念。这对我很有帮助。我也为fisheriris实现了GMM,但我没有使用fitgmdist函数,因为我没有它。所以我使用了来自http://chrisjmccormick.wordpress.com/2014/08/04/gaussian-mixture-models-tutorial-and-matlab-code/的代码。
当我读到理解高斯混合模型的概念时,Amro可以用它的标签来绘制结果,即setosa、virginica和versicolor。他是怎么做到的?经过几次迭代后,我只得到了mu、Sigma和体重。根本没有标签。我想将标签(setosa、virginica和versicolor)放到GMM迭代中的混合模型中。
回答 1
Stack Overflow用户
发布于 2014-10-18 09:08:46
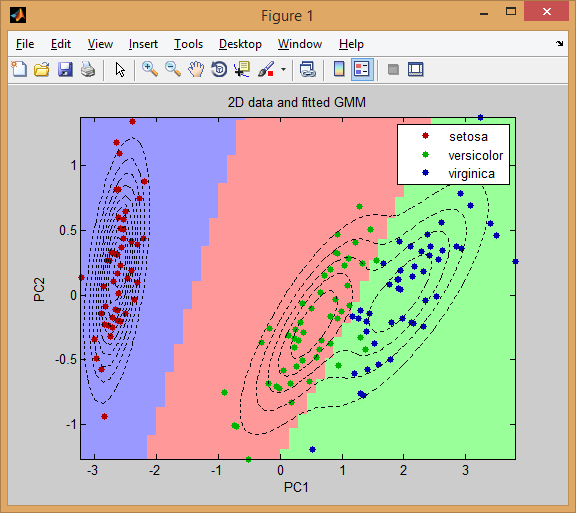
在这个情节中有两套“标签”:
- 一个是Fisher Iris数据集的“真”标签(包含每个实例的类的
species变量:setoas、versicolor或virginica)。通常,您不会在真正的数据集中拥有这些数据集(毕竟,集群的目标是在数据中发现这些组,您事先并不知道)。我只是在这里使用它们来了解EM聚类对实际事实执行得有多好(散点点根据类进行颜色编码)。 - 另一组标签是我们使用GMM发现的集群。基本上,我构建了一个包含整个数据域的50x50二维点网格,然后通过计算后验概率并选择具有最高似然度的分量,为每个点分配一个集群。我用背景色展示了这些星系团。作为一个很好的结果,我们可以看到簇之间的判别式决策边界。
您可以看到左边的点簇被很好地分开了(并且与setosa类完全匹配)。当右方的点在两个匹配的类中被分开时,尽管有实例“错误分类”(如果你愿意的话)(在边界的错误一侧的一些绿色点)。
通常,在一个真实的环境中,您将没有那些实际的类来与之进行比较,因此无法判断集群有多“准确”(聚类性能评价还有其他度量标准)。
页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/26405107
复制相关文章
相似问题

