如何从回归树rpart对象生成预测间隔?
如何从回归树rpart对象生成预测间隔?
提问于 2015-03-18 19:39:39
如何从适合使用rpart的回归树生成预测间隔?
我的理解是,回归树以叶节点的平均值作为响应的模型。我不知道如何从模型中得到叶节点的方差,但我想要做的是模拟使用叶节点的均值和方差来获得预测间隔。
Predict.rpart()没有给出间隔的选项。
示例:I将一棵树与虹膜数据相匹配,但没有选项"interval“
> r1 <- rpart(Sepal.Length ~ ., cp = 0.001, data = iris[1:nrow(iris)-1,])
> predict(r1,newdata=iris[nrow(iris),],type = "interval")
Error in match.arg(type) :
'arg' should be one of “vector”, “prob”, “class”, “matrix”回答 2
Stack Overflow用户
回答已采纳
发布于 2015-03-30 16:16:51
我不清楚回归树的置信区间意味着什么,因为它们不是像线性模型那样的经典统计模型。我主要看到两种用途:描述你的树的确定性,或者描述对树的每一片叶子的预测的精确性。下面给出每一种可能性的答案。
描述你的树的确定性
如果您正在为一个拆分节点寻找一个置信值,那么party将直接提供它,因为它使用置换测试,并在统计上确定哪些变量是最重要的,并将p值附加到每个拆分中。party的ctree函数比rpart的这里有很大的优越性。
回归树集叶的置信区间
第三,如果你在为每一片叶子的值寻找一个区间的可信度,那么0.025,0.975分位数的间隔是你最想要的。party中的默认绘图在显示每片叶子的输出值时采用类似的方法:
library("party")
r2 <- ctree(Sepal.Length ~ .,data=iris)
plot(r2)
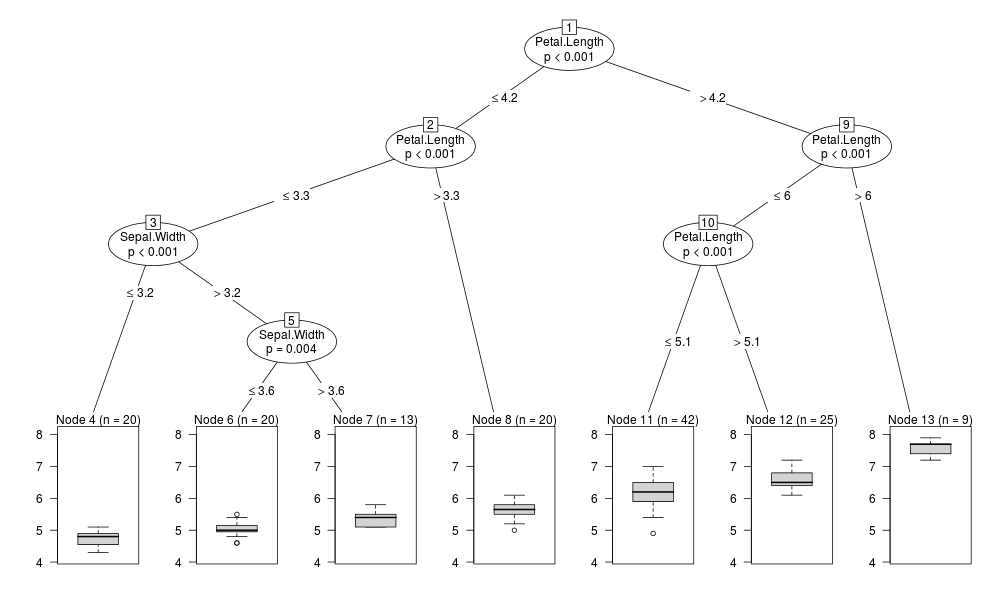
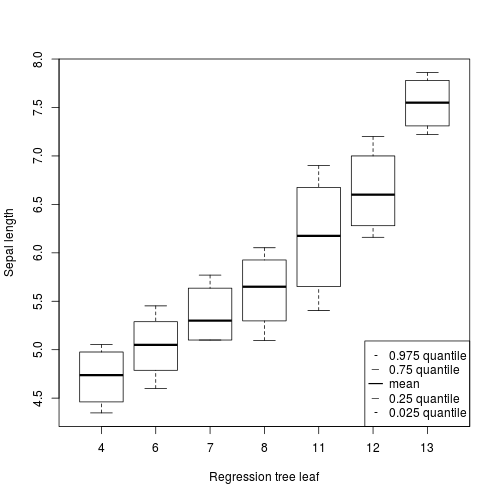
检索相应的间隔只需:
iris$leaf <- predict(r2, type="node")
CIleaf <- aggregate(iris$Sepal.Length,
by=list(leaf=iris$leaf),
quantile,
prob=c(0.025, 0.25, 0.75, 0.975))很容易想象:
plot(as.factor(CIleaf$leaf), CIleaf[, 2],
ylab="Sepal length", xlab="Regression tree leaf")
legend("bottomright",
c(" 0.975 quantile", " 0.75 quantile", " mean",
" 0.25 quantile", " 0.025 quantile"),
pch=c("-", "_", "_", "_", "-"),
pt.lwd=0.5, pt.cex=c(1, 1, 2, 1, 1), xjust=1)
Stack Overflow用户
发布于 2015-03-31 13:25:52
也许一种选择是简单地引导您的培训数据?
library(rpart)
library(boot)
trainData <- iris[-150L, ]
predictData <- iris[150L, ]
rboot <- boot(trainData, function(data, idx) {
bootstrapData <- data[idx, ]
r1 <- rpart(Sepal.Length ~ ., bootstrapData, cp = 0.001)
predict(r1, newdata = predictData)
}, 1000L)
quantile(rboot$t, c(0.025, 0.975))
2.5% 97.5%
5.871393 6.766842 页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/29131254
复制相关文章
相似问题

