如何利用R将干涉图变换成红外光谱?
如何利用R将干涉图变换成红外光谱?
提问于 2015-04-15 18:00:30
大家好,在互联网上,我有一个干涉图(时域),想要把它转换成红外光谱(频域),得到分子中官能团的峰。傅里叶变换红外光谱( FTIR )是测定分子中官能团的一种先进方法。所谓的干涉仪就是这样建造的:
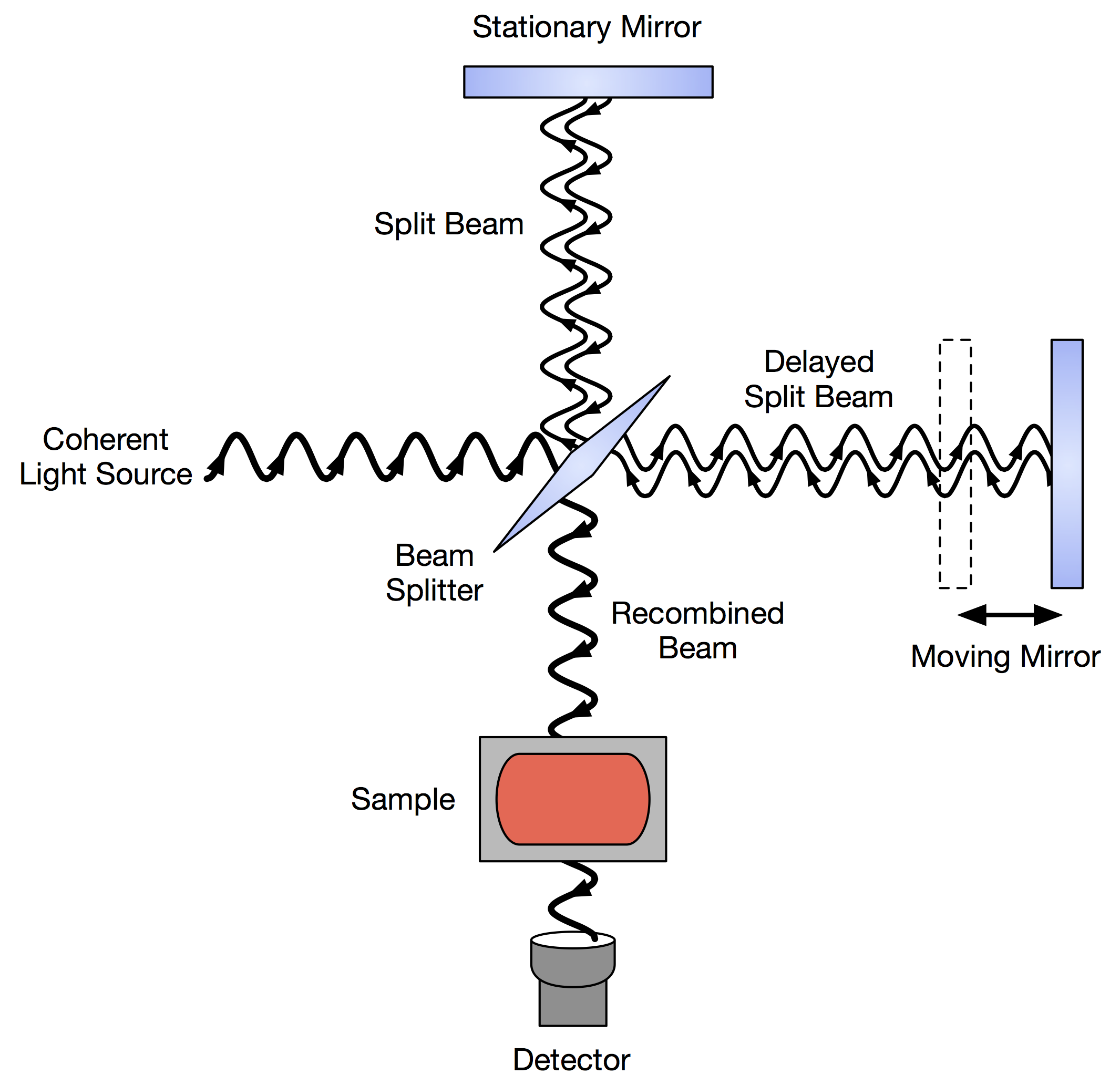
干涉图以伏特和镜像为单位,以纳米为单位绘制信号强度,如下文所示:
par(family="mono", font.axis=1)
data <- read.table("D13-4-aminobenzoic_acid_interferogram.asc")
x <- data[,1]
y <- data[,2]
plot(x,y,
type="l",
xlab="Mirror position [mm]",
ylab="Signal intensity [V]",
axes=F,
)
axis(1)
axis(2)下面是带有测量数据的链接到.asc文件。在傅里叶变换之后,光谱应该如下所示:
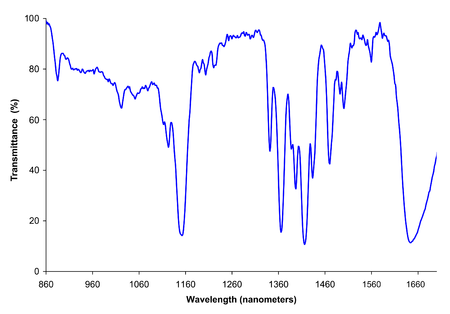
我的问题是:如何利用fft()从干涉图到红外光谱,在R中进行快速离散傅里叶变换?是R中从光谱到干涉图的反向变换,如果是的话,它是如何实现的。干杯,克里斯
回答 1
Stack Overflow用户
回答已采纳
发布于 2015-04-15 19:50:31
试试这个解决方案:
d <- read.table('D13-4-aminobenzoic_acid_interferogram.asc')
f <- fft(d[,2])
# do fft(f,inverse=T) to get the unnormalized inverse
f2 <- sqrt(Re(f)^2 + Im(f)^2)
c <- 2.9979e8 # speed of light
lambda.laser <- 632.8e-9 # (nm) HeNe
nu.Nyquist <- 1e-2/lambda.laser # upper limit of the wavenumber
delta.nu <- nu.Nyquist/nrow(d) # wavenumber spacing
i.nu <- 1:floor(length(f2)/2) # show plot up to the Nyquist limit
plot((i.nu-1)*delta.nu,f2[i.nu],type='l')页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/29657480
复制相关文章
相似问题

