显微研究中异种织构组织周围的边界绘制
显微研究中异种织构组织周围的边界绘制
提问于 2015-08-31 20:34:29
我正在整理一个OpenCV脚本来分析免疫组织化学染色的心脏组织。我们的染色程序使表达特定蛋白质的细胞类型在其质膜上在光学显微镜下可见色素,我们用来拍摄这些图像。
到目前为止,我已经成功地将图像分割到基于颜色范围的不同层次,使用了通过OpenCV社区(colorspaces.html)提供的经常引用的颜色分割脚本的修改版本。
原始图像的屏幕截图:
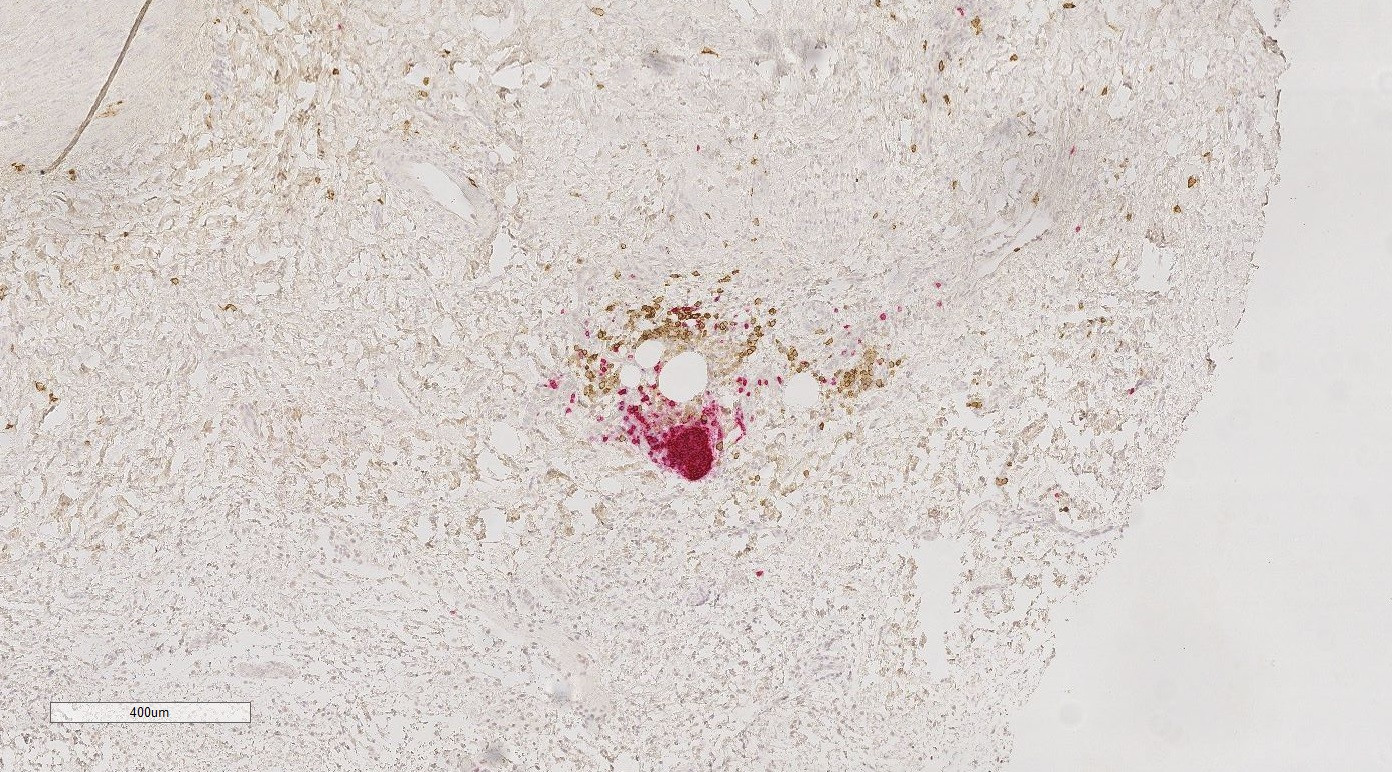
B-细胞层显示:

在这一点上,我想计算B细胞与未染色组织的面积之比。此操作提示根据颜色范围提取背景单元格层:

显然,这些结果仍有许多有待改进之处。
,有人对如何处理这个问题有想法吗?再次,我想分割背景组织(透明)层,不幸的是,这是相当海绵状的纹理。我的目标是创造一个不染色组织区域的面具代表。似乎是一种模糊的技术来填补组织中的空白,但是这种方法在准确性上的损失是显而易见的。
回答 1
Stack Overflow用户
回答已采纳
发布于 2015-09-01 15:13:01
在样本图像中,通道看起来高度相关。如果你应用去相关伸展到图像,你应该能够看到更多的细节。这里,在我的博客文章中,我用C++实现了去相关的扩展(不是偶然的,不是Python)。
使用博客中的示例代码,我做了以下分割单元格区域的工作:
- 用下面的targetMean和tergetSigma显示CIE实验室的图像。 mu3 = {128.0f,128.0f,128.0f};sd3 = {128.0f,5.0f,5.0f};
- 将扩展的CIE实验室图像转换回BGR。
- 用3x3矩形结构元素一次侵蚀这个BGR图像。
- 将kmeans聚类应用于这个k= 2的受侵蚀图像。
我不知道这个分割有多好。我认为,通过对上述参数(均值、σ、结构元素大小和图像被侵蚀次数)尝试不同的值,可以获得更好的分割效果。
(以下图片与原始比例尺不同)
原件:
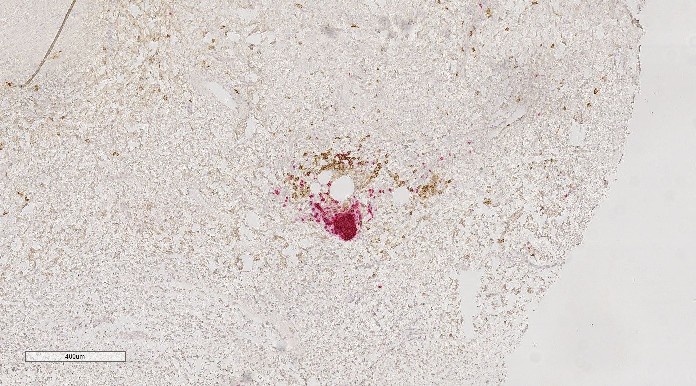
扩展的CIE实验室转换为BGR:
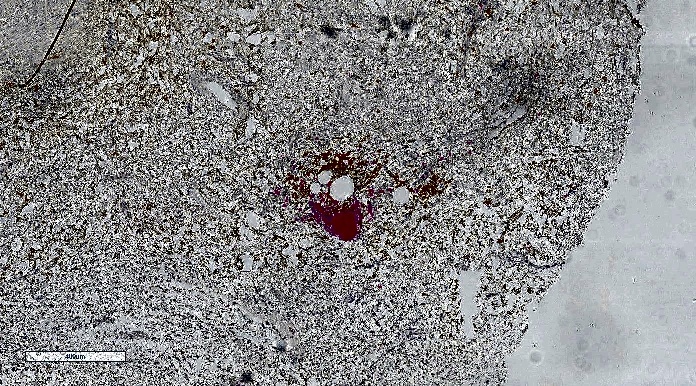
侵蚀:

K=2的kmeans:

页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/32319534
复制相关文章
相似问题

