如何在R不重叠的情况下旋转系统发育的节点?
如何在R不重叠的情况下旋转系统发育的节点?
提问于 2016-07-07 20:16:00
我正在努力为我在R中的系统发育做一个很好的数字(我正在努力地使用R来进行分析)。这些树是用加利和MrBayes制作的。加利(最大的可能性)树的行为一直与所有典型的猿类,动物工具,和水牛命令。我有颜色,分类标签,节点标签等等。
但是我的MrBayes树没有表现出来。编辑-现在我的另一棵栀子树也不合作。
节点不能正常旋转。还有什么事我需要改变吗?
下面是一个示例:
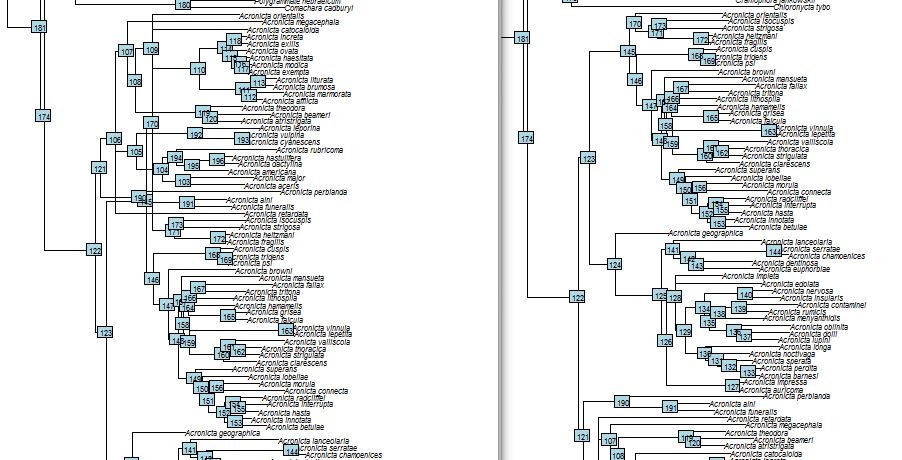
mbcpart <- read.nexus("acronicta_101_taxa.nex.con.tre")
outgroup <- c("Nacna_malachitis", "Nacna_sugitanii", "Gerbathodes_paupera", "Belciades_niveola", "Moma_kolthoffi", "Moma_alpium")
rmbcpart <- root(mbcpart, outgroup, resolve.root=TRUE)
rmbcpart <- rotate(rmbcpart, 175)
rmbcpart <- rotate(rmbcpart, 122)
rmbcpart <- rotate(rmbcpart, 108)然后,我做了一个新的窗口,并绘制了这个东西。连在一起的是两棵树。右边的是原图,没有旋转。左边的那个是旋转后的。看起来它们是,大多是旋转的,但是有几个分类群被拖走或落在后面。这会导致重叠的分支。
有人知道我能做些什么来解决这个问题吗?
Stack Overflow用户
发布于 2020-06-04 21:16:39
这实际上只是一个包装黑客的函数,但我建议您尝试phytools::untangle。例如:
library(phytools)
tree<-untangle(tree,"read.tree")页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/38254500
复制相关文章
相似问题

