ggplot2 (geom_path,geom_bar,ggbio)的Karyogram和SNP
我想用SNP标记画一个核型图。
它与函数segments一起工作,但是我想使用ggplot2包来显示一个优雅的图形。

- ggbio:
我用函数
layout_karyogram检查了ggbio包,但是染色体是在垂直位置绘制的。我没有找到一种方法来旋转每个染色体下面的名字的图表,并写我的SNP的名字在他们的片段旁边。

geom_bar: 然后我尝试了来自包geom_bar的ggplot2:
数据<- data.framework(chromosome=paste0(“chr”,1:4),size=c(100,400,300,200),stringsAsFactors = FALSE) dat$染色体<-因子(dat$染色体,level=dat$染色体)SNP<-data.framework(chromosome=c(1,1,2,3,4),Position=c(50,70,250,20,290,110),Type=c("A","B","B"),labels=c("SNP1","SNP2","SNP3","SNP4","SNP5","SNP6")) p<-aes(data=data,aes,(y=size)+ geom_bar( stat=“恒等”,fill="grey70",宽度= .5) +theme_bw() p+ geom_segment(data=SNP,aes(x=SNP$染色体-0.2,xend=SNP$chromosome+0.2,y=SNP$Position,yend=SNP$Position,colour=SNP$Type) )+注释(“text”,标签=SNP$标签,x=SNP$染色体-0.5,y=SNP$位置,大小= 2,colour= "red")
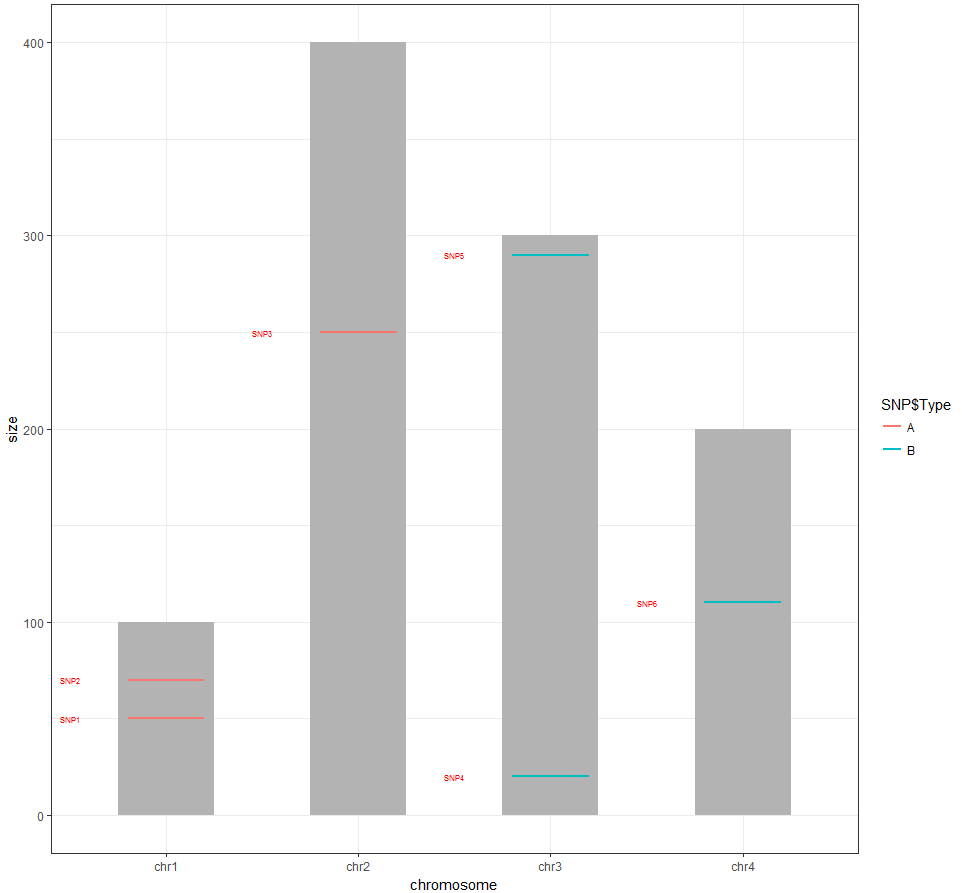
这里唯一的问题是,它看上去更像是条形图,而不是染色体。我想要圆形的四肢。I found someone who got the same problem as I am。
geom_path: 我没有使用geom_bar,而是使用geom_path和选项lineend = "round"来获得圆形的四肢。
Geom_path()+data=NULL(x=c(1,1),y=c(1,100),size=3,lineend=“圆形”)
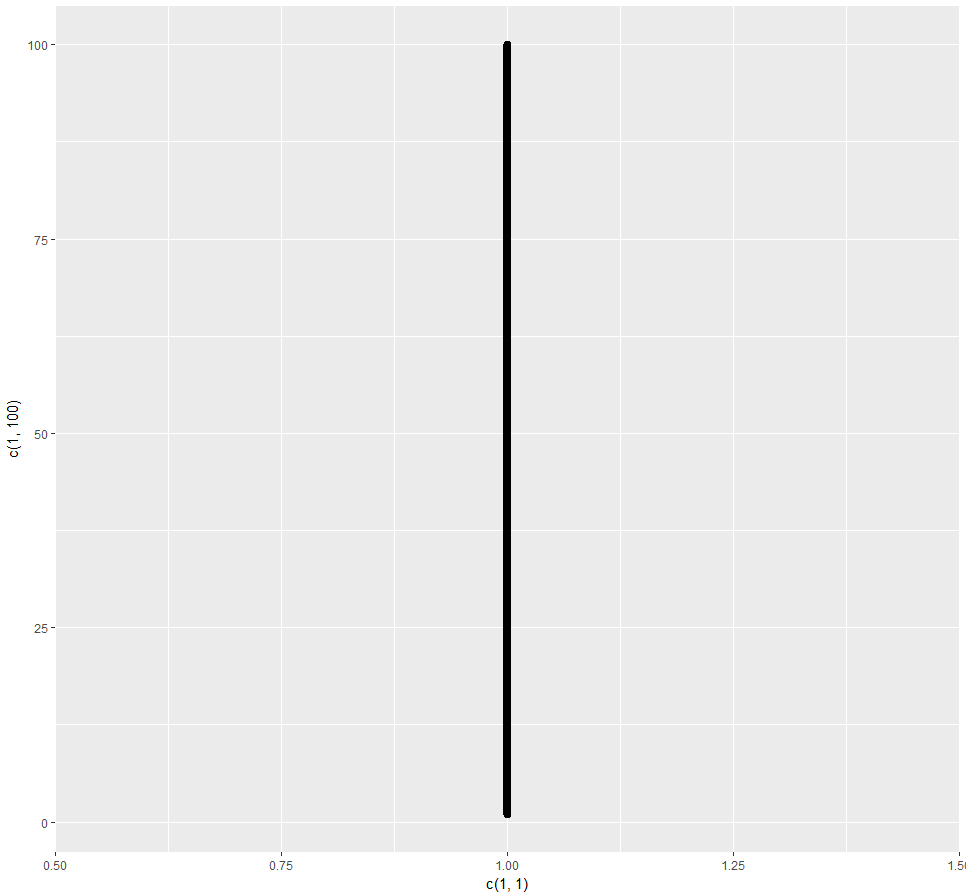
这个形状看上去很好。所以我试着运行几条染色体的代码。
数据(chromosome=paste0(“chr”,1:4),size=c(100,400,300,200),stringsAsFactors = FALSE) for (i in 1:length(data,1)){ p <- p+ geom_path(data=NULL,mapping=aes(x=c,i),y=c(1,datai,2)),size=3,lineend=“圆形”}}
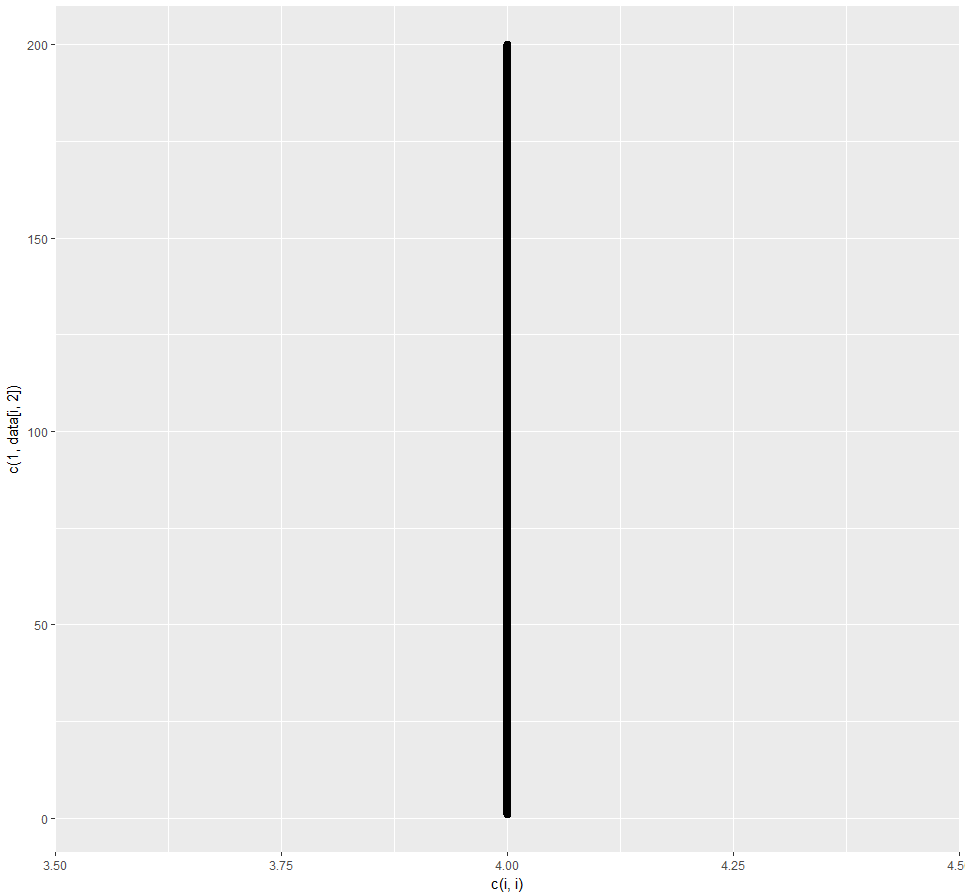
它不起作用,我不知道为什么,但是p只保存最后的染色体,而不是在我的染色体图中画出四条染色体。
对这些问题有什么建议吗?
回答 1
Stack Overflow用户
发布于 2017-03-28 12:38:56
我会去找geom_segment。SNP段的x开始/结束是硬编码的(as.integer(chr) -+ 0.05),但否则代码相当简单。
ggplot() +
geom_segment(data = data,
aes(x = chr, xend = chr, y = 0, yend = size),
lineend = "round", color = "lightgrey", size = 5) +
geom_segment(data = SNP,
aes(x = as.integer(chr) - 0.05, xend = as.integer(chr) + 0.05,
y = pos, yend = pos, color = type),
size = 1) +
theme_minimal()
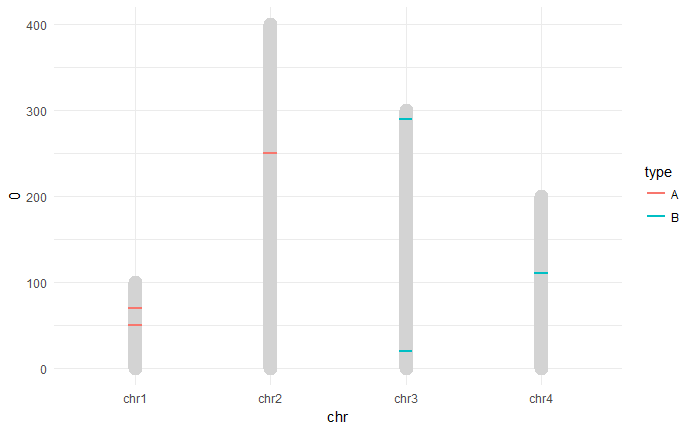
data <- data.frame(chr = paste0("chr", 1:4),
size = c(100, 400, 300, 200))
SNP <- data.frame(chr = paste0("chr", c(1, 1, 2, 3, 3, 4)),
pos = c(50, 70, 250, 20, 290, 110),
type = c("A", "A", "A", "B", "B", "B"))https://stackoverflow.com/questions/43065723
复制相似问题

