如何在R中做多个wilcox.test?
如何在R中做多个wilcox.test?
提问于 2017-09-14 02:27:27
我有这个矩阵,目的是在R(对照与案例)中做Wilcoxon检验,但我不知道如何正确地放入我的矩阵。
gene.name cont1 cont2 cont3 case1 case2 case3
A 10 2 3 21 18 8
B 14 8 7 12 34 22
C 16 9 19 21 2 8
D 32 81 17 29 43 25
..回答 1
Stack Overflow用户
回答已采纳
发布于 2017-09-14 07:41:00
你可以试试:
# load your data
d <- read.table(text="gene.name cont1 cont2 cont3 case1 case2 case3
A 10 2 3 21 18 8
B 14 8 7 12 34 22
C 16 9 19 21 2 8
B 32 81 17 29 43 25", header=T)
library(tidyverse)
# transform to long format using dplyr (included in tidyverse)
dlong <- as.tbl(d) %>%
gather(key, value,-gene.name) %>%
mutate(group=ifelse(grepl("cont",key), "control", "case"))
# plot the data
dlong %>%
ggplot(aes(x=group, y=value)) +
geom_boxplot()
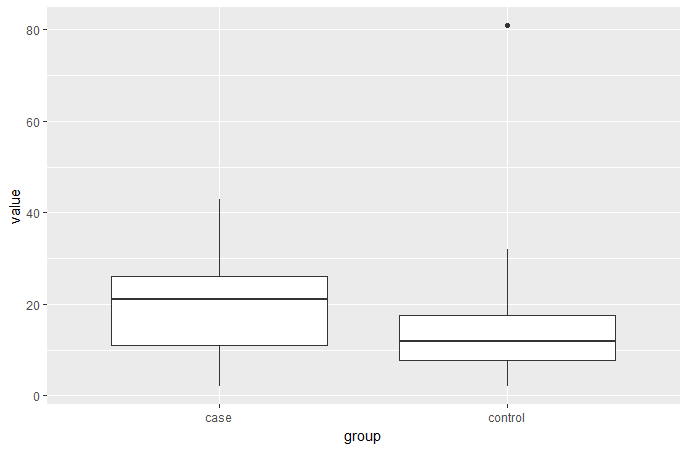
# run the test
dlong %>%
with(., wilcox.test(value ~ group))
Wilcoxon rank sum test with continuity correction
data: value by group
W = 94.5, p-value = 0.2034
alternative hypothesis: true location shift is not equal to 0编辑
# as you don't clarified how to handle the double occurence of B I assume
# thats a typo and fixed the second B to D
library(ggpubr)
dlong <- as.tbl(d) %>%
mutate(gene.name=LETTERS[1:4]) %>%
gather(key, value,-gene.name) %>%
mutate(group=ifelse(grepl("cont",key), "control", "case"))
# plot the boxplot with Wilcoxen p-values using ggpubr
dlong %>%
ggplot(aes(x=gene.name, y=value, fill=group)) +
geom_boxplot() +
stat_compare_means(method= "wilcox.test")
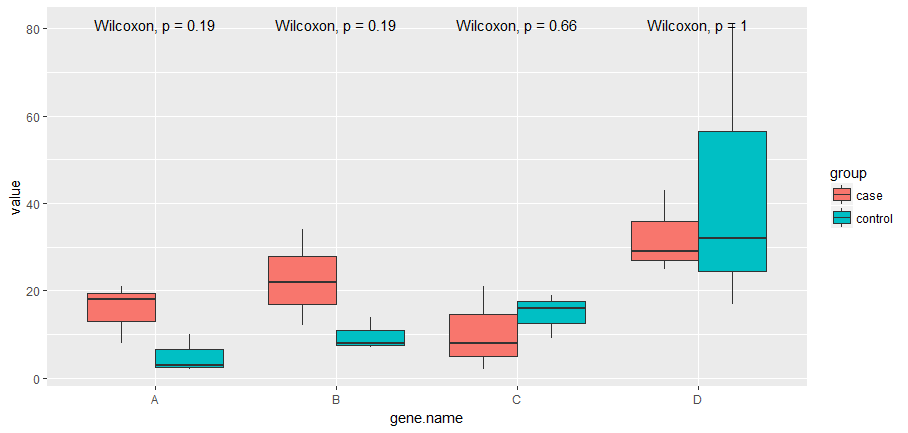
# get the pvalues
dlong %>%
group_by(gene.name) %>%
summarise(p=wilcox.test(value~group)$p.value)
# A tibble: 4 x 2
gene.name p
<chr> <dbl>
1 A 0.2
2 B 0.2
3 C 0.7
4 D 1.0或者使用apply试试R基。
res <- apply(d[,-1], 1, function(x){
wilcox.test(x ~ c(1,1,1,2,2,2))$p.value
})
cbind.data.frame(Genes=as.character(d$gene.name), p=res, BH=p.adjust(res, method = "BH"))
Genes p BH
[1,] 1 0.2 0.4000000
[2,] 2 0.2 0.4000000
[3,] 3 0.7 0.9333333
[4,] 2 1.0 1.0000000页面原文内容由Stack Overflow提供。腾讯云小微IT领域专用引擎提供翻译支持
原文链接:
https://stackoverflow.com/questions/46209548
复制相关文章
相似问题

