用numpy计算向量的自相关
我正在努力想出一种非混淆的,高效的方法,用numpy来计算一组3D向量中的自相关函数。
我在三维空间中有一组向量,保存在一个数组中。
a = array([[ 0.24463039, 0.58350592, 0.77438803],
[ 0.30475903, 0.73007075, 0.61165238],
[ 0.17605543, 0.70955876, 0.68229821],
[ 0.32425896, 0.57572195, 0.7506 ],
[ 0.24341381, 0.50183697, 0.83000565],
[ 0.38364726, 0.62338687, 0.68132488]])它们的自相关函数定义为
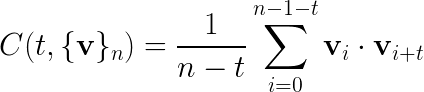
如果上面的图像不可用,公式也打印在下面: C(t,{v}n) = \frac 1{n-t}\sum{i=0}^{n-1-t}\vec v_i\cdot\vec v_{i+t}
我正努力用一种有效的、非混淆的方式对此进行编码。我可以用两个嵌套的for循环来解释这一点,但这很慢。通过使用numpy中的嵌入式函数之一,有一种快速的方法,但它们似乎使用的是完全不同的相关函数定义。这里已经解决了一个类似的问题,如何使用numpy.correlate进行自相关?,但它不能处理向量。你知道我该怎么解决这个问题吗?
回答 3
Stack Overflow用户
发布于 2018-02-17 21:27:42
NumPy例程用于一维数组。作为“最小”的改进,对规范化步骤使用向量化操作(在最后一行中使用np.arange ):
def vector_autocorrelate(t_array):
n_vectors = len(t_array)
# correlate each component indipendently
acorr = np.array([np.correlate(t_array[:,i],t_array[:,i],'full') for i in xrange(3)])[:,n_vectors-1:]
# sum the correlations for each component
acorr = np.sum(acorr, axis = 0)
# divide by the number of values actually measured and return
acorr /= (n_vectors - np.arange(n_vectors))
return acorr对于较大的数组大小,应考虑使用傅里叶变换算法进行相关。如果您对此感兴趣,请查看库滴定动力学的示例(免责声明:我编写了库,它只依赖于NumPy)。
作为参考,这里列出了NumPy代码(我为测试编写的)、代码vector_autocorrelate和时间动态的时间。
size = [2**i for i in range(4, 17, 2)]
np_time = []
ti_time = []
va_time = []
for s in size:
data = np.random.random(size=(s, 3))
t0 = time.time()
correlate = np.array([np.correlate(data[:,i], data[:,i], mode='full') for i in range(data.shape[1])])[:,:s]
correlate = np.sum(correlate, axis=0)/(s-np.arange(s))
np_time.append(time.time()-t0)
t0 = time.time()
correlate = tidynamics.acf(data)
ti_time.append(time.time()-t0)
t0 = time.time()
correlate = vector_autocorrelate(data)
va_time.append(time.time()-t0)您可以看到结果:
print("size", size)
print("np_time", np_time)
print("va_time", va_time)
print("ti_time", ti_time)尺寸16,64,256,1024,4096,16384,65536 np_time 0.00023794174194335938,0.0002703666687011719,0.0002713203430175781,0.001544952392578125,0.0278470516204834,0.36094141006469727,6.922360420227051 va_time 0.00021696090698242188,0.0001690387725830078,0.000339508056640625,0.0014629364013671875,0.024930953979492188,0.34442687034606934,7.005630731582642 ti_time 0.0011148452758789062,0.0008449554443359375,0.0007512569427490234,0.0010488033294677734,0.0026645660400390625,0.007939338684082031,0.048232316970825195
或者策划他们
plt.plot(size, np_time)
plt.plot(size, va_time)
plt.plot(size, ti_time)
plt.loglog()对于非常小的数据序列,"N**2“算法是不可用的。
Stack Overflow用户
发布于 2020-05-06 18:27:04
嗨,我遇到了一个伊米拉问题。这是我的主意
def fast_vector_correlation(M):
n_row = M.shape[0]
dot_mat = M.dot(M.T)
corr = [np.trace(dot_mat,offset=x) for x in range(n_row)]
corr/=(n_row-np.arange(n_row))
return corr其思想是dot_mat包含行向量之间的所有标量积。要计算不同t值下的相关性,只需将对角线之和(右上角部分),如图片中所示。
Stack Overflow用户
发布于 2018-02-17 18:01:59
我在这里发布答案,以防其他人需要它,因为我花了相当长的时间才找到一个可行的方法。我最终通过定义以下函数来解决这个问题
def vector_autocorrelate(t_array):
n_vectors = len(t_array)
# correlate each component indipendently
acorr = np.array([np.correlate(t_array[:,i],t_array[:,i],'full') for i in xrange(3)])[:,n_vectors-1:]
# sum the correlations for each component
acorr = np.sum(acorr, axis = 0)
# divide by the number of values actually measured and return
acorr = np.array( [ val / (n_vectors - i) for i,val in enumerate(acorr)])
return acorr如果有人有一个更好的主意,我真的很想听到,因为我认为目前的一个仍然没有那么紧凑,它应该是。这总比没有好,这就是为什么我要在这里张贴它。
https://stackoverflow.com/questions/48844295
复制相似问题
