如何从MRI图像中去除一个模态- Python
我正在尝试使用MRI脑成像数据作为深度学习模型。目前,我的图像有4个维度,如下所示,但我只想保留MRI图像的T1c模式,因为我的模型输入应该是1通道三维磁共振成像(T1c)。
我确实尝试过使用Nibabel包,如下所示
import nibabel as nib
ff = glob.glob('imagesTr\*')
a = nib.load(ff[0])
a.shape这将返回以下输出
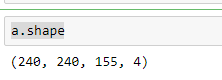
我也在粘贴“a”的标题信息。
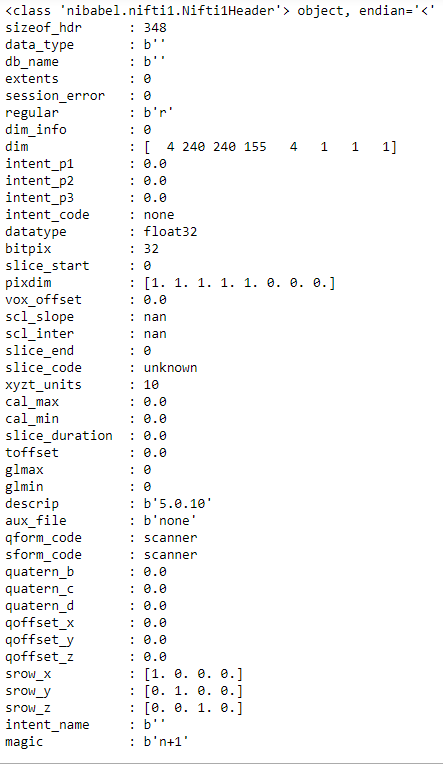
由此,哪一个维度被用来识别像T1、T2、T1c、FLAIR等核磁共振成像方式?我怎么能只保留T1c呢?你能帮忙吗?
回答 2
Stack Overflow用户
发布于 2019-06-21 08:23:45
首先,您需要识别第四维度中图像存储的顺序。
标题可能会有帮助:
print(a.header)接下来,要只保留一个模式,可以使用以下方法:
data = a.get_fdata()
modality_1 = data[:,:,:,0]编辑1:
根据挑战网站:
所有的BraTS多模式扫描都可用NIfTI文件(.nii.gz)和描述a)本机(T1)和b)后对比T1加权(T1Gd),c T2加权(T2)和d) T2液体衰减反转恢复(FLAIR)体积,并通过不同的临床方案和来自多个(n=19)机构的各种扫描仪获得,这里提到的数据贡献者。
和
所提供的数据经过预处理后分布,即共同注册到相同的解剖模板,内插到相同分辨率(1mm^3)和颅骨剥离。
,所以在这种情况下,标头不会有帮助(由于预处理,所有模式的尺寸相等)。
如果您正在寻找后对比T1加权(T1Gd)图像,那么它是二维的,所以请使用:
data = a.get_fdata()
modality_1 = data[:,:,:,1]另外,我们可以可视化每个3D卷(data[:,:,:,0], data[:,:,:,1],data[:,:,:,2], data[:,:,:,3])并验证我的声明。
Stack Overflow用户
发布于 2019-11-04 11:06:18
无法从Nifti头部识别出MRI的类型。您需要原始的DICOM映像来派生这种类型的信息。
然而,你可以在视觉上检查你的图像,并比较白质、灰质和脑室的对比度/颜色,以确定你的图像是否是T1、T2、FLAIR等。例如,在T1图像中,你会期望更深的灰质、较轻的白质和黑色的脑脊液。在T2图像中,你会看到较轻的灰质、较暗的白质和白色的脑脊液。天赋和T2是一样的,但是有“倒置”脑脊液。参见这里的一些脑图像示例:https://casemed.case.edu/clerkships/neurology/Web%20Neurorad/t1t2flairbrain.jpg
话虽如此,你似乎有一个4维的图像,这意味着某种时间序列,所以我假设你的数据是DTI或fMRI或类似的东西。
也不可能将一种类型的磁共振成像转换成另一种类型,所以如果你的数据集还不是T1,那么就无法在一个期望干净的T1数据的模型中使用它。
我强烈鼓励你更多地了解MRI和你所使用的数据类型。否则就不可能解释你的结果了。
https://stackoverflow.com/questions/56698087
复制相似问题

