R绘图|基因表达水平分布图绘制
在整理转录组下游的时候,看到中科新生命的报告中的基因表达水平分布部分有这么一个图
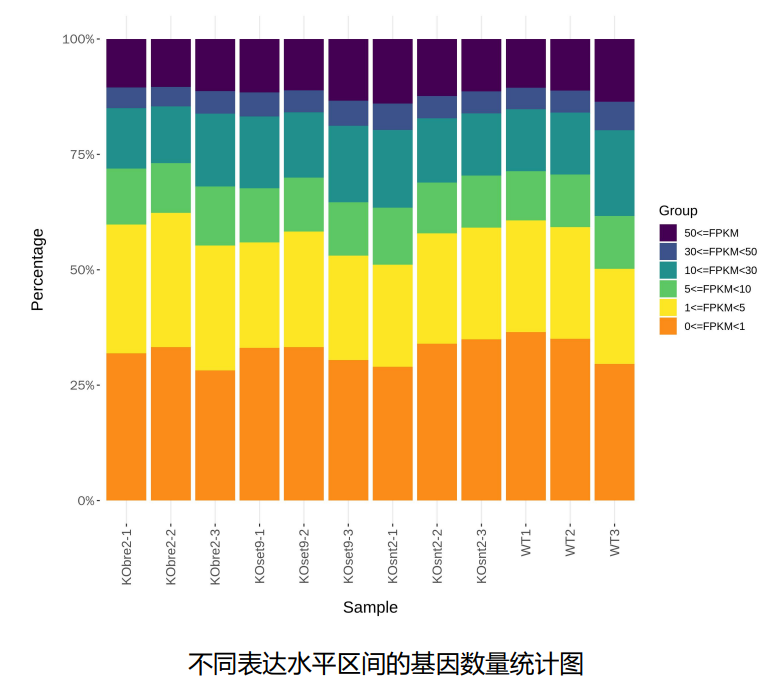
从图中可以非常直观的看出来不同样本在不同表达区间的分布情况。由于报告没有给出源代码,我们模仿的画一画。
想要画出这样一个基因表达水平分布图,我们需要两个东西
- 基因表达矩阵
- 数据的分布情况
基因表达矩阵
原始表达矩阵比较容易获取,为了方便演示,我们直接采取edgeR[1]的cpm标准化拿到基因表达矩阵。
rawcount <- read.table("data/rawcounts.txt",row.names = 1,
sep = "\t", header = T)
library(edgeR)
express_cpm <- log2(cpm(rawcount)+1)
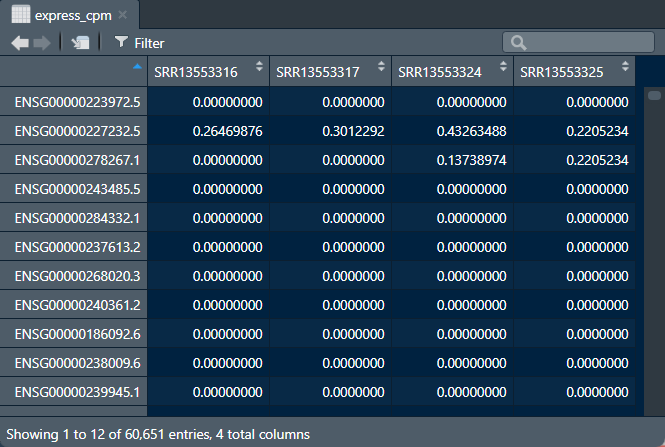
标准化后的基因表达矩阵
数据的分布情况
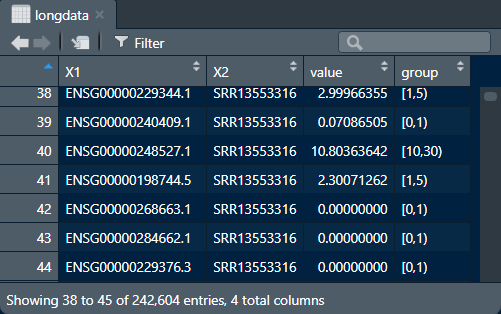
接下来我们需要将现有的表达情况按一定标准分类,需要用到R包reshape[2]
# 载入R包
library(reshape)
# 宽变长
longdata <- melt(data = express_cpm)
# 将数据划分成6个区间
cut_data <- cut(as.numeric(longdata$value), breaks = c(0,1,5,10,30,50,Inf),right = F,include.lowest = TRUE)
# 将分组结果添加到longdata
longdata$group = cut_data
# 给区间命名
levels(cut_data) <- c("0<=CPM<1", "1<=CPM<5","5<=CPM<10","10<=CPM<30","30<=CPM<50","50<=CPM<+∞")
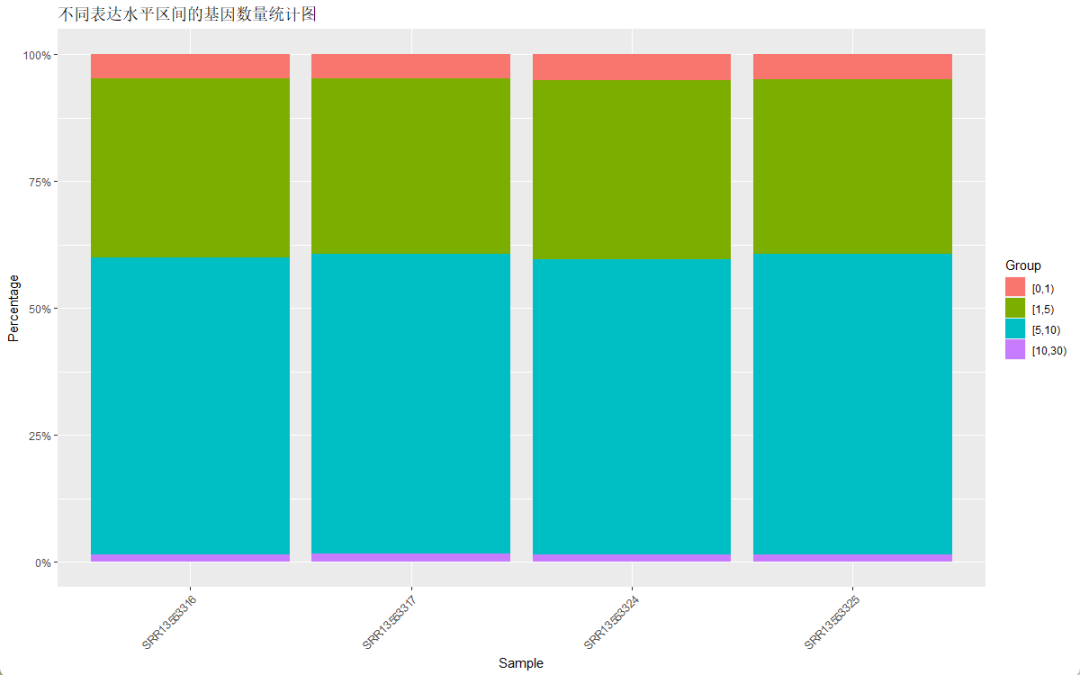
画图
画图需要用到R包ggplot2[3]
# 载入R包
library(ggplot2)
# 画图
ggplot(longdata, aes(x = X2, fill = group, y = value)) +
geom_bar(aes(fill = group), position = "fill",stat = "identity")+
scale_y_continuous(labels = scales::percent)+
theme(axis.text.x = element_text(angle = 45, hjust = 1))+
# 添加横纵坐标和title
labs(title = "不同表达水平区间的基因数量统计图",
x = "Sample", y = "Percentage", fill = "Group")
- angle = 45:设置样本名倾斜角度为45°
- hjust = 1:设置样本名距离图形的距离为1(自行调试)
- labs( ):添加坐标轴
参考资料
[1] edgeR: https://bioconductor.org/packages/release/bioc/html/edgeR.html
[2] reshape: https://www.rdocumentation.org/packages/stats/versions/3.6.2/topics/reshape
[3] ggplot2: https://www.rdocumentation.org/packages/ggplot2/versions/3.3.5
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2021-12-11,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
