物种分类原理
一、微生物物种鉴定发展历史
微生物由于形态微小,数目众多,分类鉴定一直都是一大难题,传统的微生物鉴定方法,主要是根据微生物的表型特征,例如形态学、生理生化学,生态学等特征来推断微生物的系统发育。但是由于微生物形体微小、结构简单等特点,微生物的分类鉴定除了传统的分类方法之外,还必须寻找新的特征作为分类鉴定的依据。分子生物学的发展,使我们不仅可以根据表型特征,而且可以从分子水平上,通过研究和比较微生物乃至整个生物界的遗传型特征,甚至生物的基因组特征来研究生物的进化、发育和分类鉴定。
1 形态学特征
2 生理生化特征
3 血清分型
4 DNA 碱基组成
5 DNA 杂交
6 16S 序列测序
7 宏基因组序列测序
二、商业微生物检测平台
One Codex:https://www.onecodex.com/
Diversigen :https://www.diversigen.com/
CosmosID :https://www.cosmosid.com/
Real Time Genomics:https://www.realtimegenomics.com/三、纳米孔微生物鉴定平台
1、epi2me WIMP:https://epi2me.nanoporetech.com/
2、NanoSPEC:https://nanospc.mmmoxford.uk/
3、Bugseq:https://bugseq.com/
4、NanoPipe:http://bioinformatics.uni-muenster.de/tools/nanopipe/index.hbi?
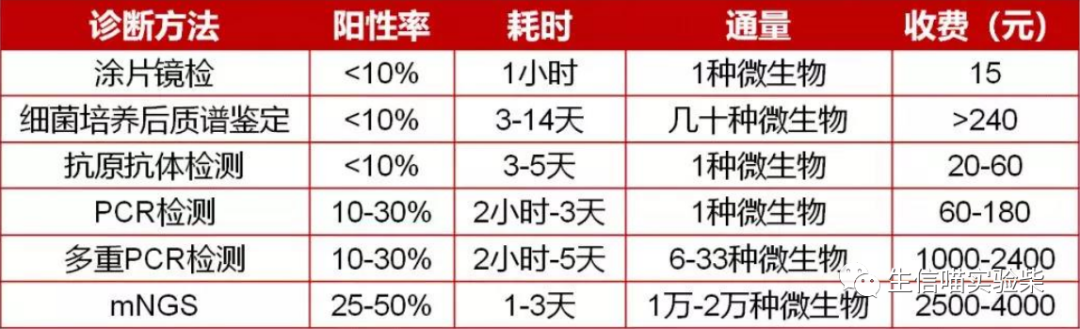
5、NanoOK:https://github.com/richardmleggett/NanoOK四、几种检测方法费用比较
五、微生物世界中的“种”
自然界的物种之间可能是连续进化的,是一个量的变化,而分类则是定性概念。因此就会出现很多处于中间状态的特例,比如分类上即接近于A,又接近于B。
种是基本的分类单元,它指的是“物种”,而物种的概念目前还是生物学中尚未完全解决的问题。在高等生物中,物种通常被看做是彼此杂交能繁殖的自然居群。由于原核生物缺乏严格意义的有性生殖,所以,原核生物很难确定“种”这个概念。
目前,一般讲DNA杂交同源性在70%以上,并且16S序列同源性达到97%以上的菌株定义为同一个种。
六、宏基因测序物种分类原理
当前使用的宏基因组物种分类方法,主要是使用 NCBI 的物种分类数据库进行分类。将测序数据与 NCBI 的序列数据库经行比对,例如 nt 库,nr 库,refseq 数据库等。如果测序数据与数据库中序列具有很好的相似性,而认为二者为同源序列,具有相同的祖先。将测序 ID 转换为 NCBI Accession ID,然后将 Accession ID 转换为物种分类 Tax ID。则得到了测序数据的物种分类信息。物种分类本质上则是根据序列相似性进行三种 ID 相互转换的过程。
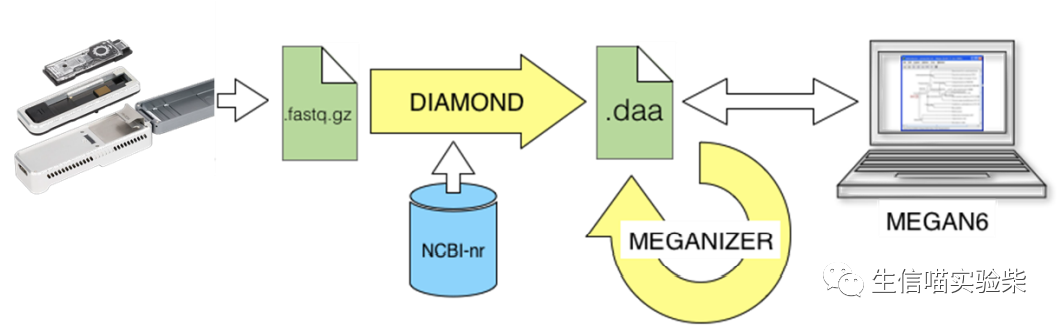
利用纳米孔测序进行快速鉴定示意图
三者之间的关系:测序的 ID 可以比对到多个 Accession ID,序列越长,唯一性越好,选择比对最好的 Accession ID。由于物种分类包括过个层级,一个 Accession 对应唯一 Taxon ID。一个 Taxon ID 中可以包含多个 Accession ID。每个 Taxon 还对应一个 parent taxid,对应一个 division id。
举例:
有一条序列,比对到了 Accession ID 是 NC_000001,NC_000001 对应的 Taxon ID 为 9606。9606 对应的 name 为 Homo sapiens,9606 的 parent tax_id 为 9605,对应 Homo。对应的division id 为 5,Primates 灵长类。
七、不同物种分类算法比较
LCA:“lowest common ancestor”,最小公共祖先法。
所谓 LCA,是当给定一个有根树 T 时,对于任意两个结点 u、v,找到一个离根最远的结点 x,使得 x 同时是 u 和 v 的祖先,x 便是 u、v 的最小公共祖先。
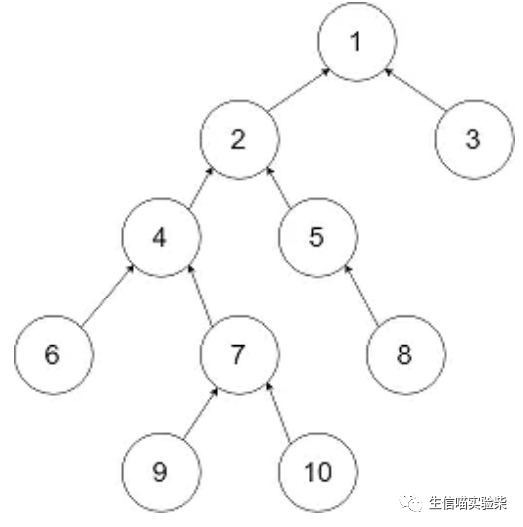
LCA 原理
对于这棵树来说 lca(9, 10) = 7, lac(6, 10) = 4, lac(3, 6) = 1,Kraken,Kraken 2,Opal,CLARK,与 MetaOthello 等软件是基于 kmer 的比对,利用 LCA 算法。
序列相似性:
相似性(similarity):是指所检测的序列与目标序列之间相同的碱基或氨基酸占整个序列的比例。相似性越高,同源性越高。但是需要注意,同源必须相似,但是相似不一定同源。
基于相似性比对的方法,可以比对全基因组序列,也可以比对 Marker 基因,例如 16S 等。blast,diamond,last,Megan,MetaPhlan,GASiC,MG-RAST 等软件基于序列相似性方法进行物种分类。基于相似性的缺点是比较速度较慢。
分类器:
基于机器学习的判别法进行分类,例如贝叶斯和 embase 估计 Bracken, MetaKallisto,Pathoscope;基于线性模型和混合线性模型分类 PhyloPythia,DiTASiC 和 MetaPalette;马尔科夫模型 Phymm/PhymmBL 基于支持向量机 PhyloPythia+,Burrows-Wheeler 转换,例如 Centrifuge。
八、biom 文件格式
biom(The Biological Observation Matrix)格式是宏基因组研究中最常用的结果保存格式,可将 OTU 或 Feature 表、样本属性、物种信息等多个表保存于同一个文件中,且格式统一,体积更小巧,目前被微生物组领域几乎所有主流软件所支持。biom 主要用来展示不同物种在不同样品中的丰度分布,类似与基因表达矩阵。如果该物种在某个样品中不存在,就是 0,与基因表达矩阵不同的是,不同样品中物种差别可能很大,所以会存在很多 0 的情况。通过biom 格式文件,方便不同软件之间相互调用,比如可以将 metaphlan 的结果导入 megan中查看。
支持 biom 格式的软件包括 QIIME,MG-RAST,PICRUSt,Mothur,phyloseq 包,MEGAN,VAMPS,metagenomeSeq,Phinch,RDP Classifier,USEARCH,PhyloToAST,EBI Metagenomics,GCModeller,MetaPhlAn 2 等。
官方网站:
http://biom-format.org/BIOM 目前分为 1.0 JSON 和 2.0 HDF5 两个版本;1.0 JSON 是编程语言广泛支持的格式,类似于散列的键值对结果。会根据数据松散程度,选择不同的存储结构来节省空间。2.0 HDF5是二进制格式,被许多程序语言支持,读取更高效和节约空间。
# 安装 Python 包
conda install biom-format # 2.1.7九、目前病原微生物鉴定中存在的技术问题
1、数据库信息不全,仍然有大量微生物没有被测序;
2、已有数据库准确性有待提高;
3、物种之间存在同源性,数据库冗余 ;
4、数据库过大,比对时间过长;
5、需要大量计算资源;
十、不同分析软件的比较
宏基因组物种鉴定软件有很多,但不同软件核心算法不同,使用的数据库不同,会有较大差别,且很难进行横向比较。
在比对算法上,有基于 blast 直接比对,也有使用 kmer 方法比对,有比对核酸数据库,也有比对氨基酸数据库。有使用 nt 库作为索引数据库,也有使用 marker gene 作为数据库,因此很难进行评估。
也有文章对于对于 20 种宏基因组物种鉴定工具进行横向比较。
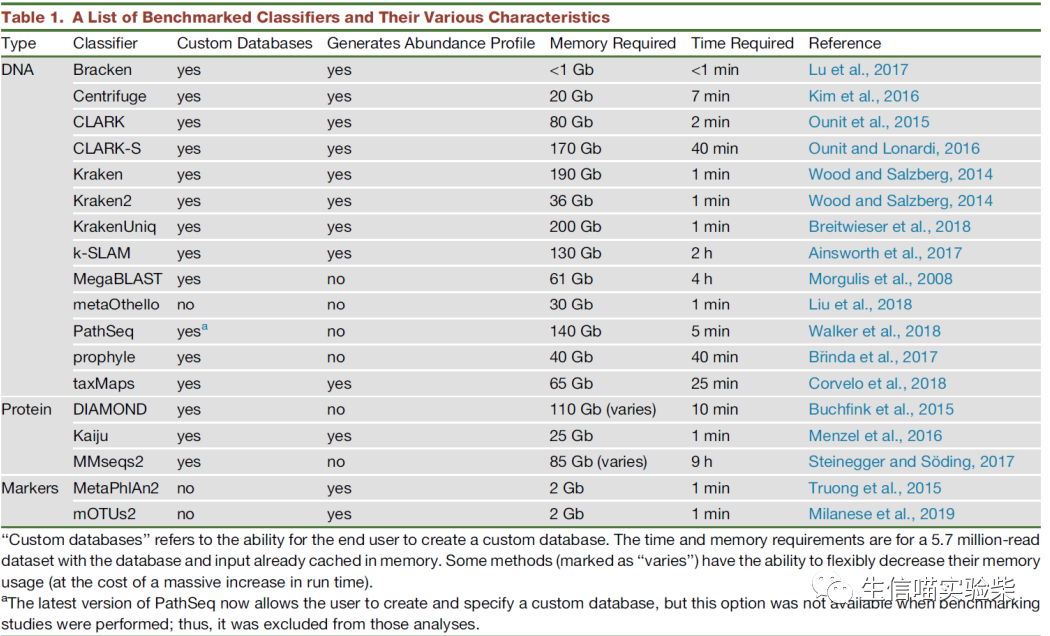
宏基因组分析方法比较
写在最后:有时间我们会努力更新的。大家互动交流可以前去论坛,地址在下面,复制去浏览器即可访问,弥补下公众号没有留言功能的缺憾。
bioinfoer.com有些板块也可以预设为大家日常趣事的分享等,欢迎大家来提建议。


