文章MSM_metagenomics(一):介绍
原创文章MSM_metagenomics(一):介绍
原创
生信学习者
修改于 2024-06-15 11:38:07
修改于 2024-06-15 11:38:07
欢迎大家关注全网生信学习者系列:
- WX公zhong号:生信学习者
- Xiao hong书:生信学习者
- 知hu:生信学习者
- CDSN:生信学习者2
介绍
用于复现Huang et al.研究分析的计算工作流程,所有复现数据和代码:生信学习者。
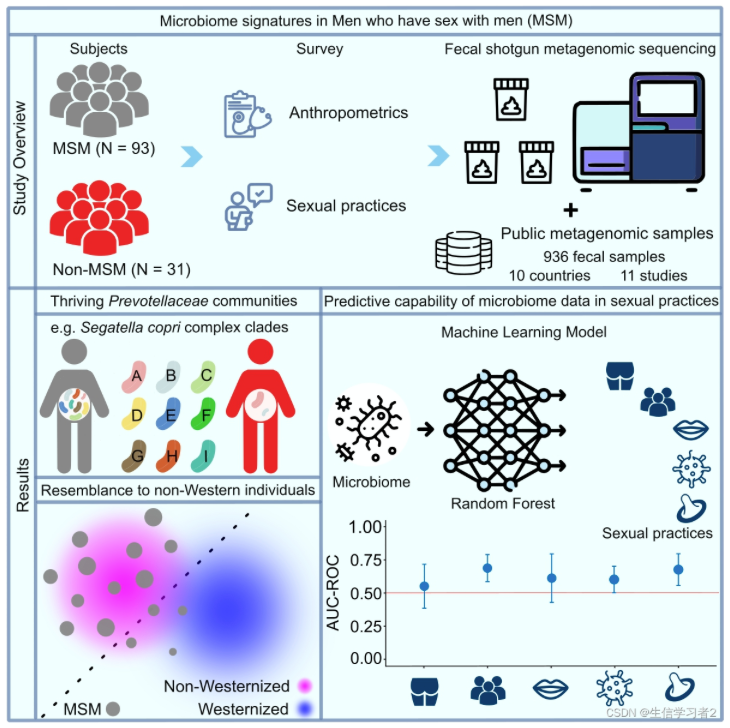
目录
- 累积分布函数Read质量检查:Reads quality inspection with cumulative distribution function
- Alpha多样性分析:Alpha diversity analysis
- Beta多样性分析:Beta diversity analysis
- 共现分析:Co-presence analysis
- 复杂热图绘制:ComplexHeatmap plotting
- 分组马赛克图:Make mosaic plot
- 机器学习分析:Machine learning analysis
- 偏差图、UpSet图、棒棒糖图:Deviation plot, UpSet plot, Lollipop plot
数据
大家通过以下链接下载数据:
- 百度网盘链接:https://pan.baidu.com/s/1f1SyyvRfpNVO3sLYEblz1A
- 提取码: WX公zhong号生信学习者后台发送 复现msm 获取提取码
用法
提供了两种类型的实用工具:1) Python脚本;2) 可导入的R函数。
- Python脚本是封装好的代码,用于执行特定的分析。
- 可导入的R函数是包装好的R代码,用于解决特定问题,只需导入它们的脚本即可重用
可视化R包
可视化用到的R包
文章MSM_metagenomics(二):累积分布函数Read质量检查
介绍
本教程介绍如何使用累积分布函数来检查宏基因组测序Reads的质量,特别是检查不同组样本中是否存在测序偏差。
数据
大家通过以下链接下载数据:
- 百度网盘链接:https://pan.baidu.com/s/1f1SyyvRfpNVO3sLYEblz1A
- 提取码: WX公zhong号生信学习者后台发送 复现msm 获取提取码
Python 包
绘制Reads计数的累积分布函数
您将使用一个Python脚本cumulative_distribution_function.py,以及一个包含每个宏基因组样本中读取数量的表格,例如./data/example_reads_stats.tsv。
cumulative_distribution_function.py代码
- usage of
cumulative_distribution_function.py
示例命令 1:
示例命令 2:
如果您希望为不同的变量类别指定特定的颜色,例如将灰色指定给MSM(男男性行为者),将红色指定给Non-MSM(非男男性行为者),您可以使用一个颜色调色板映射,如./data/reads_stats_color_map.tsv。

原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录
