单细胞4
原创1 下载并整理数据
跟上此一样,只不过下载全部数据,不用自定义。
确实网速老慢,下载的花花老师分享的文件。应该先清空列表台,再解压,忘了就顺序换了一下。要注意一个问题,要在工作目录条件下。
> untar("GSE231920_RAW.tar",exdir = "GSE231920_RAW")#解包
> library(stringr)
> fs = paste0("GSE231920_RAW/",dir("GSE231920_RAW/"))
> fs#每个样本的数据文件
[1] "GSE231920_RAW/GSM7306054_sample1_barcodes.tsv.gz" "GSE231920_RAW/GSM7306054_sample1_features.tsv.gz" "GSE231920_RAW/GSM7306054_sample1_matrix.mtx.gz"
[4] "GSE231920_RAW/GSM7306055_sample2_barcodes.tsv.gz" "GSE231920_RAW/GSM7306055_sample2_features.tsv.gz" "GSE231920_RAW/GSM7306055_sample2_matrix.mtx.gz"
[7] "GSE231920_RAW/GSM7306056_sample3_barcodes.tsv.gz" "GSE231920_RAW/GSM7306056_sample3_features.tsv.gz" "GSE231920_RAW/GSM7306056_sample3_matrix.mtx.gz"
[10] "GSE231920_RAW/GSM7306057_sample4_barcodes.tsv.gz" "GSE231920_RAW/GSM7306057_sample4_features.tsv.gz" "GSE231920_RAW/GSM7306057_sample4_matrix.mtx.gz"
[13] "GSE231920_RAW/GSM7306058_sample5_barcodes.tsv.gz" "GSE231920_RAW/GSM7306058_sample5_features.tsv.gz" "GSE231920_RAW/GSM7306058_sample5_matrix.mtx.gz"
[16] "GSE231920_RAW/GSM7306059_sample6_barcodes.tsv.gz" "GSE231920_RAW/GSM7306059_sample6_features.tsv.gz" "GSE231920_RAW/GSM7306059_sample6_matrix.mtx.gz"
> rm(list = ls()) #清空列表台
> fs = paste0("GSE231920_RAW/",dir("GSE231920_RAW/"))
> fs#每个样本的数据文件
[1] "GSE231920_RAW/GSM7306054_sample1_barcodes.tsv.gz" "GSE231920_RAW/GSM7306054_sample1_features.tsv.gz" "GSE231920_RAW/GSM7306054_sample1_matrix.mtx.gz"
[4] "GSE231920_RAW/GSM7306055_sample2_barcodes.tsv.gz" "GSE231920_RAW/GSM7306055_sample2_features.tsv.gz" "GSE231920_RAW/GSM7306055_sample2_matrix.mtx.gz"
[7] "GSE231920_RAW/GSM7306056_sample3_barcodes.tsv.gz" "GSE231920_RAW/GSM7306056_sample3_features.tsv.gz" "GSE231920_RAW/GSM7306056_sample3_matrix.mtx.gz"
[10] "GSE231920_RAW/GSM7306057_sample4_barcodes.tsv.gz" "GSE231920_RAW/GSM7306057_sample4_features.tsv.gz" "GSE231920_RAW/GSM7306057_sample4_matrix.mtx.gz"
[13] "GSE231920_RAW/GSM7306058_sample5_barcodes.tsv.gz" "GSE231920_RAW/GSM7306058_sample5_features.tsv.gz" "GSE231920_RAW/GSM7306058_sample5_matrix.mtx.gz"
[16] "GSE231920_RAW/GSM7306059_sample6_barcodes.tsv.gz" "GSE231920_RAW/GSM7306059_sample6_features.tsv.gz" "GSE231920_RAW/GSM7306059_sample6_matrix.mtx.gz"
> 花花老师是18个,我的也是18个,后面没有编号而已
> samples = dir("GSE231920_RAW/") %>% str_split_i(pattern = "_",i = 2) %>% unique();samples
[1] "sample1" "sample2" "sample3" "sample4" "sample5" "sample6"这个例子是:用strsplit_i把文件名称按照""作为分隔符(pattern = "_")分成多个部分(比如GSM7306054_sample1_barcodes.tsv.gz被分成GSM7306054、sample1、barcodes.tsv.gz三个部分),我们需要的是第2个部分(i=2),即sample1
1.1 R语言基础知识补充,字符串处理函数
str_sub()
> ?str_sub() # 用法str_sub(string, start = 1L, end = -1L),提取字符串,string原始字符串,start开始提取位置,end结束位置,omit_na = FALSE(这是默认设置),这意味着如果提取的子字符串因为某些原因(比如索引超出了原始字符串的长度)导致结果为NA,那么这个NA值将被保留,不会被省略。
> ?str_sub_all #str_sub_all(string, start = 1L, end = -1L),string可能是一个字符串向量 eg
> a = c("11223344","22334455","33445566")
> str_sub_all(a,start = 2,end = 5)
[[1]]
[1] "1223"
[[2]]
[1] "2334"
[[3]]
[1] "3445"str_remove
> ?str_remove()
> b = "a 111 b2322 c6"
> clean_b = str_remove(b, "\\d+")
>
> clean_b
[1] "a b2322 c6"
> b = "a 111 2 b2322 c6"
> clean_b = str_remove(b, "\\d+")
> clean_b #只有111和2属于数字
[1] "a 2 b2322 c6"
>
> clean_b #只有111和2属于数字,all同理
[1] "a 2 b2322 c6"str_detect()
> ?str_detect()
> ?str_detect() #一个检测字符串str_detect(string, pattern, negate = FALSE),一个逻辑值,指定是否要反转匹配逻辑。如果设置为TRUE,则函数会检测不匹配的字符串。
> d <- c("apple", "banana", "cherry", "date")
> contains_a <- str_detect(d, "a")
> contrains_a
Error: object 'contrains_a' not found
> contains_a
[1] TRUE TRUE FALSE TRUE
> fcontains_a <- str_detect(d, "a",negate = TRUE)
> fcontains_a
[1] FALSE FALSE TRUE FALSEstr_replace
> ?str_replace()
> ?str_replace() #string:需要进行替换操作的原始字符串或字符串向量。pattern:要替换的模式,可以是一个正则表达式。replacement:用于替换匹配模式的字符串。
> my_string <- "The dates are 2023-06-22 and 2024-06-22"
> replaced_string <- str_replace(my_string, "\\d+", "-")
> print(replaced_string)
[1] "The dates are --06-22 and 2024-06-22"
>
> replaced_string <- str_replace_all(my_string, "\\d+", "-")
> replaced_string
[1] "The dates are ----- and -----"
> 1.2 R 语言基础知识补充---文件处理函数
> dir() # 列出工作目录下的文件
[1] "GSE231920_RAW" "GSE231920_RAW.tar" "input"
[4] "install.R" "markers.Rdata" "obj.Rdata"
[7] "ref_Human_all.RData" "run1.R" "runmany.R"
[10] "sce.all.Rdata" "single cell.Rproj" "single_ref"
> dir() # 列出工作目录下的文件,会列出所有文件
[1] "GSE231920_RAW" "GSE231920_RAW.tar" "input"
[4] "install.R" "markers.Rdata" "obj.Rdata"
[7] "ref_Human_all.RData" "run1.R" "runmany.R"
[10] "sce.all.Rdata" "single cell.Rproj" "single_ref"
> dir(pattern = ".R$") #列出工作目录下以.R结尾的文件 $表示以……结尾
[1] "install.R" "run1.R" "runmany.R"
> dir(pattern = ".RData$") #列出工作目录下以.RData结尾的文件 $表示以……结尾
[1] "ref_Human_all.RData"
> dir(pattern = ".RData$") #列出工作目录下以.RData结尾的文件 $表示以……结尾,大小写也要注意
[1] "ref_Human_all.RData"
> dir(pattern = ".R")
[1] "GSE231920_RAW" "GSE231920_RAW.tar" "install.R"
[4] "markers.Rdata" "obj.Rdata" "ref_Human_all.RData"
[7] "run1.R" "runmany.R" "sce.all.Rdata"
[10] "single cell.Rproj"
> dir(pattern = ".R") #查找带有R的文件,而不是以R结尾的文件
[1] "GSE231920_RAW" "GSE231920_RAW.tar" "install.R"
[4] "markers.Rdata" "obj.Rdata" "ref_Human_all.RData"
[7] "run1.R" "runmany.R" "sce.all.Rdata"
[10] "single cell.Rproj"
> file.create("douhua.txt") #用代码创建文件
[1] TRUE
> file.create("1.txt") #用代码创建文件1
[1] TRUE
> file.exists("1.txt") #检查1.txt文件在工作目录下是否存在
[1] TRUE
> file.remove("douhua.txt") #用代码删除文件
[1] TRUE
> file.remove("1.txt") #用代码删除文件
[1] TRUE
> file.exists("douhua.txt") #删掉了就不存在啦
[1] FALSE
> f = paste0("douhua",1:10,".txt")#"douhua":这是要连接的第一个字符串部分。1:10:表示从1到10的整数。".txt":这是要连接的最后一个字符串部分,通常用于表示文件扩展名
> file.create(f)
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
> file.create(f) #批量创建
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
> file.remove(f)# 批量删除
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
> file.exists(f) #批量检查
[1] FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE FALSE1.3 R语言基础知识补充,lapply(据说超级无敌牛)
> lapply(1:4, print) #把1-4分被带入到print函数中,但是lapply本身返回的是一个列表,列表中的每个元素对应于原始向量中每个元素经过print函数处理的结果
[1] 1
[1] 2
[1] 3
[1] 4
[[1]]
[1] 1
[[2]]
[1] 2
[[3]]
[1] 3
[[4]]
[1] 4
> numbers = 1:5 # 创建一个数字向量
> squared_numbers = lapply(numbers, function(x) x^2) # 使用lapply对每个元素求平方
> print(squared_numbers)# 查看结果
[[1]]
[1] 1
[[2]]
[1] 4
[[3]]
[1] 9
[[4]]
[1] 16
[[5]]
[1] 25
> sums = lapply(numbers, sum) #使用内置函数sum
> sums
[[1]]
[1] 1
[[2]]
[1] 2
[[3]]
[1] 3
[[4]]
[1] 4
[[5]]
[1] 5lapply的输出结果里面有1是因为把结果放进了一个列表里,1表示列表的第一个元素,2是第二个元素,以此类推。确实有点牛
1.4 自定义函数
R语言可以自定义函数,我之前一定是学过,但是忘了,加深一下印象
> my_install = function(pkg){
+ if (!require(pkg,character.only = T))install.packages(pkg,update = F,ask = F)
+ }
> my_install = function(pkg){
+ if (!require(pkg,character.only = T))install.packages(pkg,update = F,ask = F)
+ } # my_install,函数作者自定义的,参数的名字是pkg,就是你自定义以后,你的自定义函数后面接的哪个参数会放在pkg的位置
> my_install("tidyr") #把”tidyr”代入到大括号里的pkg位置
Loading required package: tidyr
> my_install("tidyr") #把”tidyr”代入到大括号里的pkg位置,相当于运行了if (!require("tidyr",character.only = T))install.packages("tidyr",update = F,ask = F)
>
> my_install("tidyr") #把”tidyr”代入到大括号里的pkg位置,相当于运行了if (!require("tidyr",character.only = T))install.packages("tidyr",update = F,ask = F)
>
> my_install("tidyr") #把”tidyr”代入到大括号里的pkg位置,相当于运行了if (!require("tidyr",character.only = T))install.packages("tidyr",update = F,ask = F) #所以加入你要装很多包,除了用if和for循环联用的函数以外还可以这样减少劳动量
> my_install("tidyr") #把”tidyr”代入到大括号里的pkg位置,相当于运行了if (!require("tidyr",character.only = T))install.packages("tidyr",update = F,ask = F) #所以加入你要装很多包,除了用if和for循环联用的函数以外还可以这样减少劳动量,NULL,这里是在装包,没有产生任何数据结果,所以就NULL(NULL就是什么都没有的意思)。
#还要说明一下,自定义函数可以和lapply联用就更加整洁了,不亚于if和for循环sum2 = function(pkg){(pkg +1)/2} #很好理解(pkg +1)/2
sum2(3)1.5 需要把文件整理成为想要的格式,整理成Read10X要求的格式
有多个样本,就要每个样本单独一个文件夹,每个文件夹里3个固定名称的文件,不可以有前缀的。这个在之前说过的这个问题
接下来做的工作有:
1.为每个样本创建单独的文件夹
2.把每个样本的三个文件复制进去
3.所有文件改名,去掉前缀,成为前面说过的固定的名字
###1.5.1 为每个样本创建单独的文件夹
这里用到lapply套自定义函数实现了批量操作。
> ctr = function(s){ #使用 paste0 函数将字符串 "01_data/" 和参数 s 拼接起来,形成完整的目录路径
+ ns = paste0("01_data/",s) #我刚才理解的不太对,应该是新建一个01_data,在该路径下在新建文件夹
+ if(!file.exists(ns))dir.create(ns,recursive = T) # 检查ns生成的路径是否存在,不存在的话就用dir.create建一个
+ }
> lapply(samples, ctr) # 创建的ctr这个功能函数应用到lapply,批量新建文件夹get
[[1]]
[1] TRUE
[[2]]
[1] TRUE
[[3]]
[1] TRUE
[[4]]
[1] TRUE
[[5]]
[1] TRUE
[[6]]
[1] TRUE新建成功
###1.5.2 每个样本三个文件复制在单独的文件夹中
> lapply(fs, function(s){ #自定义函数直接加到lapply第二个位置上了
+ #s = fs[1]
+ for(i in 1:length(samples)){ #for循环
+ #i = 1
+ if(str_detect(s,samples[[i]])){ #检查s中是否有samplei
+ file.copy(s,paste0("01_data/",samples[[i]])) # 复制文件,
+ }
+ }
+ })
[[1]]
NULL
[[2]]
NULL
[[3]]
NULL
[[4]]
NULL
[[5]]
NULL
[[6]]
NULL
[[7]]
NULL
[[8]]
NULL
[[9]]
NULL
[[10]]
NULL
[[11]]
NULL
[[12]]
NULL
[[13]]
NULL
[[14]]
NULL
[[15]]
NULL
[[16]]
NULL
[[17]]
NULL
[[18]]
NULL代码里面有#s = fs1和#i = 1是因为写函数的过程是需要调试的,不是一遍写成功的。所以先把一个具体的值代入进去,把函数的代码调试成功再批量操作。代数调试的操作只在写函数时用到,用完打上#变成注释,函数在运行时就不会执行#号后面的代码了。这个问题确实思考了一下
fs是啥有点忘了
1.5.3 文件改名,去掉前缀
> on = paste0("01_data/",dir("01_data/",recursive = T));on
[1] "01_data/sample1/GSM7306054_sample1_barcodes.tsv.gz"
[2] "01_data/sample1/GSM7306054_sample1_features.tsv.gz"
[3] "01_data/sample1/GSM7306054_sample1_matrix.mtx.gz"
[4] "01_data/sample2/GSM7306055_sample2_barcodes.tsv.gz"
[5] "01_data/sample2/GSM7306055_sample2_features.tsv.gz"
[6] "01_data/sample2/GSM7306055_sample2_matrix.mtx.gz"
[7] "01_data/sample3/GSM7306056_sample3_barcodes.tsv.gz"
[8] "01_data/sample3/GSM7306056_sample3_features.tsv.gz"
[9] "01_data/sample3/GSM7306056_sample3_matrix.mtx.gz"
[10] "01_data/sample4/GSM7306057_sample4_barcodes.tsv.gz"
[11] "01_data/sample4/GSM7306057_sample4_features.tsv.gz"
[12] "01_data/sample4/GSM7306057_sample4_matrix.mtx.gz"
[13] "01_data/sample5/GSM7306058_sample5_barcodes.tsv.gz"
[14] "01_data/sample5/GSM7306058_sample5_features.tsv.gz"
[15] "01_data/sample5/GSM7306058_sample5_matrix.mtx.gz"
[16] "01_data/sample6/GSM7306059_sample6_barcodes.tsv.gz"
[17] "01_data/sample6/GSM7306059_sample6_features.tsv.gz"
[18] "01_data/sample6/GSM7306059_sample6_matrix.mtx.gz"
> nn = str_remove(on,"GSM\\d+_sample\\d_");nn #把想要改的名字储存在nn这个变量中,把on中的GSM\\d+_sample\\d_这部分内容删掉了
[1] "01_data/sample1/barcodes.tsv.gz" "01_data/sample1/features.tsv.gz"
[3] "01_data/sample1/matrix.mtx.gz" "01_data/sample2/barcodes.tsv.gz"
[5] "01_data/sample2/features.tsv.gz" "01_data/sample2/matrix.mtx.gz"
[7] "01_data/sample3/barcodes.tsv.gz" "01_data/sample3/features.tsv.gz"
[9] "01_data/sample3/matrix.mtx.gz" "01_data/sample4/barcodes.tsv.gz"
[11] "01_data/sample4/features.tsv.gz" "01_data/sample4/matrix.mtx.gz"
[13] "01_data/sample5/barcodes.tsv.gz" "01_data/sample5/features.tsv.gz"
[15] "01_data/sample5/matrix.mtx.gz" "01_data/sample6/barcodes.tsv.gz"
[17] "01_data/sample6/features.tsv.gz" "01_data/sample6/matrix.mtx.gz"
> file.rename(on,nn) #把on换一下名字,因为是一对一生成的向量,所以是一对一的
[1] TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE TRUE
[18] TRUE注意一下路径是从工作目录开始的,d是正则表达式里的任意数字,+代表匹配一次或多次,所以就会把所有的前缀删掉。换不同数据时需要自行改动。这块老师说可以用ai帮忙写,自己检查就i行了。
2批量读取
本次测试中用的抽样,注意实战可不能抽样
哭泣,我的电脑是32g内存,还是老老实实的用花花老师保存的抽样吧,看来还是需要更新电脑
rm(list = ls()) #清空控制台
library(Seurat) # 加载这个包
rdaf = "sce.all.Rdata" #赋值
if(!file.exists(rdaf)){ #检查文件是否存在,不存在就创建
f = dir("01_data/")
scelist = list() #创建空的列表,下面的for循环每执行一次,scelist里面就会多一个元素。
for(i in 1:length(f)){
pda <- Read10X(paste0("01_data/",f[[i]]))
scelist[[i]] <- CreateSeuratObject(counts = pda,
project = f[[i]],
min.cells = 3,
min.features = 200)
print(dim(scelist[[i]]))#输出每个文件的基因数和细胞数
}
sce.all = merge(scelist[[1]],scelist[-1]) #合并多个对象
sce.all = JoinLayers(sce.all)
#merge后,每个样本的表达矩阵是一个独立的的layer,JoinLayers是合并为一个表达矩阵
set.seed(335)
sce.all = subset(sce.all,downsample=700)#每个样本抽700个细胞
save(sce.all,file = rdaf)
}
load(rdaf)
head(sce.all@meta.data)
## orig.ident nCount_RNA nFeature_RNA
## AAAGAACAGTCTGTAC-1_1 sample1 3884 1377
## AAAGGGCTCTCGCGTT-1_1 sample1 1615 721
## AAAGGTACAATTGGTC-1_1 sample1 3806 1215
## AAAGTGAGTATCGAAA-1_1 sample1 5554 1456
## AAAGTGATCTCAACCC-1_1 sample1 3111 1328
## AACAAAGGTAAGGTCG-1_1 sample1 3658 1243
table(sce.all$orig.ident) #每个样本多少细胞
##
## sample1 sample2 sample3 sample4 sample5 sample6
## 700 700 700 700 700 700
sum(table(Idents(sce.all)))#细胞总数
## [1] 42003质控指标
在进行质控过程中,线粒体基因常常被过滤掉,除了线粒体基因核糖体和红细胞基因也是常见的过滤指标
线粒体过滤原因:线粒体基因在细胞中的拷贝数远高于核基因,这可能导致它们在测序数据中过度表达,从而掩盖其他核基因的表达模式。但是有些测量线粒体表达离群值太大或者太小那肯定不对,需要删除这样的。
核糖体过滤基因:核糖体基因负责编码核糖体RNA,核糖体是蛋白质合成的场所。由于蛋白质合成是细胞的基本功能,核糖体基因通常在所有细胞中都有很高的表达量。这样对核基因有很大的影响。但是有些测量核糖体表达离群值太大或者太小那肯定不对,需要删除这样的。
红细胞基因:在某些情况下,红细胞基因可能在特定类型的细胞(如红细胞)中高度表达,这可能会影响对其他细胞类型基因表达的分析。但是有些测量红细胞基因表达离群值太大或者太小那肯定不对,需要删除这样的。
为什么要过滤呢?过滤掉离群值大的,保留下来的才是有用的,可以认为是正确的
> load("G:/Document/R study/single cell/single cell/sce.all.Rdata")
> sce.all[["percent.rp"]] <- PercentageFeatureSet(sce.all, pattern = "^RP[SL]")
> load("sce.all.Rdata") #一定要记得加载一下,不加载找不到文件
> sce.all[["percent.mt"]] <- PercentageFeatureSet(sce.all, pattern = "^MT-")
> sce.all[["percent.rp"]] <- PercentageFeatureSet(sce.all, pattern = "^RP[SL]")
> sce.all[["percent.hb"]] <- PercentageFeatureSet(sce.all, pattern = "^HB[^(P)]")
> head(sce.all@meta.data, 3)
orig.ident nCount_RNA nFeature_RNA percent.mt percent.rp percent.hb
AAAGAACAGTCTGTAC-1_1 sample1 3884 1377 4.763131 23.09475 0
AAAGGGCTCTCGCGTT-1_1 sample1 1615 721 0.619195 35.85139 0
AAAGGTACAATTGGTC-1_1 sample1 3806 1215 7.777194 37.12559 0
> VlnPlot(sce.all, # 画出小提琴图,确定过滤范围
+ features = c("nFeature_RNA",
+ "nCount_RNA",
+ "percent.mt",
+ "percent.rp",
+ "percent.hb"),
+ ncol = 3,pt.size = 0.5, group.by = "orig.ident")
Warning: Default search for "data" layer in "RNA" assay yielded no results; utilizing "counts" layer instead. #出现这个警告意思是列表的层没有data,所以用元数据化的。一般的警告不是报错就可以忽略不计。sce.all"percent.mt" <- PercentageFeatureSet(sce.all, pattern = "^MT-") PercentageFeatureSet 函数用于计算一组特定特征(这里是由"^MT-"模式匹配的线粒体基因)在每个细胞中的相对表达量。pattern = "^MT-" 指定了一个正则表达式,用来匹配所有以"MT-"开头的基因名,这些通常表示线粒体基因。
pattern = "^RPSL" 使用正则表达式匹配以"RP"开头后跟"S"或"L"的基因名,这些通常表示核糖体蛋白基因。
pattern = "^HB^(P)" 使用正则表达式匹配以"HB"开头但不紧跟"P"的基因名,这些通常表示血红蛋白基因,但不包括某些特定的亚型。
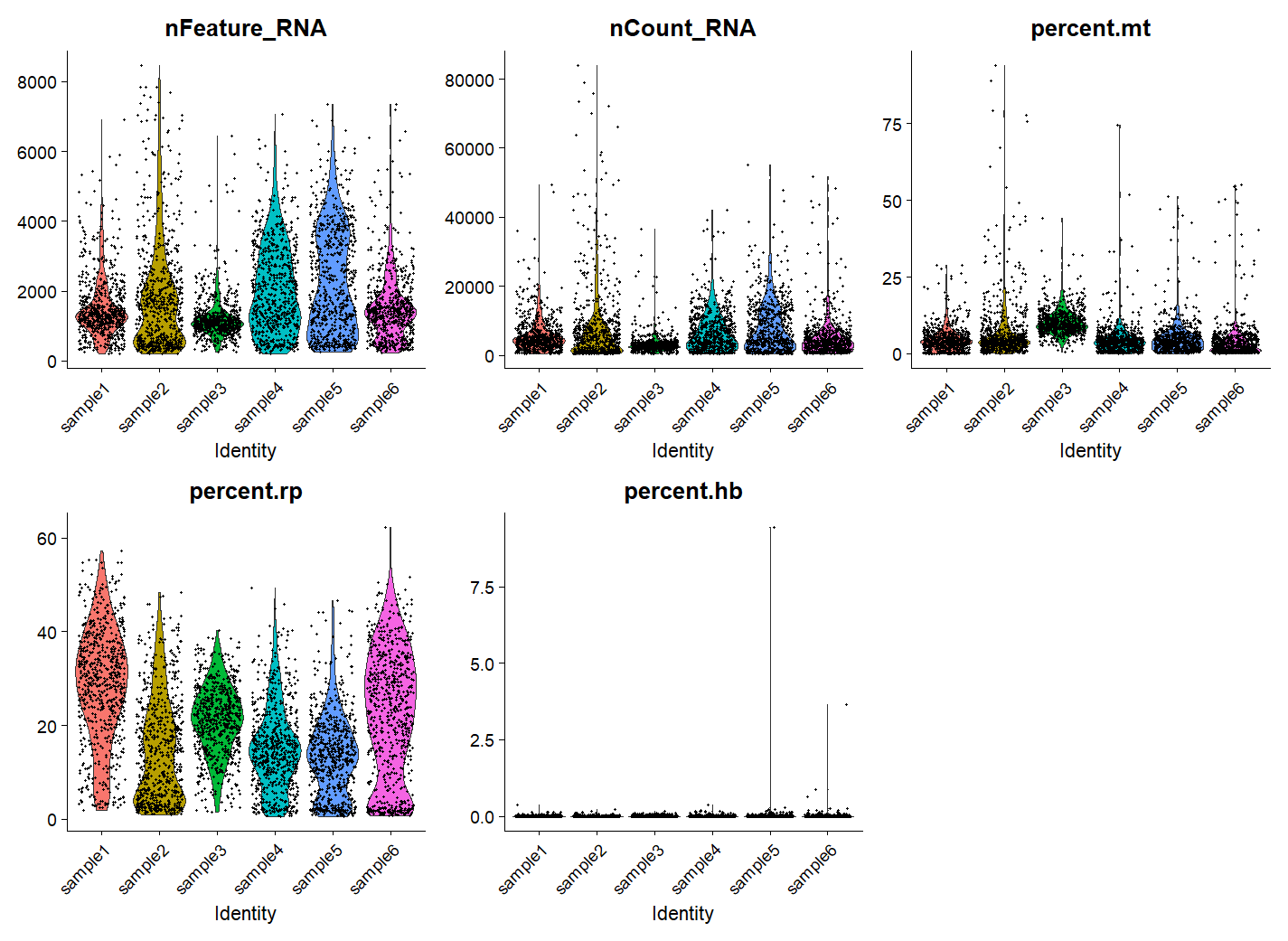
> sce.all = subset(sce.all,percent.mt < 20& #根据小提琴图确定过滤范围nFeature_RNA < 6000,nCount_RNA < 40000,percent.mt < 20,还可以在加上一个percent.hb<1
+ nCount_RNA < 40000 &
+ nFeature_RNA < 6000 &
+ percent.hb < 1)
> VlnPlot(sce.all,
+ features = c("nFeature_RNA",#重新画一个小提琴图,看看还有没有尖尖的线了
+ "nCount_RNA",
+ "percent.mt",
+ "percent.rp",
+ "percent.hb"),
+ ncol = 3,pt.size = 0.5, group.by = "orig.ident")
Warning: Default search for "data" layer in "RNA" assay yielded no results; utilizing "counts" layer instead.
> table(sce.all@meta.data$orig.ident)
sample1 sample2 sample3 sample4 sample5 sample6
687 622 683 686 677 674 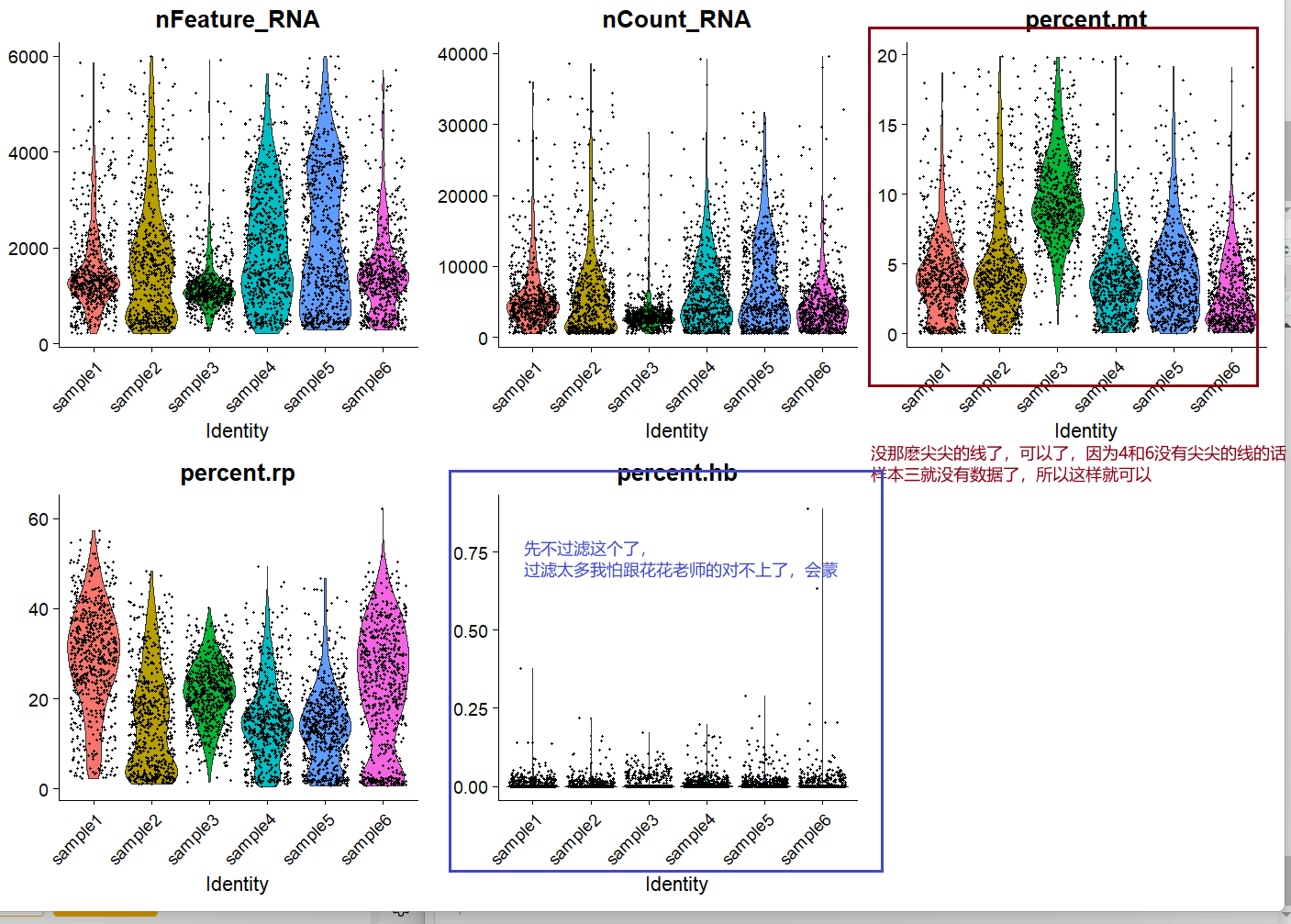
画了核糖体,但是没有用这个,轻微的用了一下红细胞基因
4 整合降维聚类分群
整合:单细胞数据集可能来自不同的实验批次或平台,这些批次效应(batch effects)可能导致数据在分析时出现偏差。整合的目的是将来自不同批次的数据调整到一个共同的参考框架中,以减少批次效应,提高数据的可比性
降维:之前写过。将高维数据映射到低维空间中
聚类分群:聚类分群的目的是将相似的细胞聚集在一起,形成不同的细胞群体或亚群,这些群体可能代表不同的细胞类型或状态。
通过这些分析可能能发现新的细胞类型,揭示细胞之间的欢喜等,同时可以减少噪声和批次效应。
多样本整合:使用harmony,它需要的计算资源少,且准确程度高,是最受欢迎的方法。
f = "obj.Rdata" #19号有一个这个,为了不跟这次的混,多以把19号那个重命名一下
install.packages("harmony")
library(harmony)
if(!file.exists(f)){
sce.all = sce.all %>%
NormalizeData() %>%
FindVariableFeatures() %>%
ScaleData(features = rownames(.)) %>%
RunPCA(pc.genes = VariableFeatures(.)) %>%
RunHarmony("orig.ident") %>%
FindNeighbors(dims = 1:15, reduction = "harmony") %>%
FindClusters(resolution = 0.5) %>%
RunUMAP(dims = 1:15,reduction = "harmony") %>%
RunTSNE(dims = 1:15,reduction = "harmony")
save(sce.all,file = f) # f 是一个变量,它应该包含了文件的路径和名称,这样就会重新生成一个f啦
}
load(f)
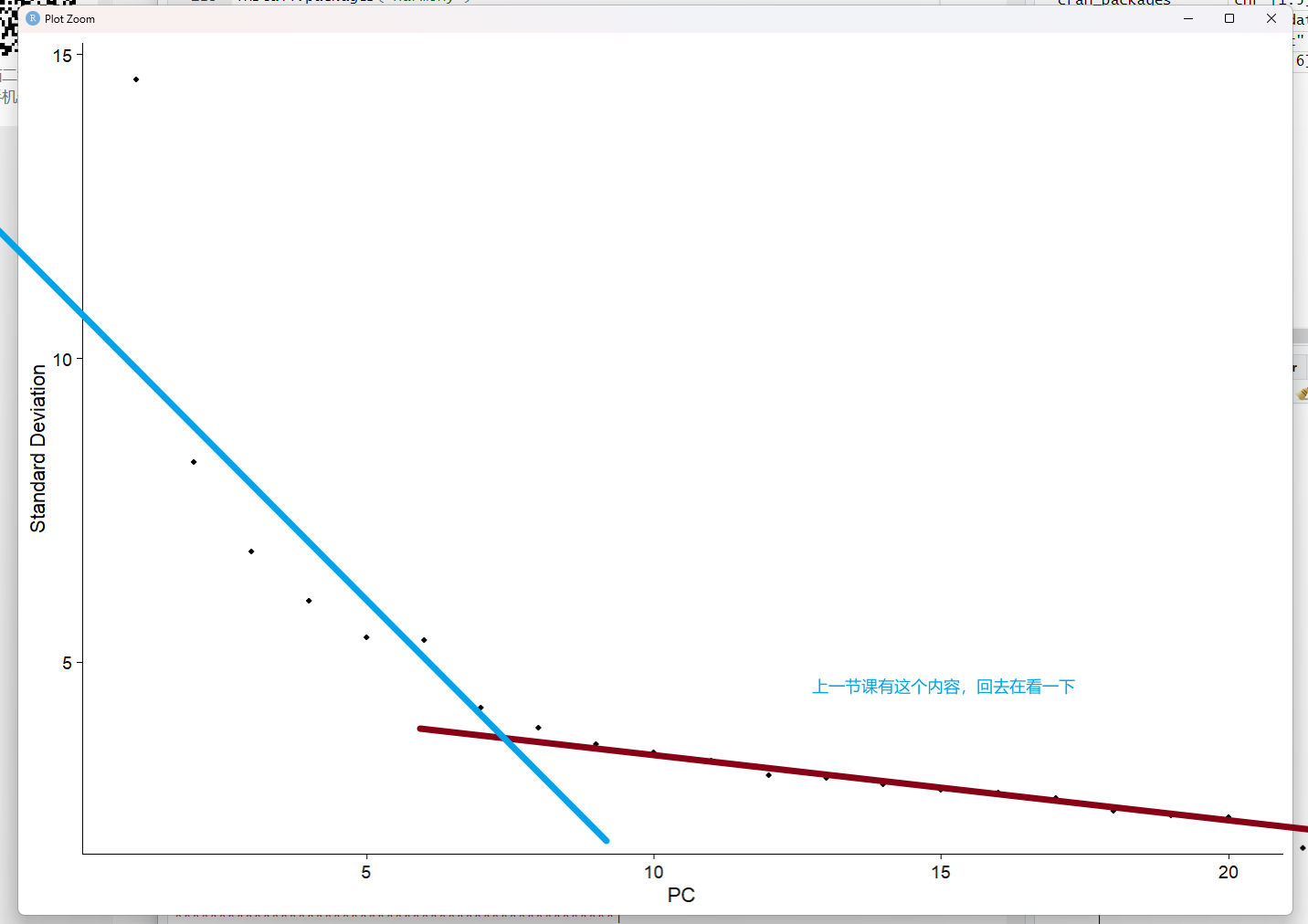
ElbowPlot(sce.all) #画了一个ElbowPlot,可以用来确定上面的拐点对不对一个是dims = 1:15,代表选择前15个维度,15是个比较折中的值,选择的数量越多计算量越大;选的太少又不能代表所有的数据。一般是10-20,根据ElbowPlot来选择拐点的横坐标(拐点就是在这个点之前纵坐标下降比较快,在这个点之后纵坐标下降比较慢)。不是很重要,只要拐点在15之前,选15就一点问题都没有。一个是resolution = 0.5,代表分群的分辨率是0.5,这个参数的选择范围是0.1-1.5之间,数值越大分出来的群越多,数值越小分出来的点越少。0.5也是一个比较折中的值,如果后面注释不困难,就不用改动
第三天课程的内容在复习一下。
> p1 = DimPlot(sce.all, reduction = "umap",label = T,pt.size = 0.5)+ NoLegend();p1 #reduction = "umap":这个参数指定了用于绘制散点图的降维技术用的是umap,label = T 或 label = TRUE:这个参数告诉 DimPlot 函数在散点图上为每个点添加标签。pt.size = 0.5:这个参数控制散点图中点的大小。NoLegend():这是Seurat包中的一个函数,用于移除图表上的图例。跟花花老师的不太一样,多了一个,反回去看看是不是因为多扣了俩点的原因,虽然我觉得应该不是,猜测应该只之前说的更新的问题。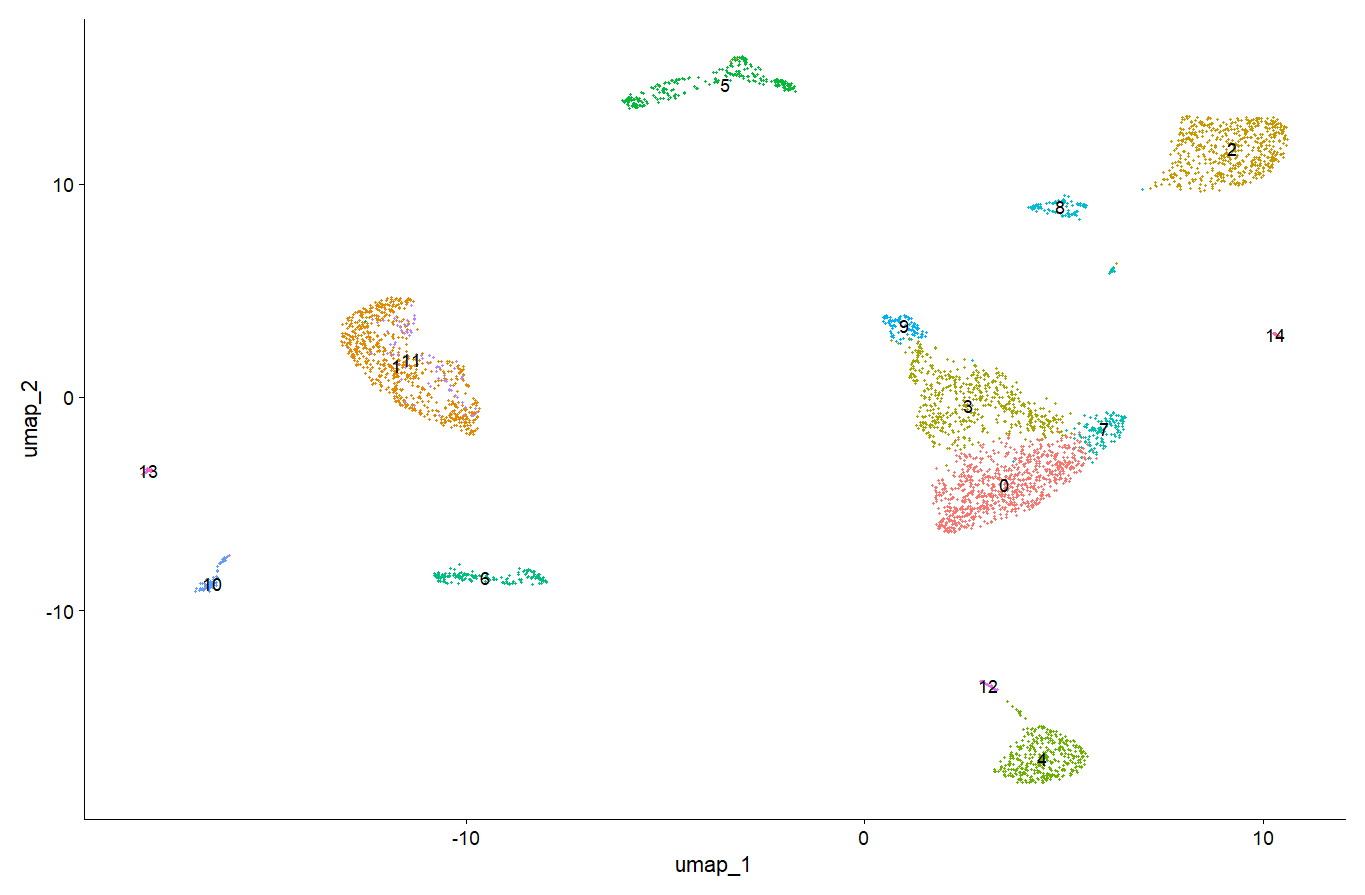
继续往下做

DimPlot(sce.all, reduction = "umap",pt.size = 0.5,group.by = "orig.ident")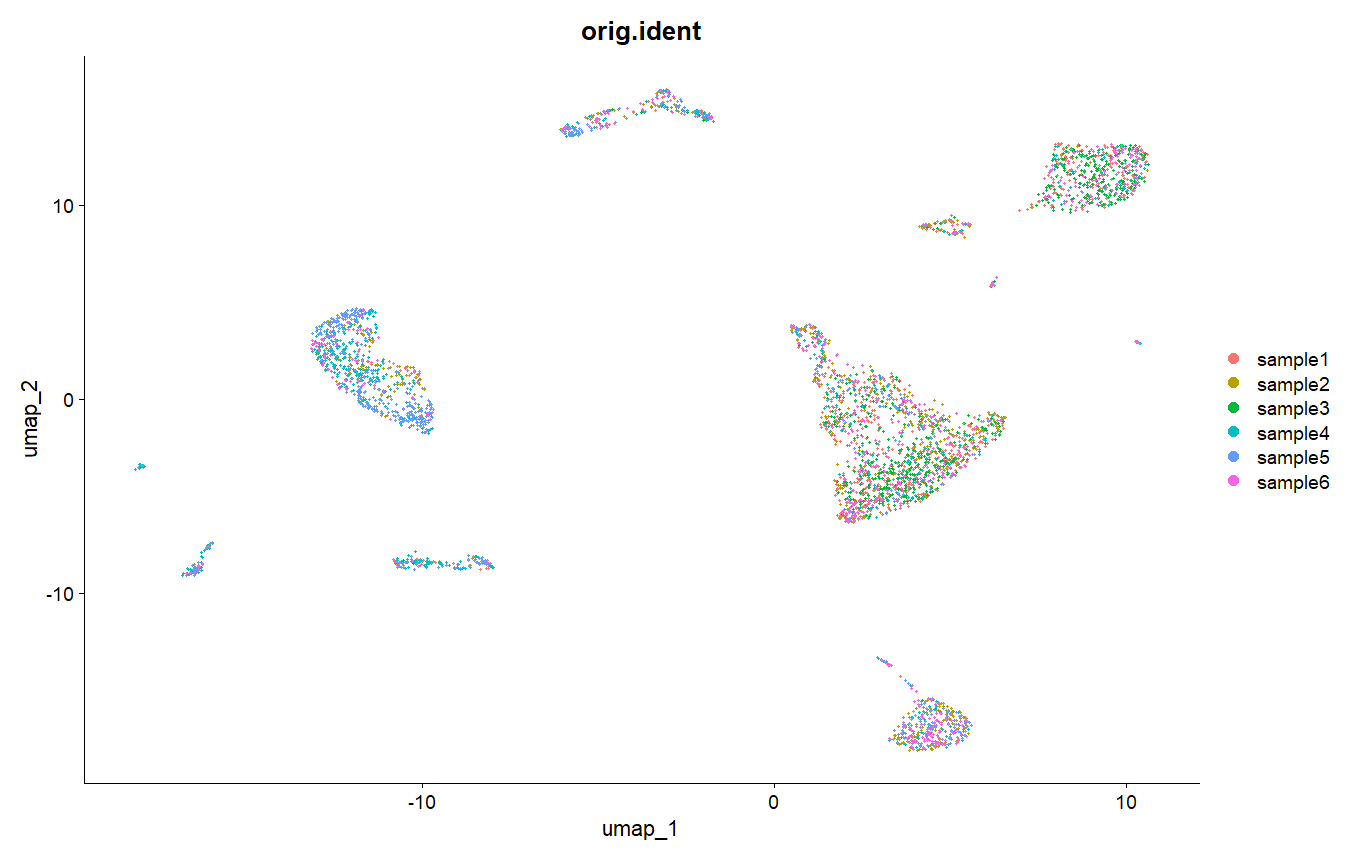
group.by = "orig.ident"就会按照样本来分配颜色,在umap图上面看,这些样本几乎都是分为这几个簇,没有每个样本自成一簇,就说明还不错,除样本间批次效应的效果。
5 细胞类型注释
跟单样本一致,
> library(celldex) #先加载包
> library(SingleR)
> ls("package:celldex")
[1] "BlueprintEncodeData" "DatabaseImmuneCellExpressionData"
[3] "defineTextQuery" "fetchLatestVersion"
[5] "fetchMetadata" "fetchReference"
[7] "HumanPrimaryCellAtlasData" "ImmGenData"
[9] "listReferences" "listVersions"
[11] "MonacoImmuneData" "MouseRNAseqData"
[13] "NovershternHematopoieticData" "saveReference"
[15] "searchReferences" "surveyReferences"
> ls("package:celldex") #这个花花老师下载了一部分,我们先用那里面的
[1] "BlueprintEncodeData" "DatabaseImmuneCellExpressionData"
[3] "defineTextQuery" "fetchLatestVersion"
[5] "fetchMetadata" "fetchReference"
[7] "HumanPrimaryCellAtlasData" "ImmGenData"
[9] "listReferences" "listVersions"
[11] "MonacoImmuneData" "MouseRNAseqData"
[13] "NovershternHematopoieticData" "saveReference"
[15] "searchReferences" "surveyReferences"
> f = "single_ref/ref_BlueprintEncode.RData"
> if(!file.exists(f)){
+ ref <- celldex::BlueprintEncodeData()
+ save(ref,file = f)
+ }
> ref <- get(load(f))
> library(BiocParallel)
> scRNA = sce.all #前面处理好了
> test = scRNA@assays$RNA$data
> pred.scRNA <- SingleR(test = test,
+ ref = ref,
+ labels = ref$label.main,
+ clusters = scRNA@active.ident)
> pred.scRNA$pruned.labels
[1] "CD4+ T-cells" "Fibroblasts" "B-cells" "CD8+ T-cells"
[5] "Neutrophils" "Monocytes" "Adipocytes" "CD4+ T-cells"
[9] "B-cells" "NK cells" "Fibroblasts" "Adipocytes"
[13] "Monocytes" "Endothelial cells" "Monocytes"
> new.cluster.ids <- pred.scRNA$pruned.labels #提取
> names(new.cluster.ids) <- levels(scRNA)
> scRNA <- RenameIdents(scRNA,new.cluster.ids)
> save(scRNA,file = "scRNA.Rdata") #这步需要点时间
> p2 <- DimPlot(scRNA, reduction = "umap",label = T,pt.size = 0.5) + NoLegend()
> p1+p2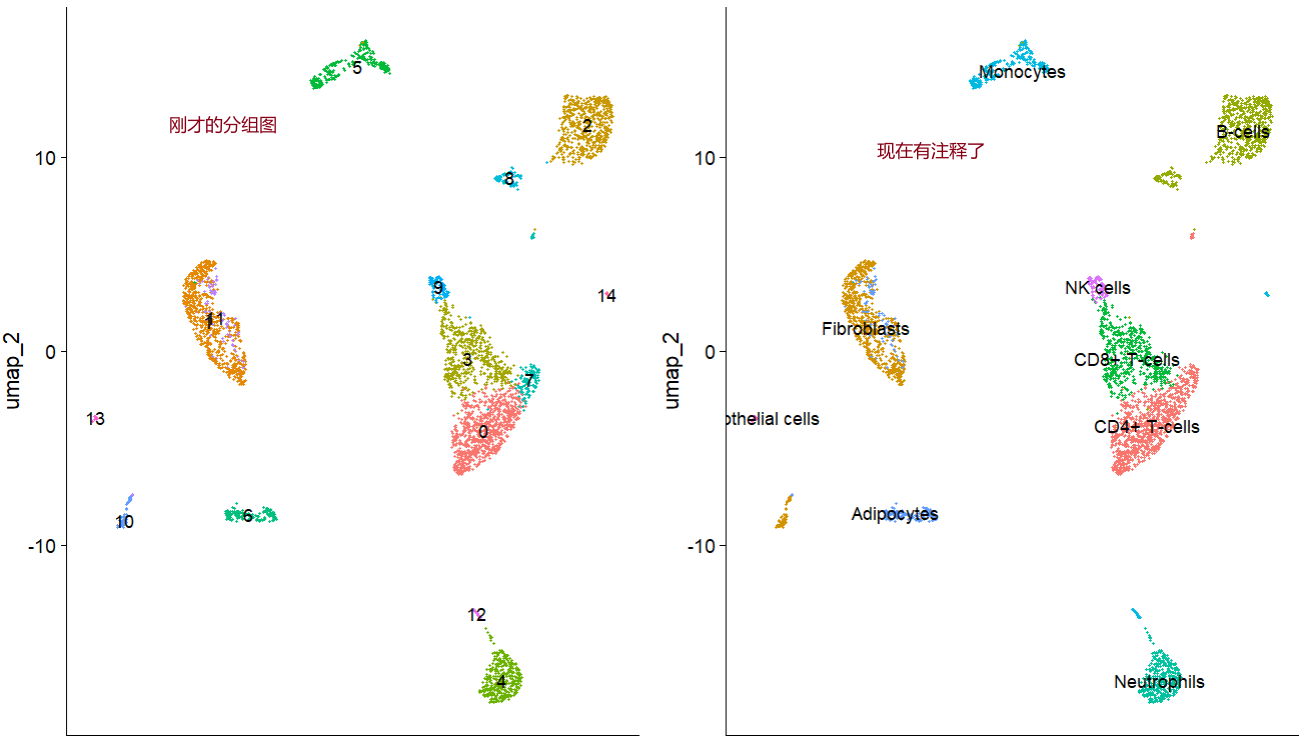
(要去下载那块看看这些样本有没有分组,没有分组现在就结束了,能得到的结果就是都有什么细胞,然后又分组的话还要进行其他处理)
6 分组可视化及组间细胞比例比较

看分组的快捷方法
BiocManager::install("clusterProfiler")
library(clusterProfiler)
install.packages("tinyarray")
library(tinyarray)
options("repos"="https://mirrors.ustc.edu.cn/CRAN/")
if(!require("BiocManager")) install.packages("BiocManager",update = F,ask = F)
options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
BiocManager::install("Biobase")
library(Biobase)
BiocManager::install("AnnoProbe")
library(AnnoProbe)
BiocManager::install("GEOquery")
library(GEOquery)
edat = geo_download("GSE231920") #终于成功了,要安装崩溃了,依赖包好多,一定要设置镜像,要不真的慢的可怕
pd = edat$pd之后按照后面的图点击就可以看到了
library(Seurat) #得先加载一下这个包
scRNA$seurat_annotations = Idents(scRNA) #把细胞类型设置为meta.data中的一列6.1 R语言基础知识补充,==和%in%
==:这是一个比较操作符,用于比较两个值是否相等。一一对应的关系
%in%:一个集合操作符,用于检查一个值是否存在于一个向量或列表中。并不是一一对应的关系
6.2 R语言基础知识补充 ifelse
ifelse(test, yes, no) :test:一个逻辑向量,每个元素是TRUE或FALSE,yes:当 test 为TRUE时返回的向量,no:当 test 为FALSE时返回的向量.
6.3 添加分组信息
scRNA$group和scRNA@meta.data$group是一样的,都会给meta.data添加一列,名为group
##Idents(scRNA) 返回一个向量,包含了 scRNA 对象中每个细胞的标签或身份。
> scRNA$group = ifelse(scRNA$orig.ident %in% c("sample1","sample2","sample3"), "treat","control")
> DimPlot(scRNA, reduction = "umap", group.by = "group")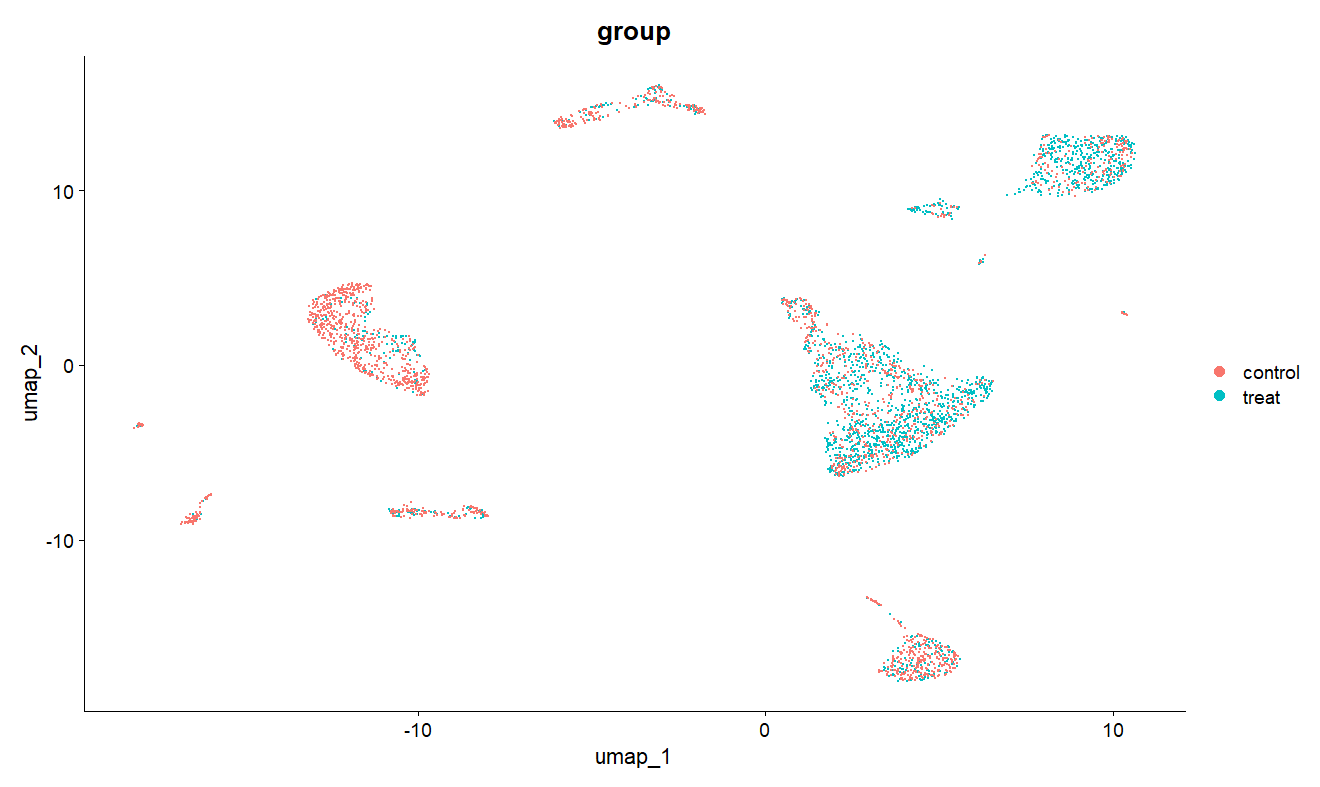
通过这张图发现对照组和处理组每种细胞占比不同了
6.4 计算每个亚群的细胞数量和占全部细胞的比例
#,table 函数对 scRNA 对象中由 Idents 函数返回的细胞身份(聚类结果)进行计数,table 函数计算每个唯一身份标签出现的次数,结果存储在 cell_counts 变量中
> cell_counts <- table(Idents(scRNA)) # 每种细胞的数量和比例
#这行代码使用 cbind 函数创建一个新的向量 cell.all,它由两列组成:cell_counts 是上面计算的每种细胞类型的计数。cell_Freq 是每种细胞类型的比例,计算方法是将 cell_counts 中的每个计数除以细胞总数(sum(cell_counts)),然后乘以100并四舍五入到小数点后两位。
> cell.all <- cbind(cell_counts = cell_counts,
+ cell_Freq = round(prop.table(cell_counts)*100,2))
> cell.num.group <- table(Idents(scRNA), scRNA$group) #各组中每种细胞的数量和比例
> cell.freq.group <- round(prop.table(cell.num.group, margin = 2) *100,2)
> cell.all = cbind(cell.all,cell.num.group,cell.freq.group)
> cell.all = cell.all[,c(1,3,4,2,5,6)]
> colnames(cell.all) = paste(rep(c("all","control","treat"),times = 2),
+ rep(c("count","freq"),each = 3),sep = "_")
> cell.all
all_count control_count treat_count all_freq control_freq treat_freq
CD4+ T-cells 1001 279 722 24.84 13.70 36.24
Fibroblasts 757 659 98 18.79 32.35 4.92
B-cells 670 185 485 16.63 9.08 24.35
CD8+ T-cells 551 207 344 13.68 10.16 17.27
Neutrophils 378 259 119 9.38 12.71 5.97
Monocytes 310 207 103 7.69 10.16 5.17
Adipocytes 247 168 79 6.13 8.25 3.97
NK cells 87 51 36 2.16 2.50 1.81
Endothelial cells 28 22 6 0.69 1.08 0.30
> 我的分群和花花老师不太一样,所以后面比例这块有点差距。
7.差异分析
刚才网络突然中断,眼泪差点流出来,时刻保存草稿
7.1 找某一细胞内部的组间差异基因
> sub.obj = subset(scRNA,idents = "NK cells") #找nk细胞内部的组件差异基因
> dfmarkers <- FindMarkers(scRNA, ident.1 = "treat", group.by = "group",min.pct = 0.25, logfc.threshold = 0.25,verbose = F)
#FindMarkers是Seurat包中的一个函数,用于检测在特定细胞群或条件下表达水平有显著差异的基因标记。ident.1 = "treat"这个参数指定了你想要检测的一组细胞或条件的标识符,group.by = "group"告诉FindMarkers函数根据scRNA对象中的group变量来分组细胞。logfc.threshold = 0.25这个参数设置了对数倍数变化(log2 fold change)的阈值。只有当基因表达的对数倍数变化大于0.25时,才会被认为是有差异表达的,verbose = F,F表示FALSE,即不输出额外信息。
For a (much!) faster implementation of the Wilcoxon Rank Sum Test,
(default method for FindMarkers) please install the presto package
--------------------------------------------
install.packages('devtools')
devtools::install_github('immunogenomics/presto')
--------------------------------------------
After installation of presto, Seurat will automatically use the more
efficient implementation (no further action necessary).
This message will be shown once per session
> head(dfmarkers)
p_val avg_log2FC pct.1 pct.2 p_val_adj
XIST 0.000000e+00 -13.158248 0.000 0.655 0.000000e+00
RPS4Y1 0.000000e+00 12.919666 0.650 0.000 0.000000e+00
DDX3Y 0.000000e+00 12.552343 0.531 0.000 0.000000e+00
RPS26 5.212174e-301 1.877581 0.905 0.733 1.269529e-296
CFD 2.668149e-197 -4.424226 0.142 0.575 6.498810e-193
IGHG4 3.235562e-197 6.488025 0.444 0.037 7.880858e-1937.2 找conserved marker基因
if(!require("multtest"))BiocManager::install('multtest') #安包
if(!require("metap"))install.packages('metap')
> sub.markers <- FindConservedMarkers(scRNA, ident.1 = "NK cells", grouping.var = "group", min.pct = 0.25, logfc.threshold = 0.25,verbose = F)
> #上面那个代码就是找conserved marker基因
> head(sub.markers)
treat_p_val treat_avg_log2FC treat_pct.1 treat_pct.2 treat_p_val_adj
KLRF1 4.902284e-283 8.831252 0.861 0.005 1.194049e-278
FGFBP2 3.052720e-204 7.954269 0.750 0.008 7.435511e-200
GNLY 2.119593e-156 6.906460 1.000 0.034 5.162693e-152
S1PR5 2.155042e-177 7.384016 0.556 0.004 5.249037e-173
GZMB 9.549209e-135 5.964544 0.944 0.036 2.325901e-130
PRF1 2.169569e-113 5.923958 0.972 0.051 5.284420e-109
control_p_val control_avg_log2FC control_pct.1 control_pct.2 control_p_val_adj
KLRF1 1.866839e-177 7.464802 0.647 0.010 4.547060e-173
FGFBP2 9.794843e-247 7.196772 0.843 0.011 2.385730e-242
GNLY 1.959075e-187 6.570793 0.902 0.027 4.771718e-183
S1PR5 1.653375e-84 5.602195 0.392 0.010 4.027127e-80
GZMB 1.276982e-173 5.664061 0.882 0.028 3.110345e-169
PRF1 3.321701e-168 5.983944 0.941 0.036 8.090667e-164
max_pval minimump_p_val
KLRF1 1.866839e-177 9.804567e-283
FGFBP2 3.052720e-204 1.958969e-246
GNLY 2.119593e-156 3.918149e-187
S1PR5 1.653375e-84 4.310085e-177
GZMB 9.549209e-135 2.553964e-173
PRF1 2.169569e-113 6.643402e-168FindConservedMarkers和前面的FindMarkers 不大一样。它结合了“分组”和“细胞类型”,是找出不同细胞类型之间在不同条件(treat和control)下都有差异的基因。我理解的就是control中nk和其他细胞的差异基因,和treat和其他细胞之间的差异基因,他俩取交集就是无论在control和treat组中NK和其他细胞都有差异的基因,这个会叫NK和其他细胞的差异保守的基因。
7.3 组间比较的气泡图
markers.to.plot = c("CD3D", "CREM", "HSPH1", "SELL", "GIMAP5", "CACYBP", "GNLY", "NKG7", "CCL5",
"CD8A", "MS4A1", "CD79A", "MIR155HG", "NME1", "FCGR3A", "VMO1", "CCL2", "S100A9", "HLA-DQA1",
"GPR183", "PPBP", "GNG11", "HBA2", "HBB", "TSPAN13", "IL3RA", "PRSS57") #一组感兴趣的基因,挑出来赋值
#如果idents有NA会报错https://github.com/satijalab/seurat/issues/8772
#scRNA <- subset(scRNA, seurat_annotations %in% na.omit(scRNA$seurat_annotations))
DotPlot(scRNA, features = markers.to.plot, cols = c("blue", "red"),
dot.scale = 8, split.by = "group") +
RotatedAxis() #之前看过这个图,原来是用这个画出来的。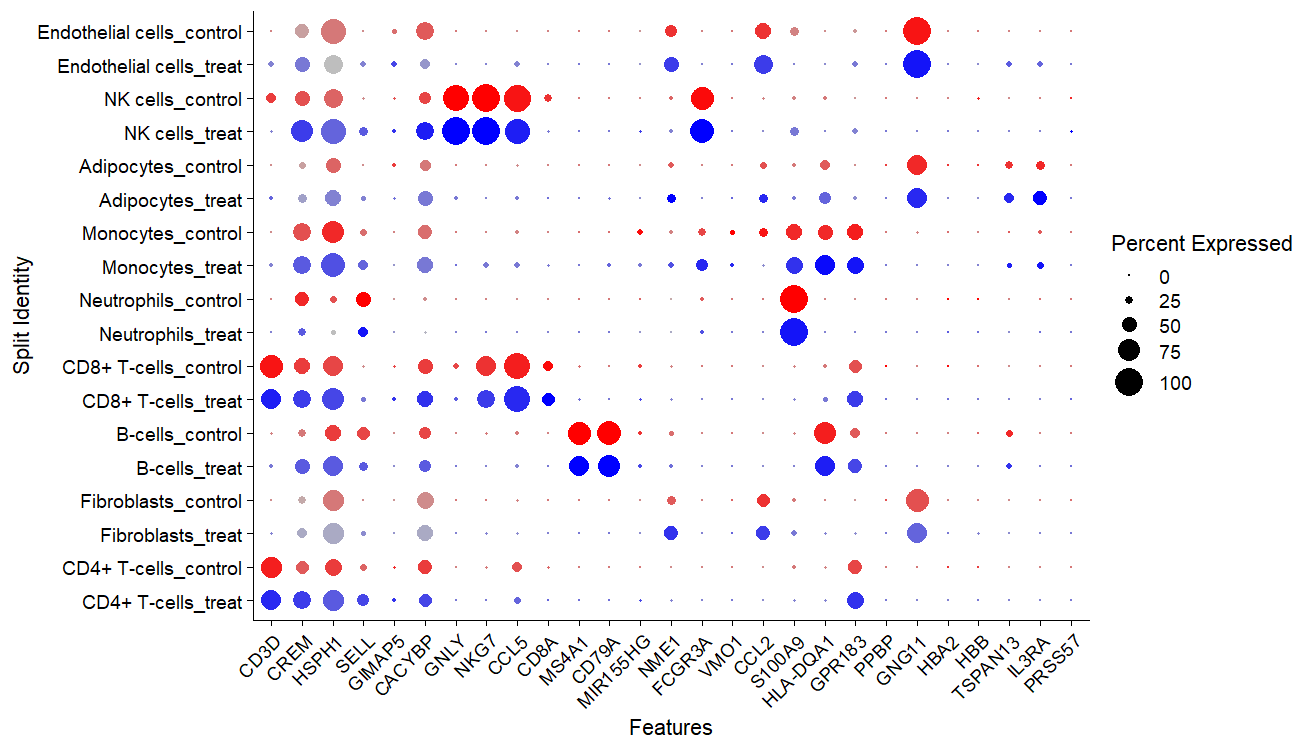
点的大小和颜色表示表达量
7.4 FeaturePlot
一行是一个基因,一列是一个分组,可以很直观的对比。但是有一个致命缺点,想看的基因多的话数据太多了
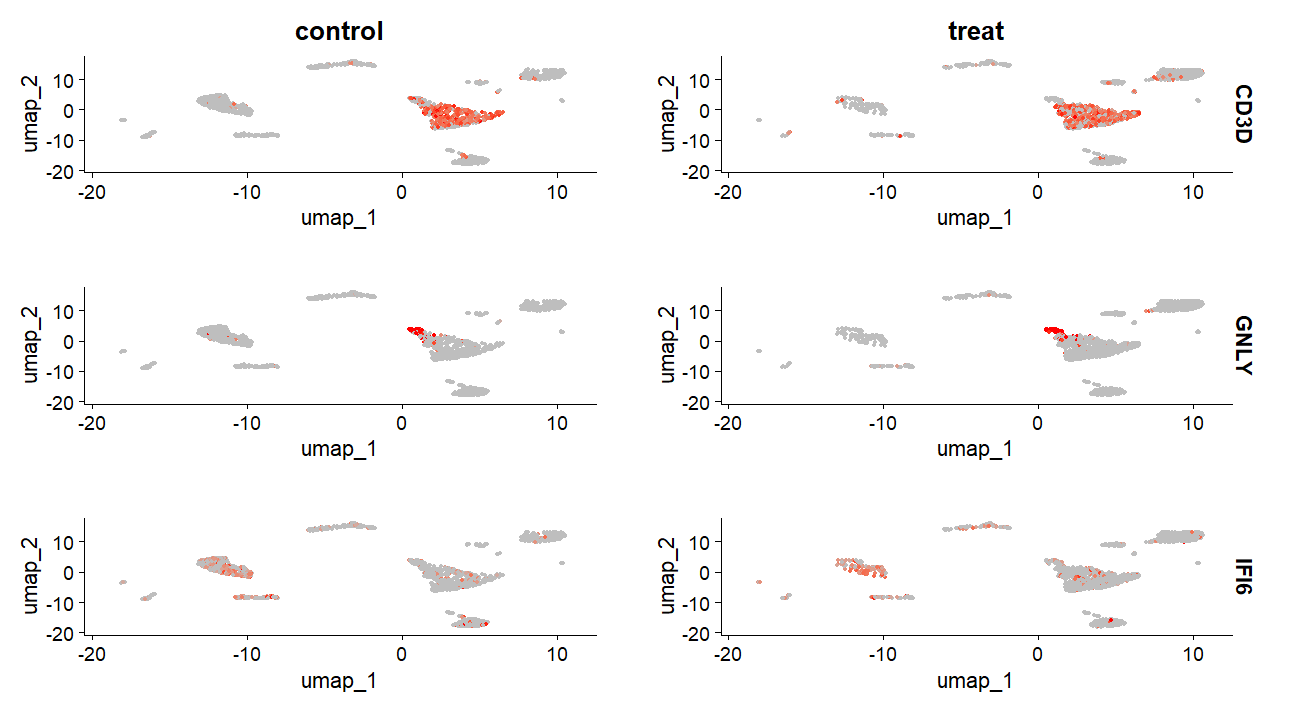
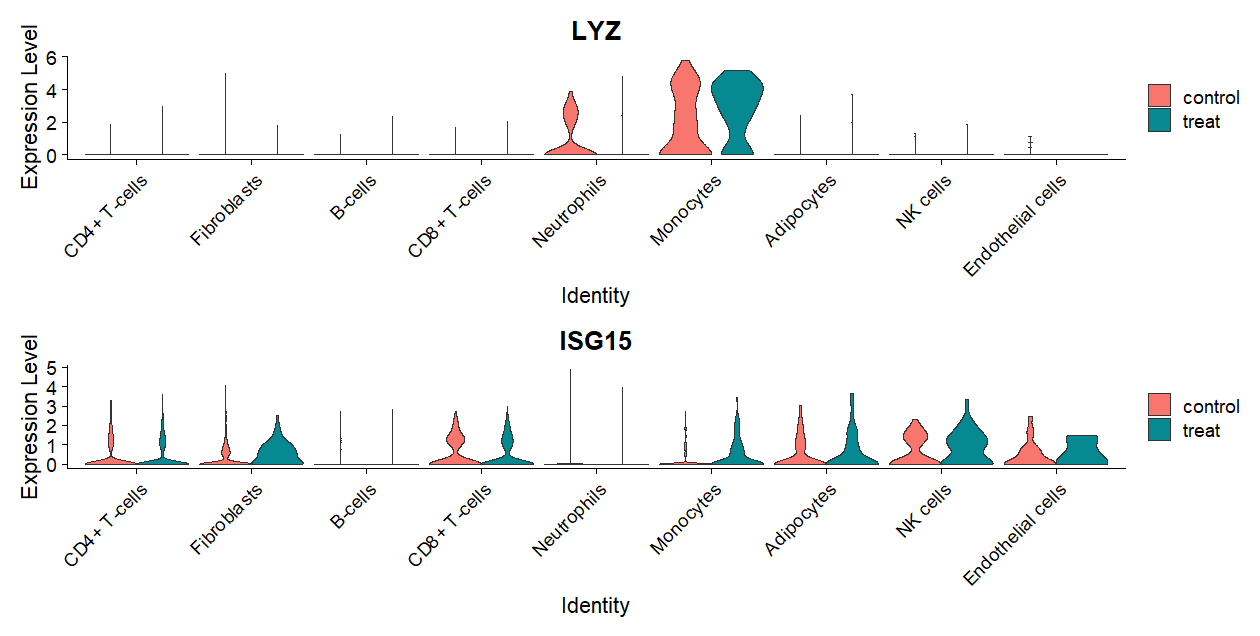
7.5 VlnPlot
三个基因有点拥挤,换成两个了
8.伪bulk 转录组差异分析
bulk转录组:"Bulk"转录组通常指的是传统的、非单细胞分辨率的转录组分析
多样本才能做这个分析 #意义是什么,好吧看到后面理解啦,话火山图用的,要不怎么花差异基因图
AggregateExpression是把单细胞数据整合为常规转录组数据的方式。group.by = c("seurat_annotations","orig.ident", "group")就是同一细胞类型、同一样本、同一分组的细胞汇总在一起成为一个“样本”。
# 伪bulk
bulk <- AggregateExpression(scRNA, return.seurat = T, slot = "counts", assays = "RNA", group.by = c("seurat_annotations","orig.ident", "group"))
colnames(bulk)#整合成了多个“样本”
# 明白了,可以只取出一种细胞,然后找treat和control之间的差异基因
sub <- subset(bulk, seurat_annotations == "CD8+ T-cells")
colnames(sub)
Idents(sub) <- "group"
BiocManager::install("DESeq2")
library(DESeq2)
de_markers <- FindMarkers(sub, ident.1 = "treat", ident.2 = "control", slot = "counts", test.use = "DESeq2",
verbose = F)
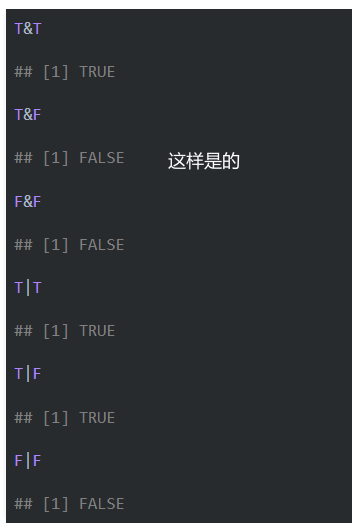
de_markers$gene <- rownames(de_markers)8.2 R语言基础知识补充 逻辑值连接符号
&(shift+7)是并且,用&连接的两个或多个条件都是T才返回T; | (shift+回车上方的)是或者,用|连接的两个或多个条件只要有一个T就返回T。
8.3 火山图
#火山图
k1 = de_markers$avg_log2FC< -1 & de_markers$p_val <0.01
k2 = de_markers$avg_log2FC> 1 & de_markers$p_val <0.01
de_markers$change <- ifelse(k1,"down",ifelse(k2,"up","not"))
#满足k1就是down,不满足的话再看是否满足k2,满足k2就是up,不满足就是not。
library(ggplot2)
ggplot(de_markers, aes(avg_log2FC, -log10(p_val),color = change)) +
geom_point(size = 2, alpha = 0.5) +
geom_vline(xintercept = c(1,-1),linetype = 4)+
geom_hline(yintercept = -log10(0.01),linetype = 4)+
scale_color_manual(values = c("#2874C5", "grey", "#f87669"))+
theme_bw() +
ylab("-log10(unadjusted p-value)") 
花花老师推荐ggplot2学习资料:https://r4ds.hadley.nz/data-visualize
花花老师代码参考https://satijalab.org/seurat/articles/integration_introduction
https://satijalab.org/seurat/articles/parsebio_sketch_integration
用时非常长,长到破防。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

