【分子生物学】单基因遗传突变造成的蛋白质三级结构变化-从NCBI到swissmodel到pymol实操
【分子生物学】单基因遗传突变造成的蛋白质三级结构变化-从NCBI到swissmodel到pymol实操
前言:
在临床研究中发现患者致病基因点突变,从而影响基因的表达与翻译。如何研究一个基因的点突变是否会造成氨基酸序列在三级结构发生改变,氨基酸残基的相互作用是否也存在改变?带着这个问题,今天我们来进行短暂的学习研究。
操作步骤
1.获取基因的蛋白质序列
1.1 NCBI网站获取DDX58蛋白序列并进行同源性比较
在NCBI数据库中选择【protein】选项,输入DDX58后,自动默认选择为人类DDX58,然后点击中间Orthologs[同源]按钮。
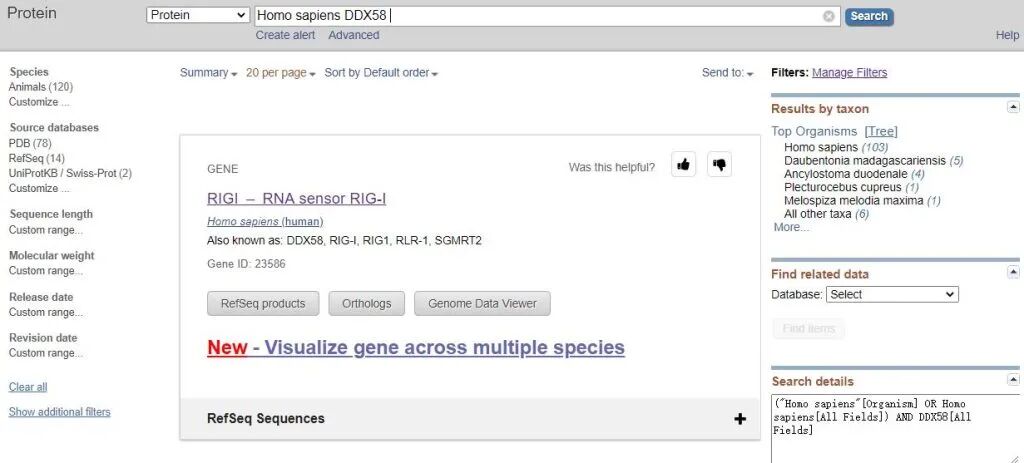
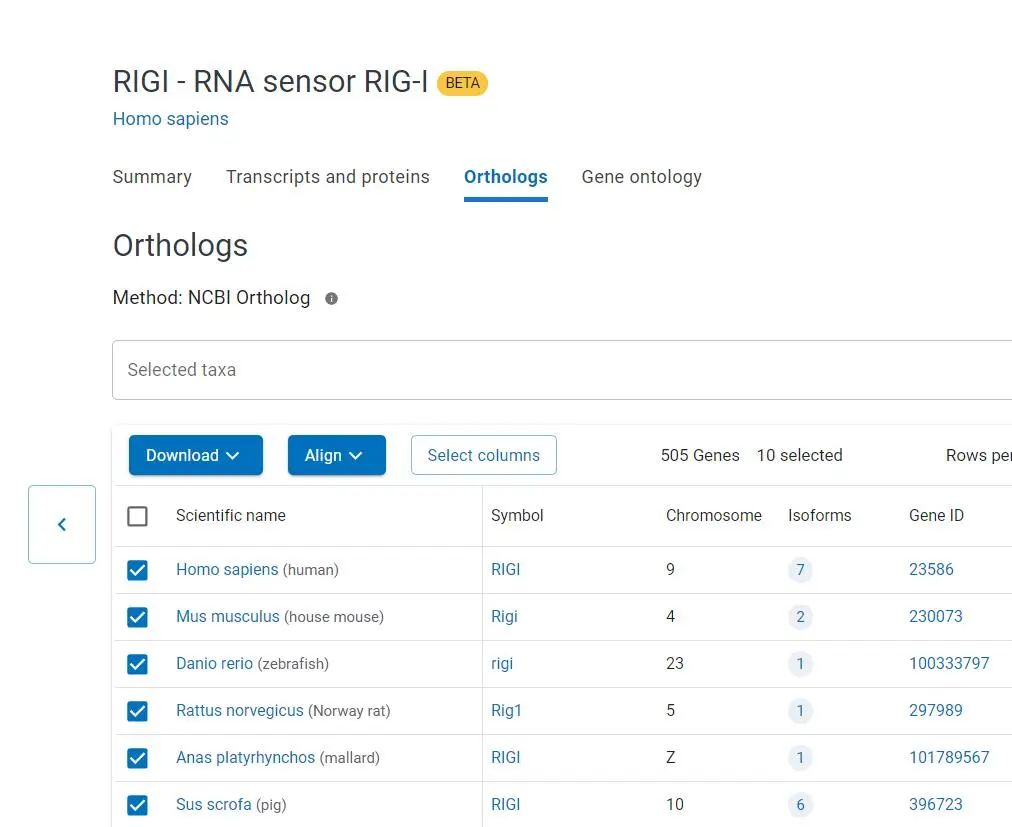
如下图所示,在弹出的页面中,再次点击【Orhtologs】,出现如下界面。选择不同物种,在这里选择了前10个物种进行比较。
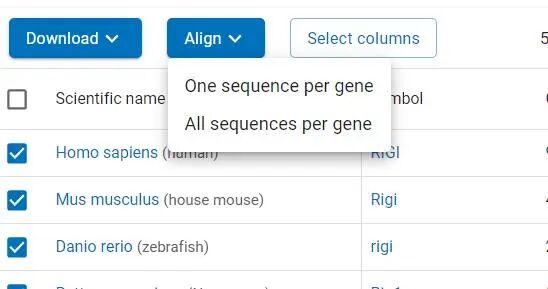
比对:点击ALign,选择第一个选项,一个基因选择一条序列进行比较。
出现如下界面,在COBALT输入框出现每一个物种的蛋白ID号,然后点击Align。等待比对结果的出现。
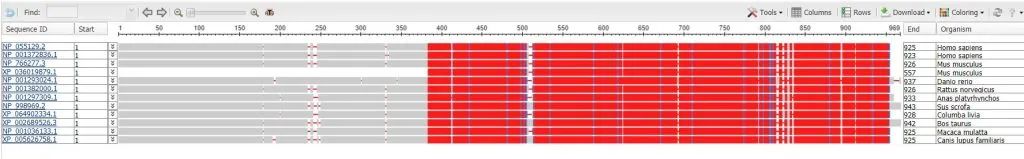
如图所示,物种之间在370位置氨基酸后到最后位置的序列保守性比较强。
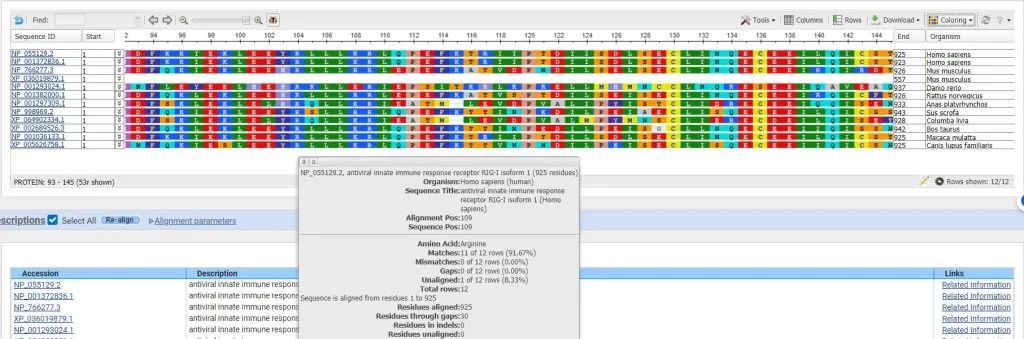
已知在人DDX58出现 R109C突变,所以进一步观察109号氨基酸位置附近氨基酸的同源性比较,在【Coloring】界面调整颜色设置,选择临近氨基酸差异更大的颜色设置。如图所示,所有物种中109号氨基酸序列都是R,小鼠中出现了两条序列,其中一条序列缺乏这一段氨基酸。总体来说,从94-142位点,不同物种整体的同源性还是存在,部分区域氨基酸发生改变。
1.2 选择合适的氨基酸序列进行下游分析
比对人的DDX58氨基酸序列的相似性。人的DDX58氨基酸序列存在7种不同的氨基酸多肽,4条序列在109号氨基酸存在R,因此我们选择这4条中的之一进行氨基酸序列的获取。我们选择Isoform1这一条,因为这一条是最长的,925个氨基酸,也就是下图的最后一条序列。当然,还可以根据表达高低进行选择。

如图下图所示,这就是DDX58(RIG-1) isoform1蛋白的信息,点击【FASTA]选项,鼠标选择氨基酸序列输入到目的网站Swissmodel进行下一步操作。https://swissmodel.expasy.org/interactive


2.网站Swissmodel获得DDX58蛋白三维结构图
2.1 将DDX58氨基酸序列输入到文本框,可填写project Title 和 Email以便于任务完成后在邮箱查看。也可以不填写,直接点击Search for templates 或者Build Model。对于未知的序列可以用前者,对于已知序列,其实可以直接点击Build model进行后续操作。
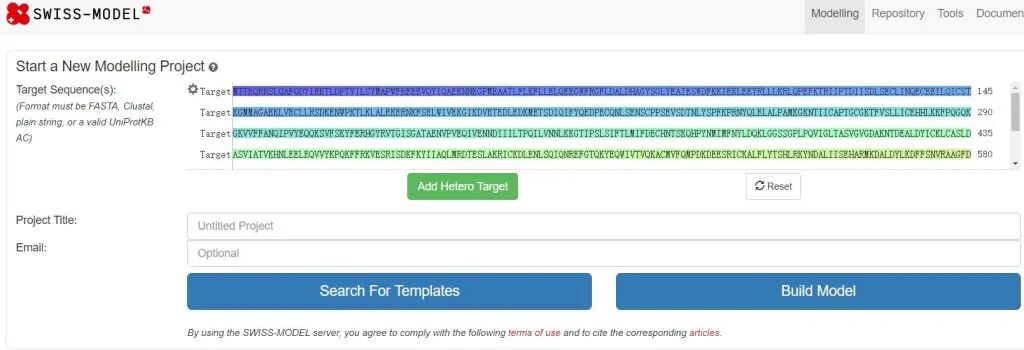
我们点击了【Search for templates】开始进行序列相似性检索比对。
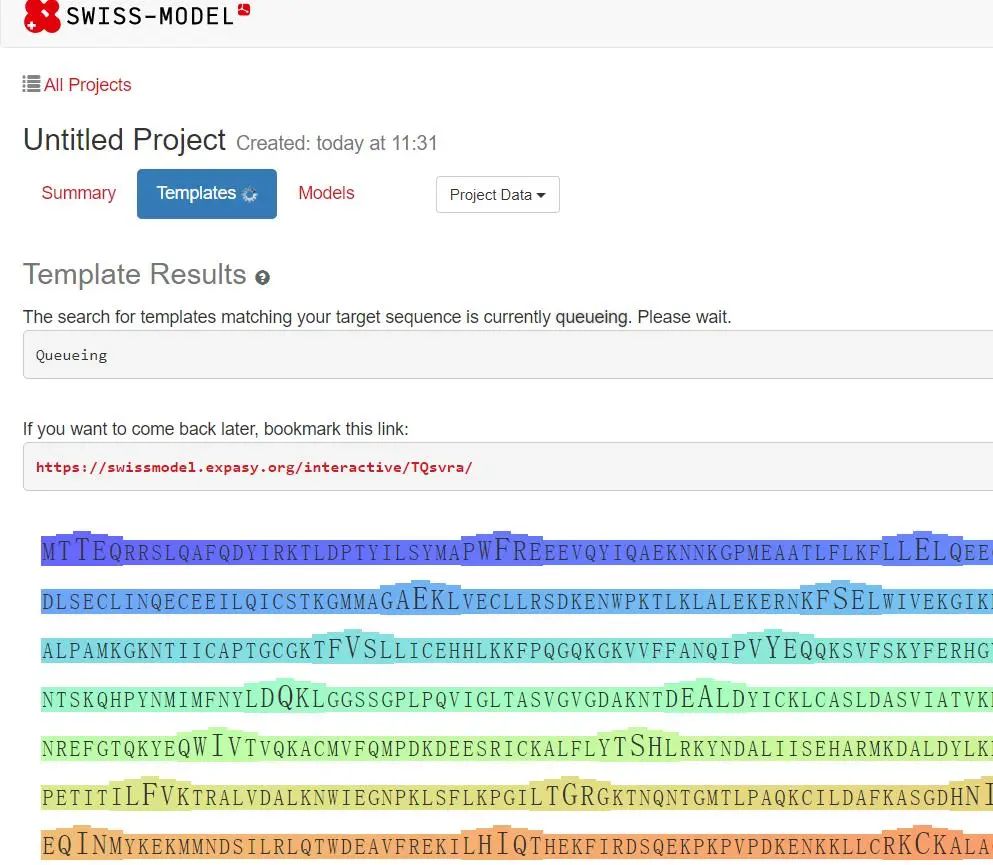
后续结果显示搜索到的模板结果,如下图所示,第一个是AlphaFold 对倭黑猩猩的RIG-1蛋白三维模拟的结果,第2-3是cryo-EM(冷冻电镜)
对RIG-1的三维结构结果。我们选择这三个template 进行BUild Models。
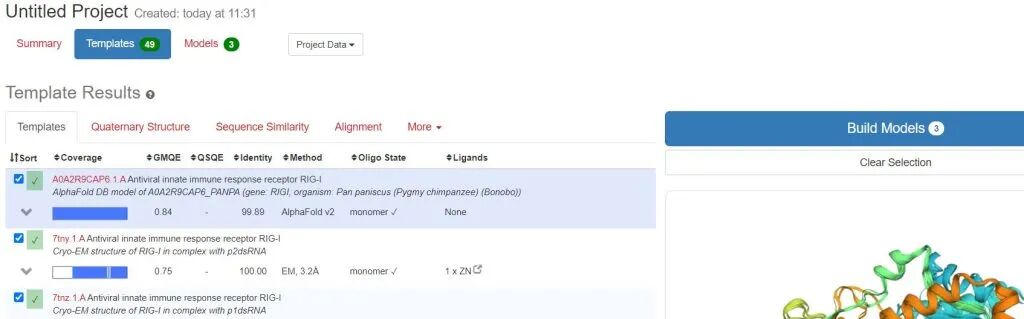
如图所示,Models共模拟了三个结果,其中Model -3 GMQE最高。选择下载PDB format文件。同时点击Structure Assessment进行详细查看蛋白质三级结构信息。
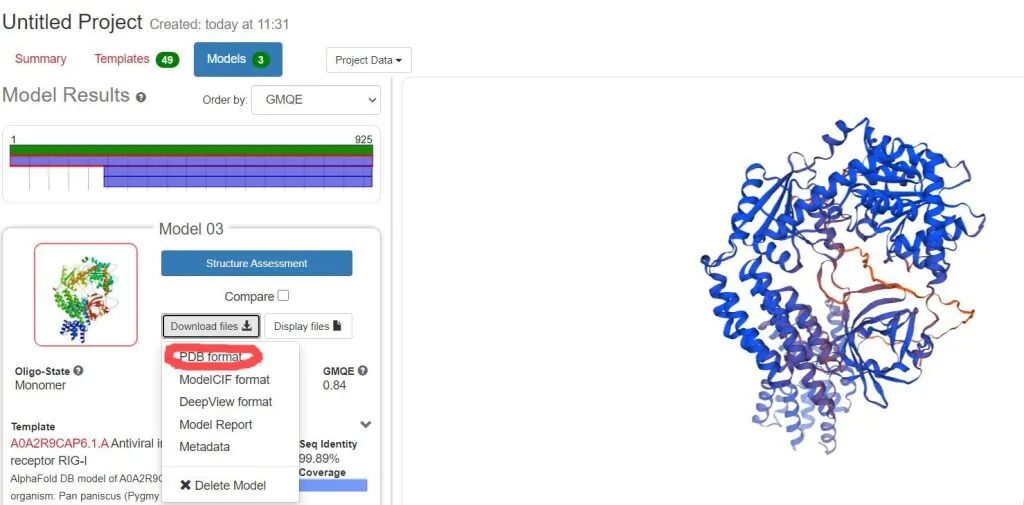
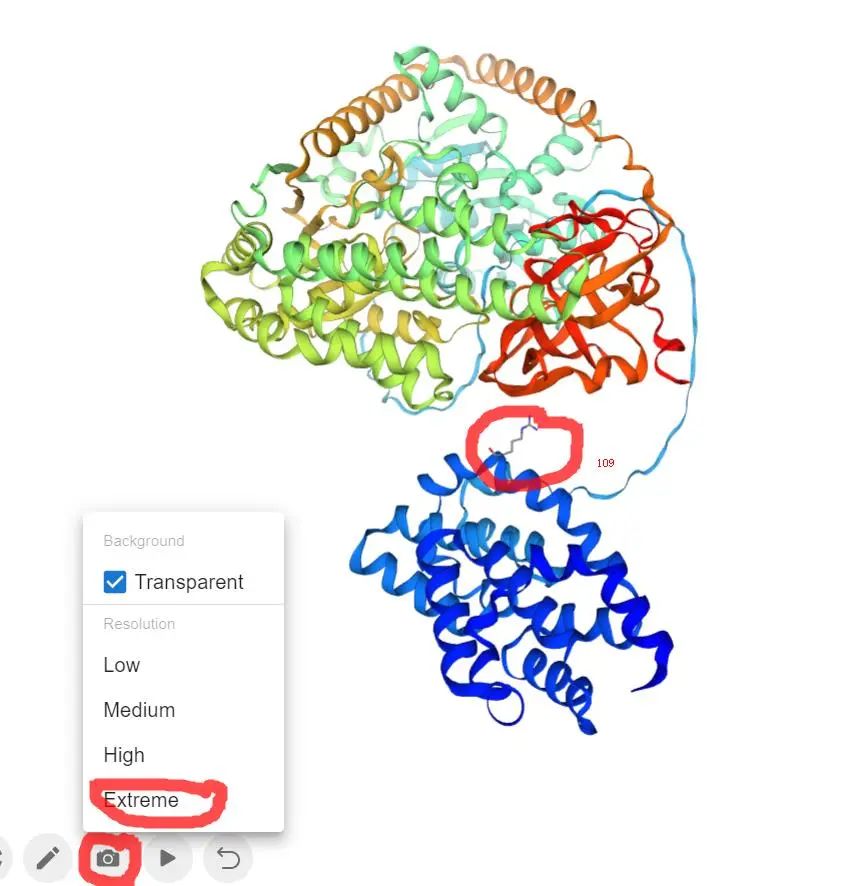
后续进入新界面后,还可以下载蛋白质多色三维结构图,调整好方向,并且突出109号氨基酸的残基位置。下载高清png格式图片。
3.Pymol软件下进行DDX58蛋白三维结构及109号氨基酸氢键互作可视化
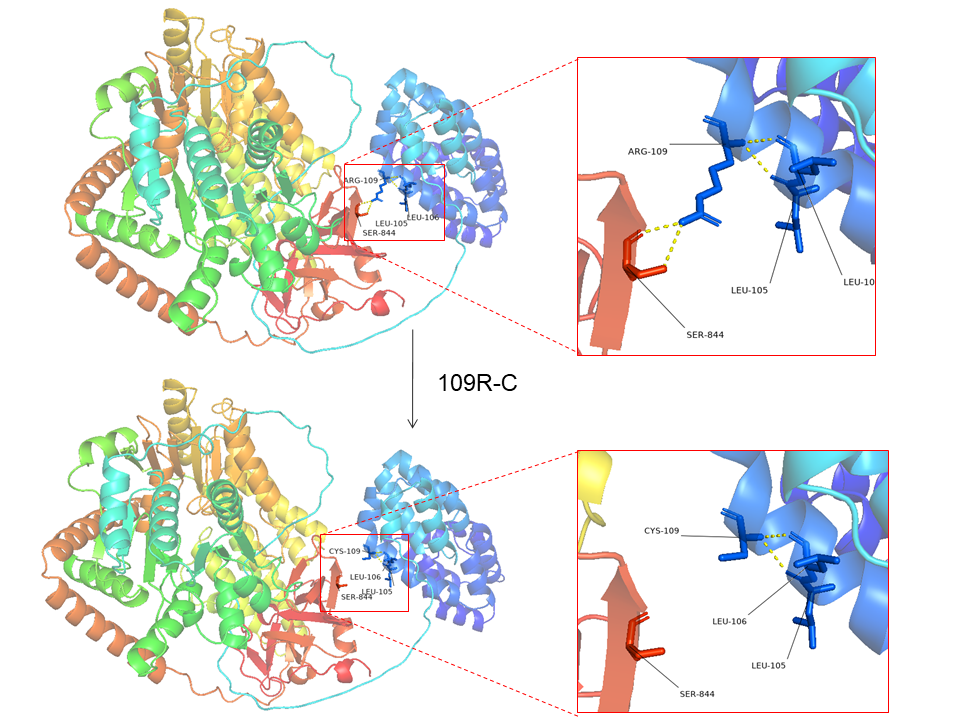
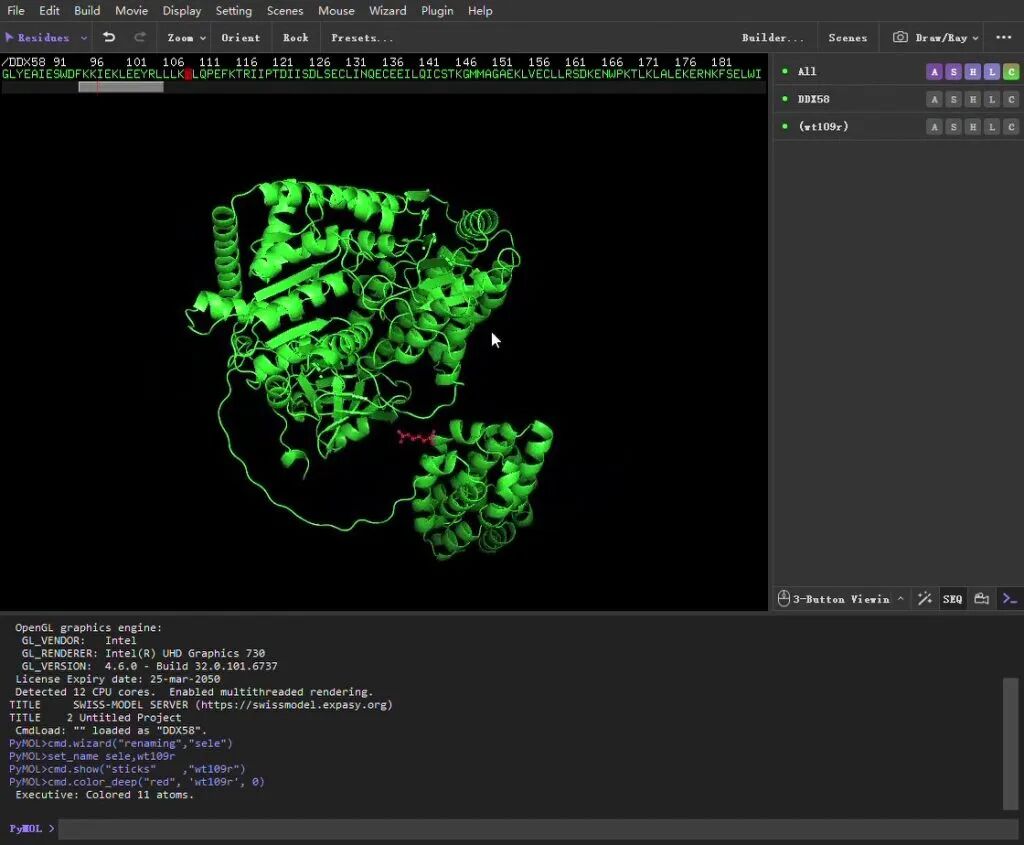
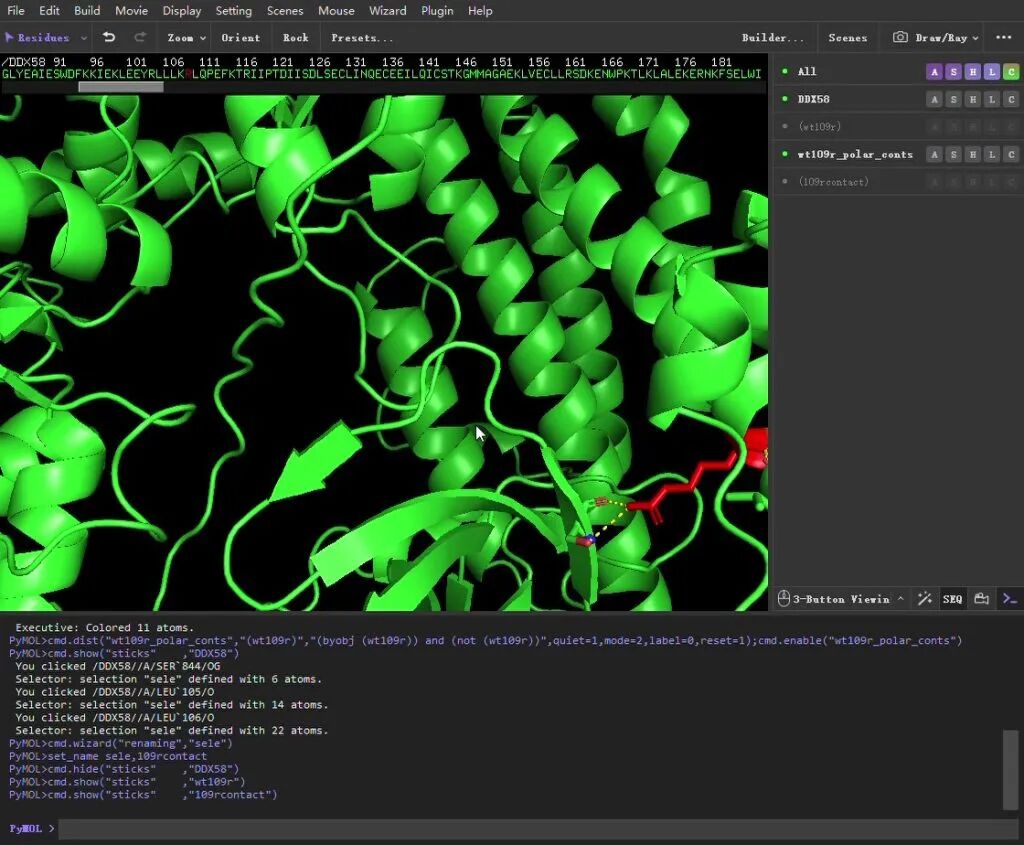
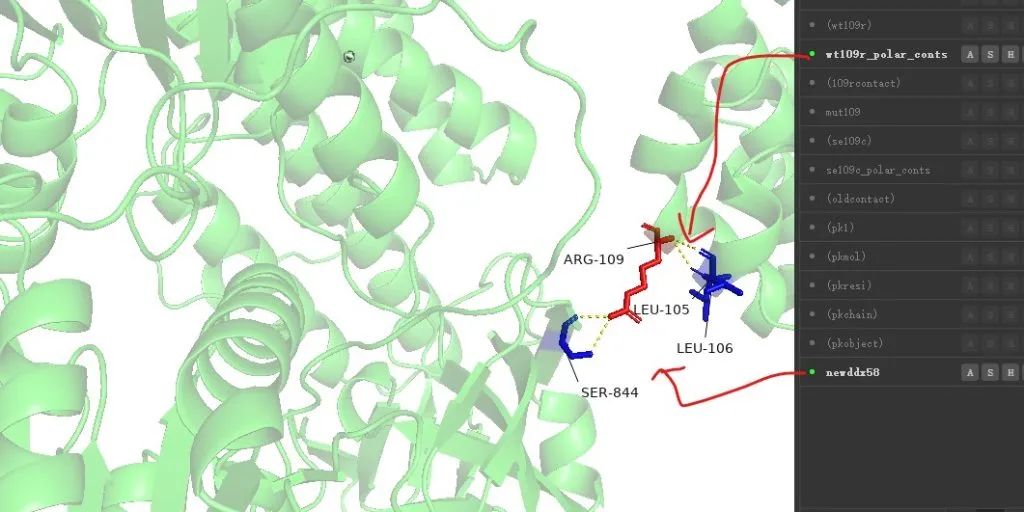
3.1 FIle 目录下导入DDX58 蛋白的PDB文件。选择109号氨基酸,命名为wt109r,通过S(show)选择Stick,点击C(color)选择red.如图所示便可用红色展示109号氨基酸位置以及显示其氨基酸结构特征。
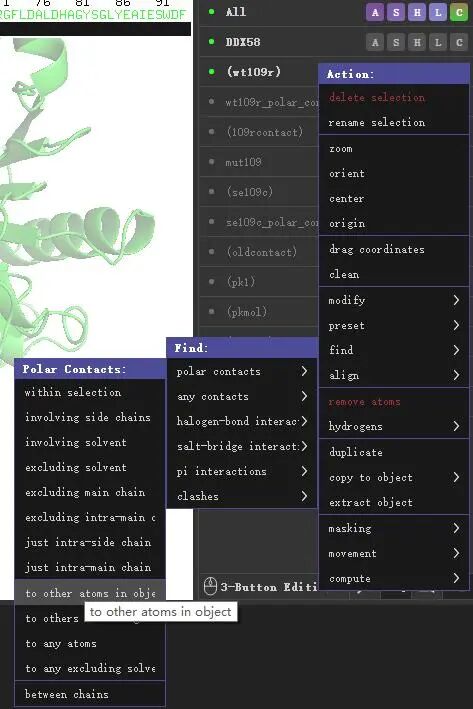
3.2 找出109号氨基酸与DDX58蛋白其他氨基酸氢键相互作用。选择wt109r这个对象,点击A菜单下【find】-【plar contacts】-【to others atoms in subject】
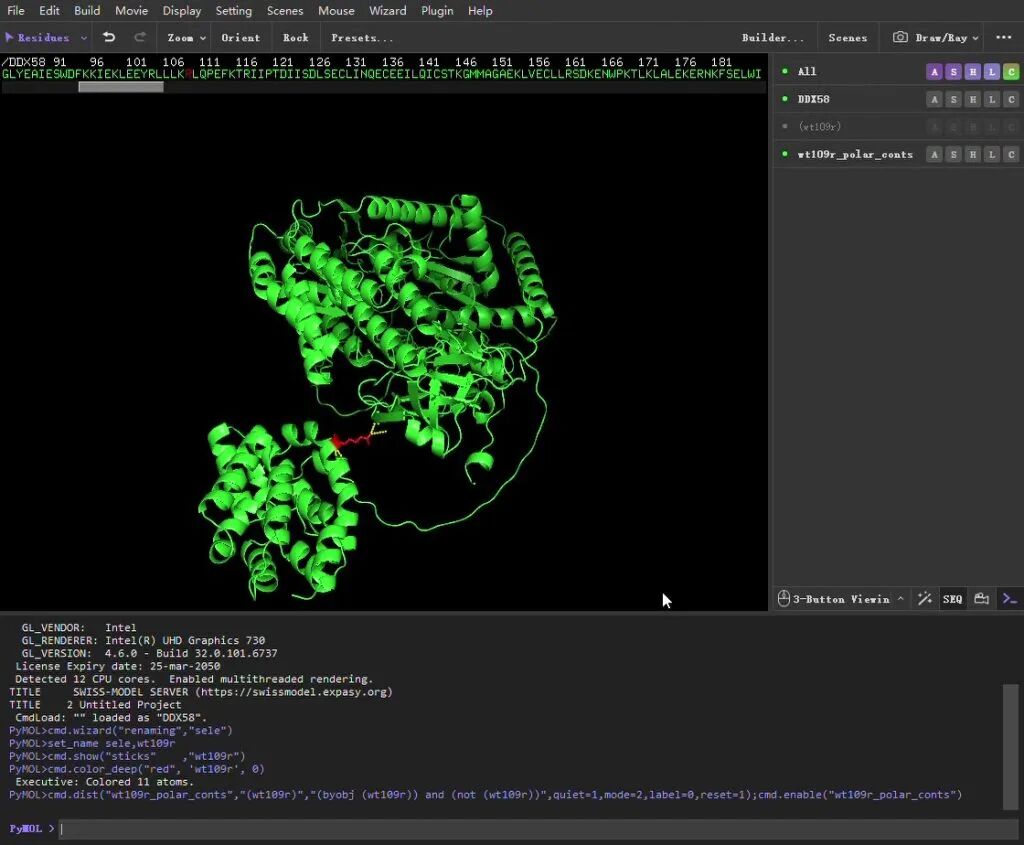
如图所示,便可显示109号氨基酸与其他氨基酸的氢键形成情况。
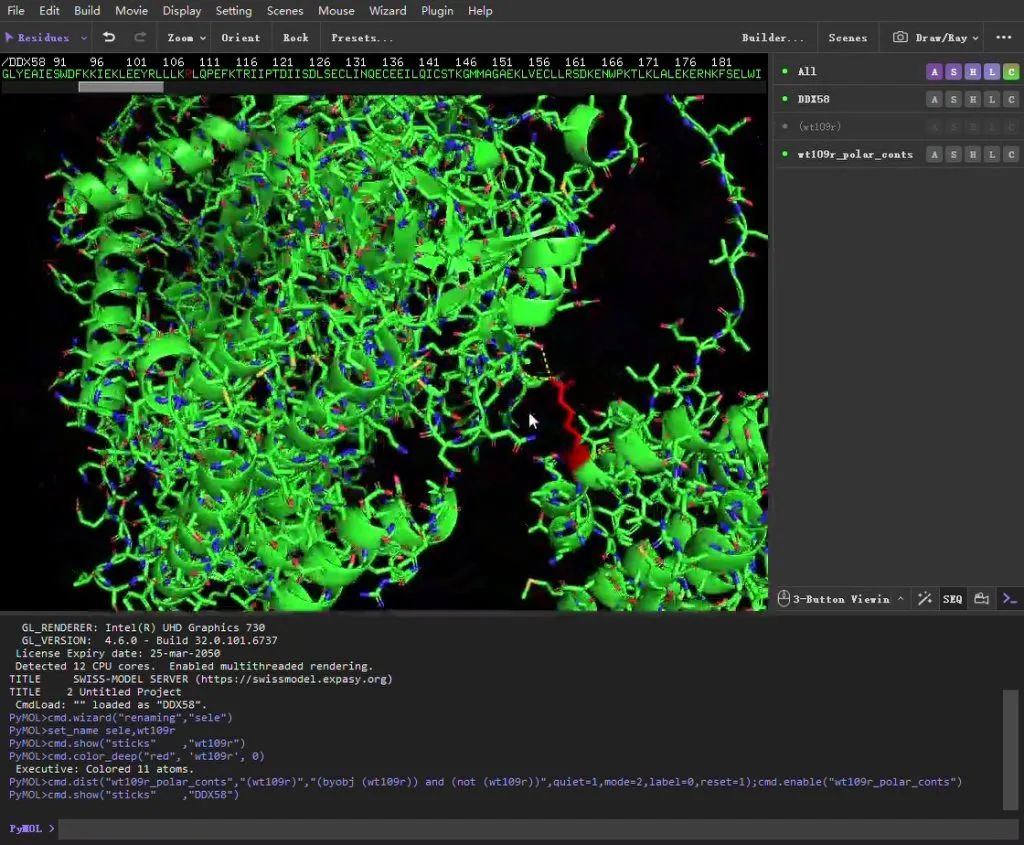
然后通过对DDX58 文件【S】点击Stick选项,以Stick形式展示蛋白上所有氨基酸情况,查看与109号氨基酸形成氢键的氨基酸是哪几个,用鼠标进行选择。
将所有与109号氨基酸形成氢键的氨基酸都选择好后,将这些对象重新命名109rcontact,然后点击【DDX58】这个对象,在【H】选项中隐藏Stick.在【109rcontact】文件中【S】点击Stick,如此以来,只显示了与109号氨基酸形成氢键的氨基酸。
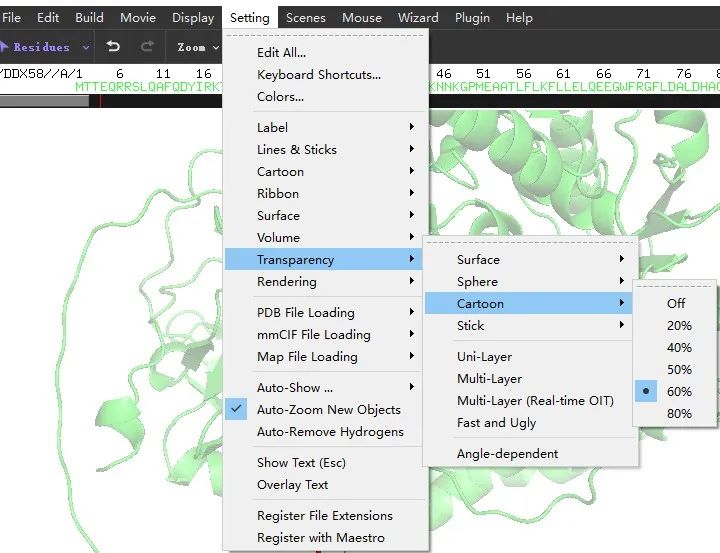
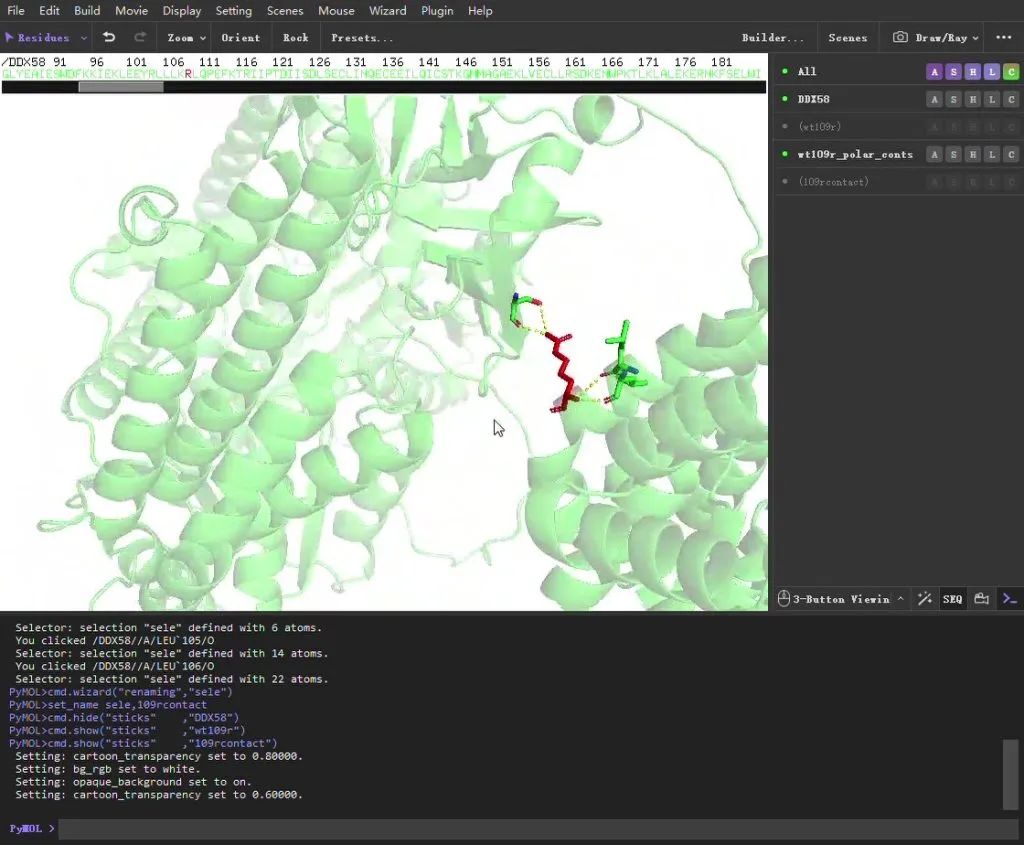
背景肽链透明化:通过【Setting】-【Transparency】-【Catoom】-【60%】实现背景肽链透明度增加。突出109号氨基酸与其他氨基酸的相互作用。
如下图所示
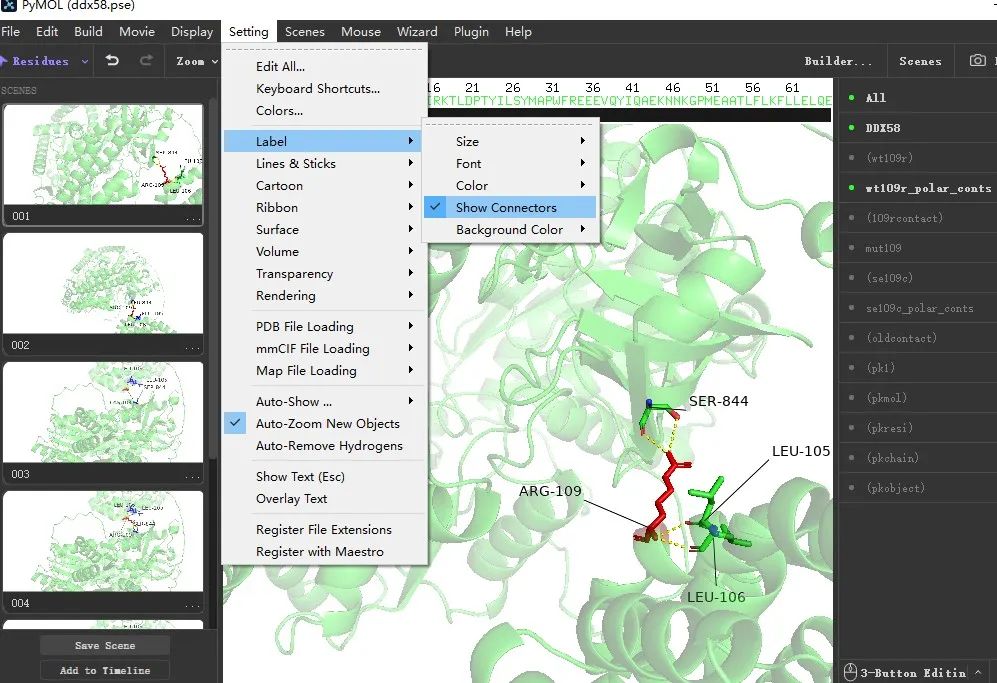
显示氨基酸名称并增加连线:选择氨基酸对象【wt109r】或者【109rcontact】 点击【L】-【residues】显示氨基酸名称。在上方【Setting】-【Label】-【Show Connetcors】进行鼠标连线操作。将文字用鼠标左键+Ctrl调整到图面空白位置即可。
4. DDX58蛋白109号氨基酸从ARG精氨酸变成 后的氢键互作改变可视化
4.1 新建一个DDX58的对象。点击DDX58【A]】-【copy to object]-【new】,然后点击new对象,【A]】-【rename object】进行重命名.如图所示。
4.2点击魔法棒按钮,弹出【Wizards】界面,选择【ProteinMutagenesis】
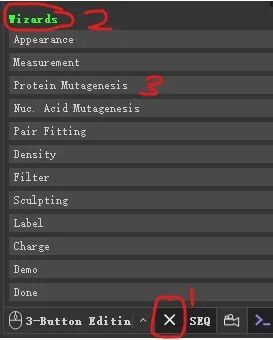
4.3 选择Mutant to CYS

4.4 选择需要更换的氨基酸-109号R氨基酸,软件将生成3个不同的cys氨基酸在空间中的位置,通过左右方向按键,选择rotamer 1/2/3中,strain至最小的一个,然后点击Apply
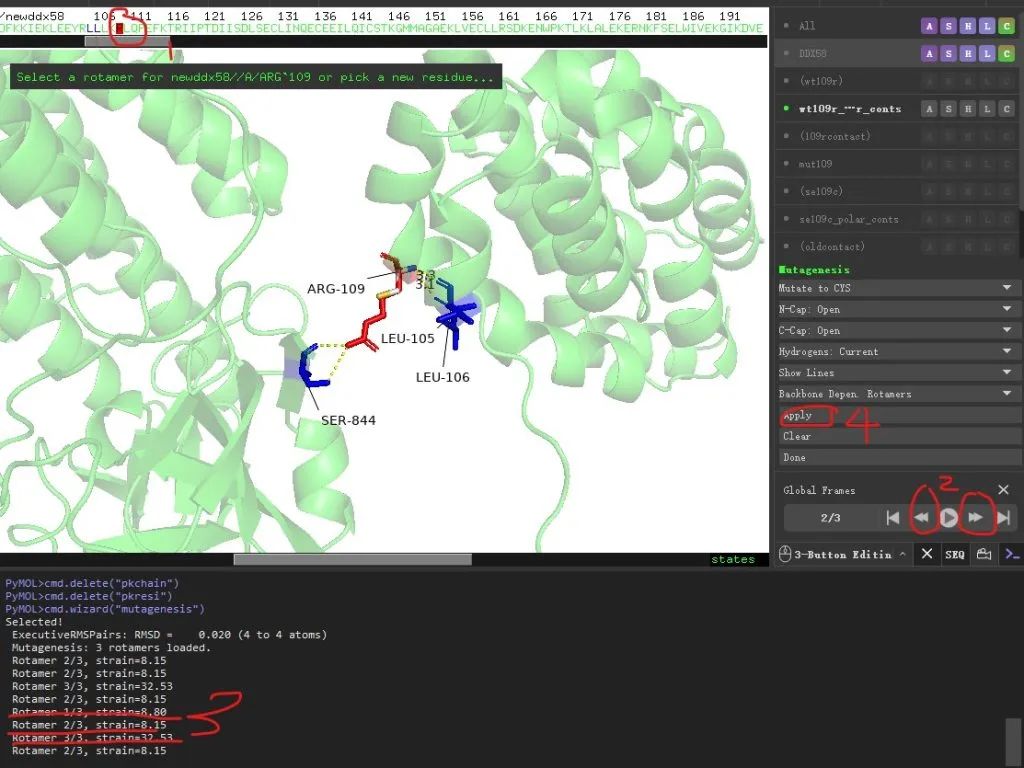
4.5 如下图所示,此时109号氨基酸从ARG精氨酸变成了CYS半胱氨酸。依然保留着原来氢键互作线条,点击wt109r contact取消选中。

4.6 重新选择109号氨基酸对象,并命名为109cys.随后点击A菜单下【find】-【plar contacts】-【to others atoms in subject】进行109号半胱氨酸与其他氨基酸形成氢键的可能性预测。
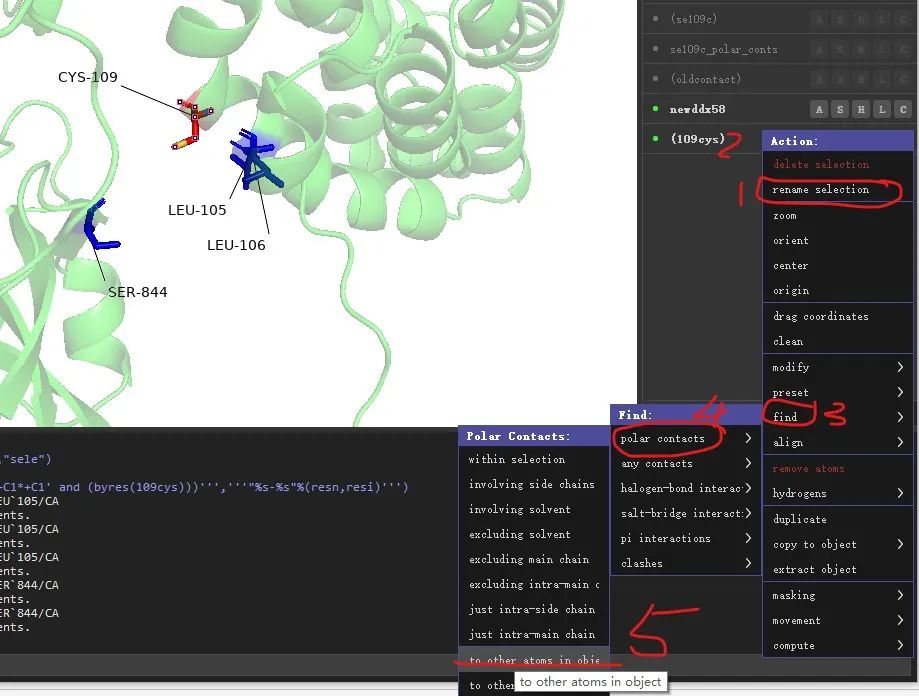
4.7 如下图所示,当109号氨基酸变成半胱氨酸CYS后,只与105号,106号氨基酸有氢键的形成。点击【Scenes】按钮,然后点击【save scenes】拍摄图片。
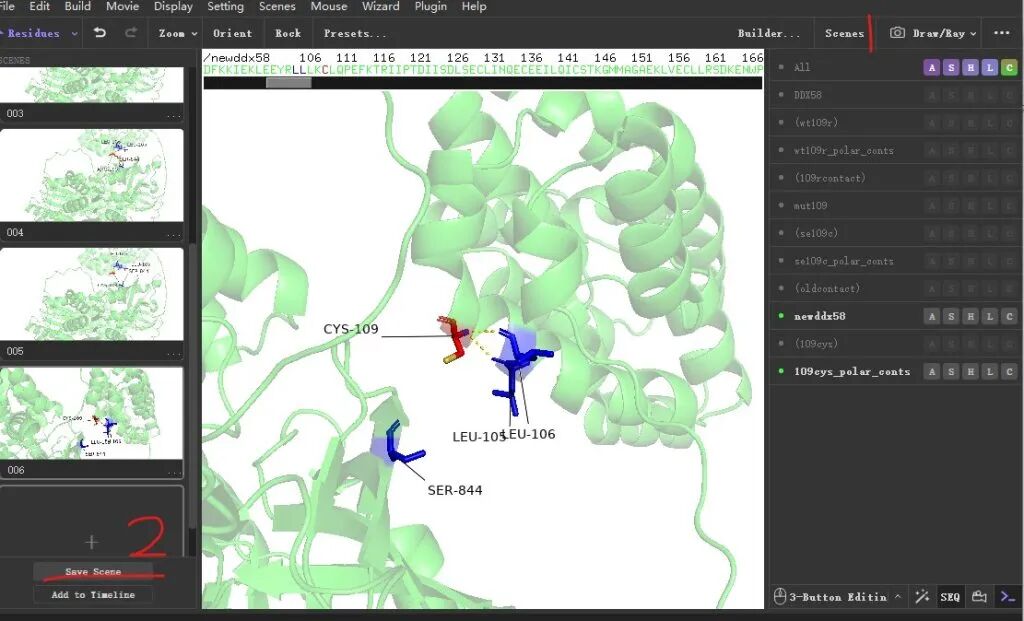
4.8 在同一视角下,取消对newddx58及相关对象 的选择,选择突变前的文件对象DDX58以及wt109r_ploar conts,同样拍摄图片。

4.9 保持图片到电脑中,选择【file】-【Export image AS】 -【PNG 】
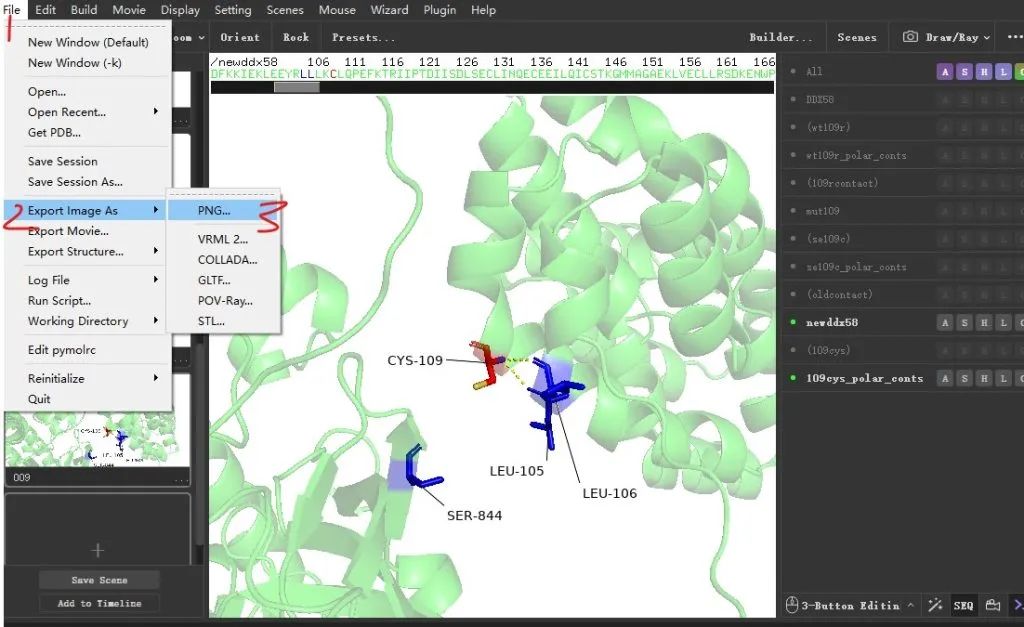
弹出窗口选择其一模式进行保持。
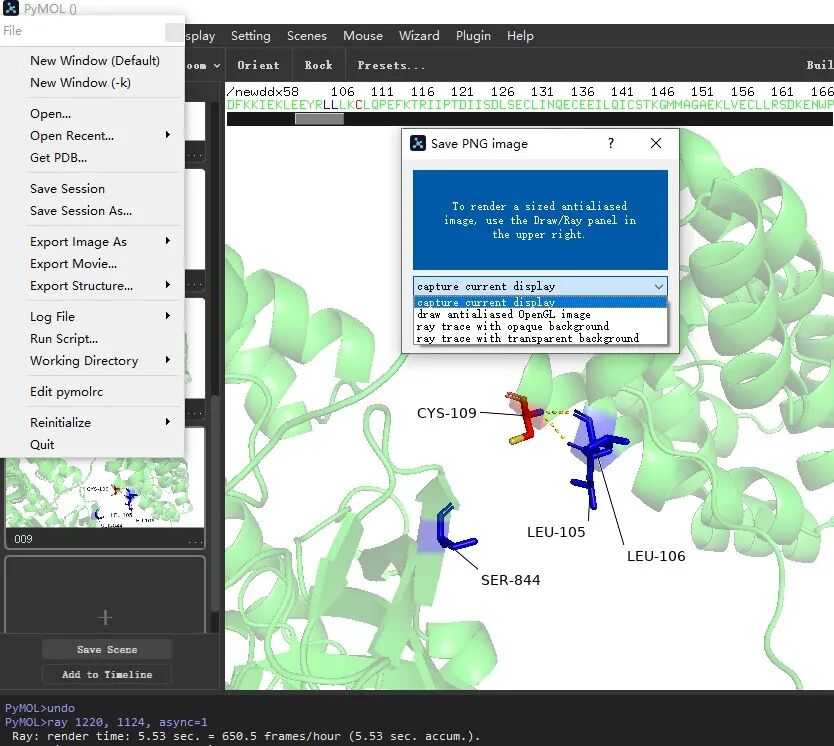
4.10 也可以通过Draw/Ray按钮选择300DPI进行Draw或者Ray模式图片保持到电脑中。

5.最后调整不同对象的颜色,图片排版后如下图所示。