【零代码生信分析】RNA-seq中级分析2-差异基因集【药物】富集分析
【零代码生信分析】RNA-seq中级分析2-差异基因集【药物】富集分析
前言
本期我们将进行差异基因药物富集分析,利用已有的药物处理细胞的转录组数据库。分析我们给的差异基因集与哪些药物处理特定细胞后基因转录数据有明显的交集。由此来提示是哪些类型的药物影响我们差异基因的表达,其实也是对差异基因进行GO、KEGG富集分析的一个补充。例如如果差异基因富集在免疫炎症相关GO、KEGG通路中,进行药物富集分析也富集到抗炎药物或者免疫抑制或者激活药物,便可提示差异基因潜在的特征。
实操过程
步骤1:登陆Webgestalt 网站,选择WebGestalt 2019版本。因为在最新网站版本中去除了药物富集分析选项。
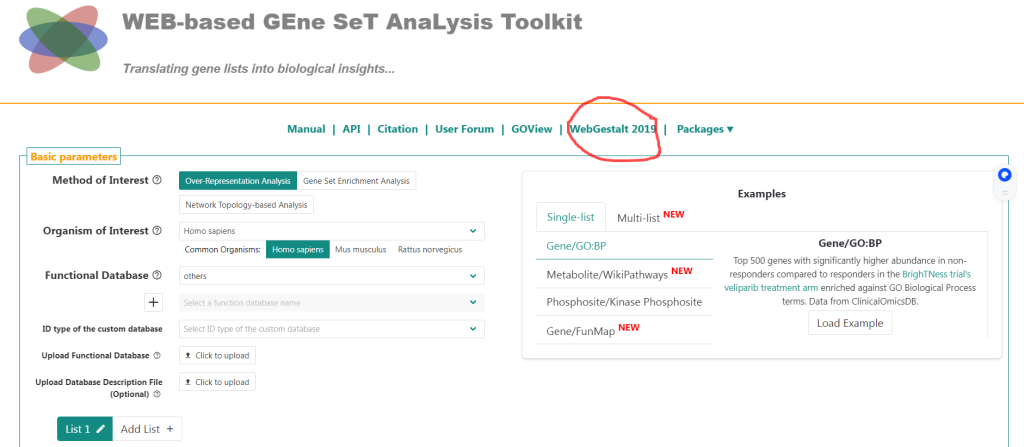
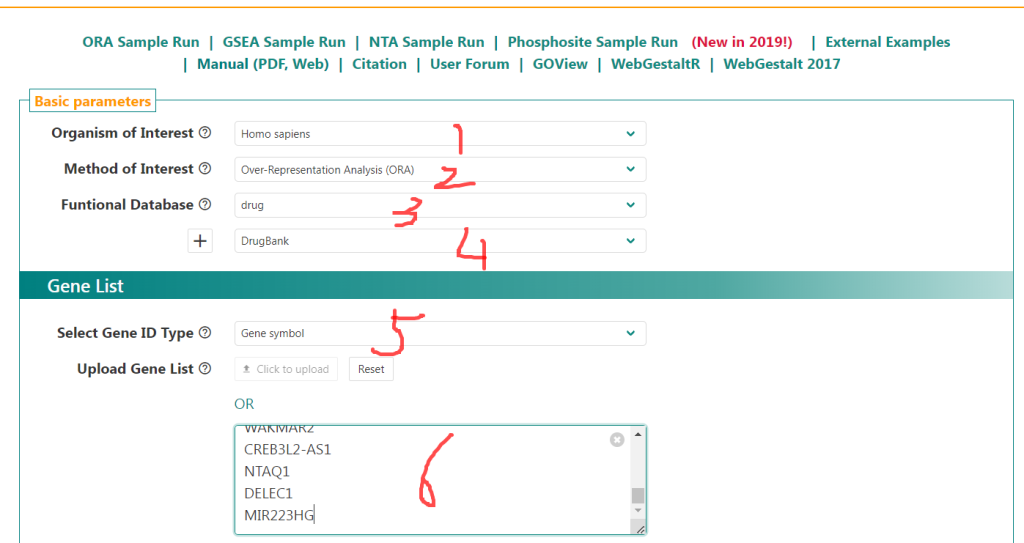
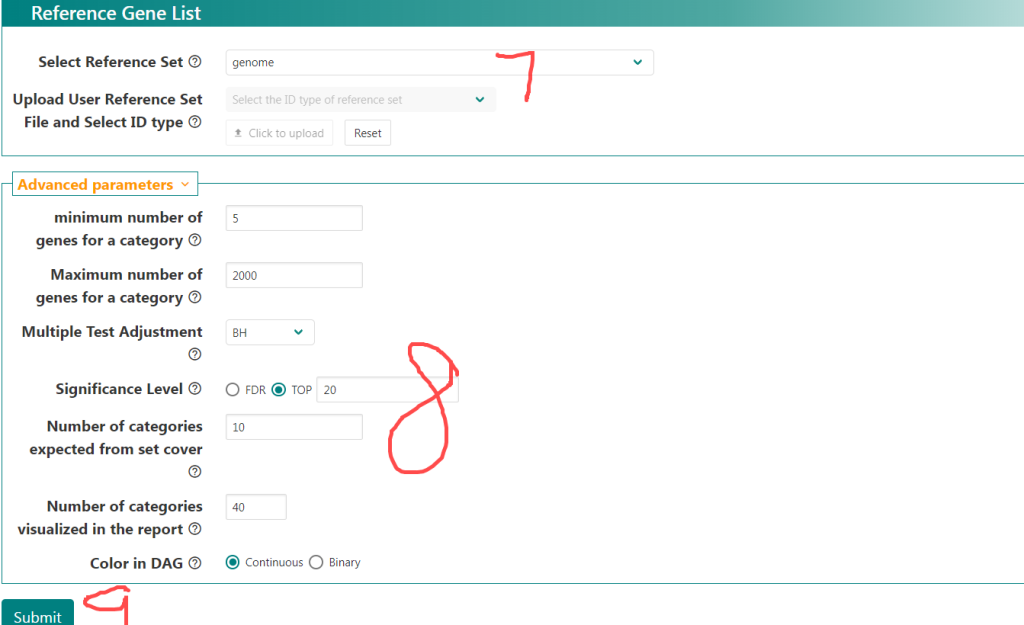
步骤2:在WebGestalt 2019页面中,按照下图选择1,2,3,4进行选择。选项4中有两个数据库,选择其中之一。默认选择Drugbank选项。5是将要输入的基因ID类型,一般选择Gene symbol。6是输入基因名。7,8按照图上进行选择。这里富集结果选择前20的富集药物,9是提交。
步骤3:富集结果:图下图所示。在展示的前20富集药物中,只有一个是FDR<0.05,其他药物都是FDR>0.05.
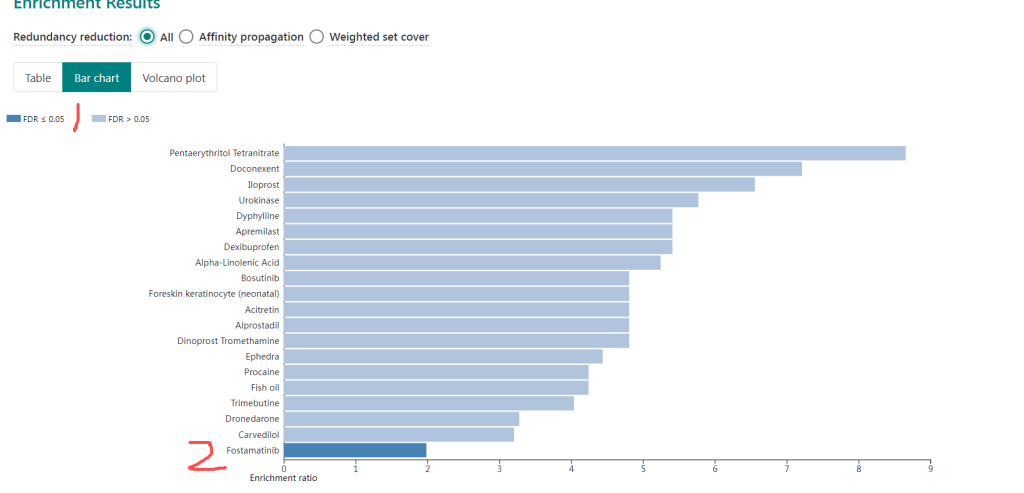
步骤4.我们可以Redundancy reduction选项,如下图选择了Weighted set cover来减少类似基因富集的药物。
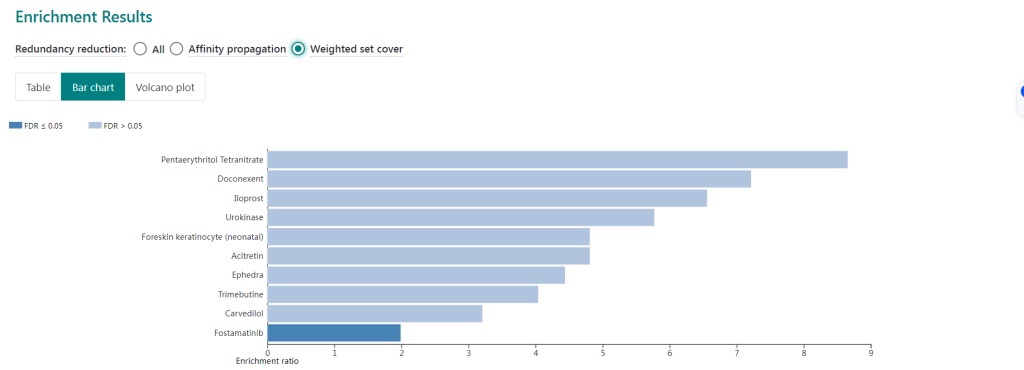
步骤5.点击页面Result Download进行富集结果下载。
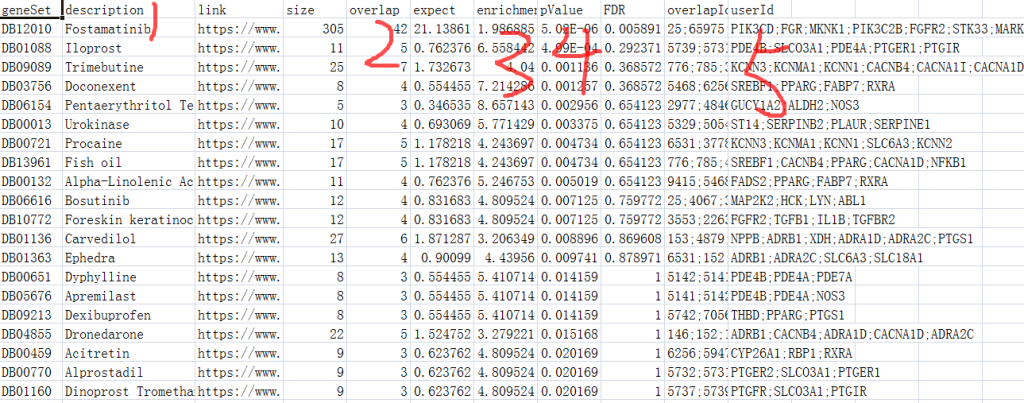
步骤6.在下载的压缩文件中有一个是富集结果文件。通常排在第2或者第3位。下图标记1-5是比较重要的信息。后续的作图中我们将选择1(富集的药物),5(userID基因名)进行下游绘图。
步骤7:在微生信 网页进行可视化绘图。选择GO-pathway 图中弦图进行绘制
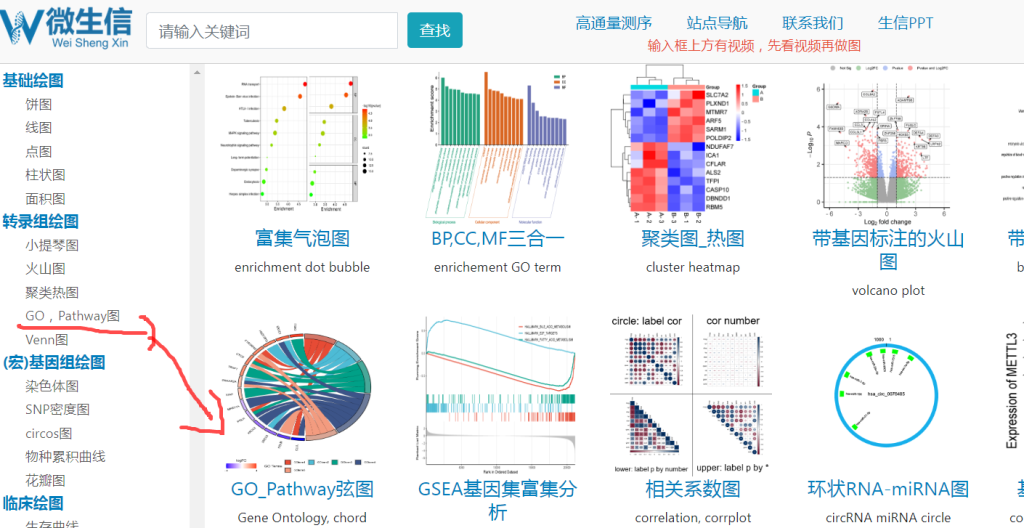
步骤8:先用示例2数据进行载入,点击提交,便可看到示例2数据绘制的图。
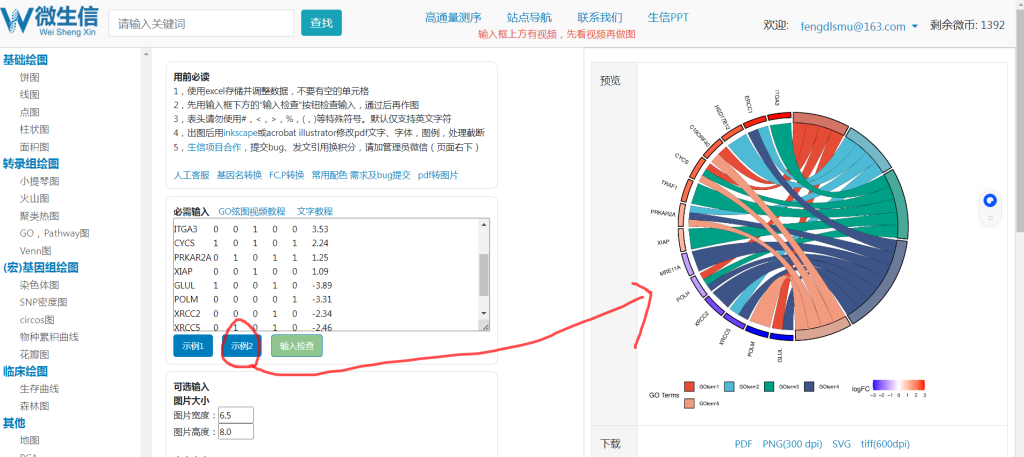
步骤9:理解数据结果。示例2中的数据,第一列是基因名称,第2到倒数第2列是GO term名称(我们数据中是药物名称),最后一列是logFC值就是RNA-seq差异基因的logFC值。首先将差异基因富集结果中的药物名称与对应到的差异基因列复制到新的单元格中。
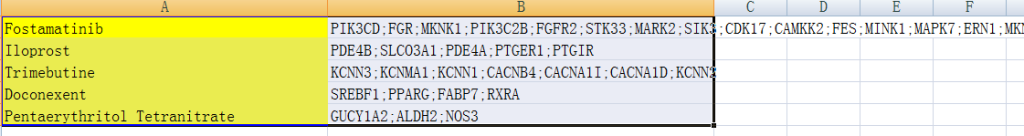
步骤10:然后分别将我们的数据与示例2中的数据分别直接粘贴到百度AI助手对话框中(也可以用其他AI对话),要求按照示例2的格式进行整理。
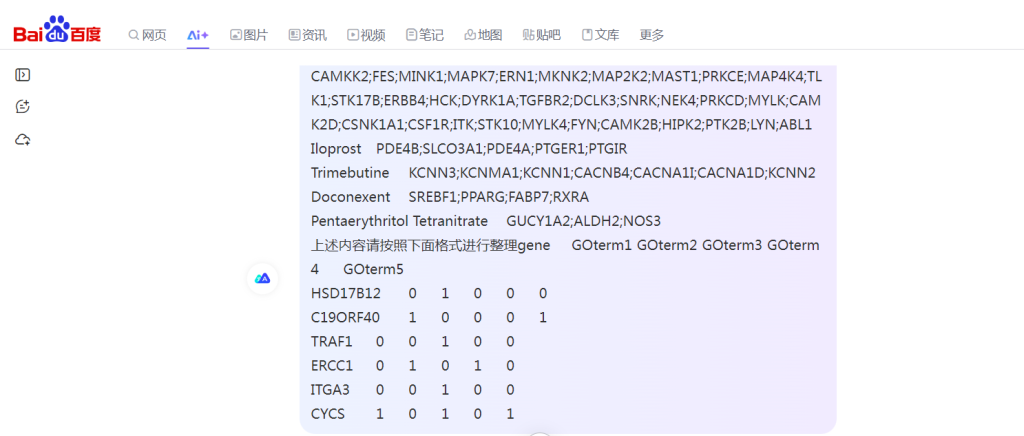
步骤11:如果没有按照要求进行调整。则多次指出回答错误的地方,给到要正确完成的格式。如下图所示,得到调整后的文件格式,直接复制到电子表格中。
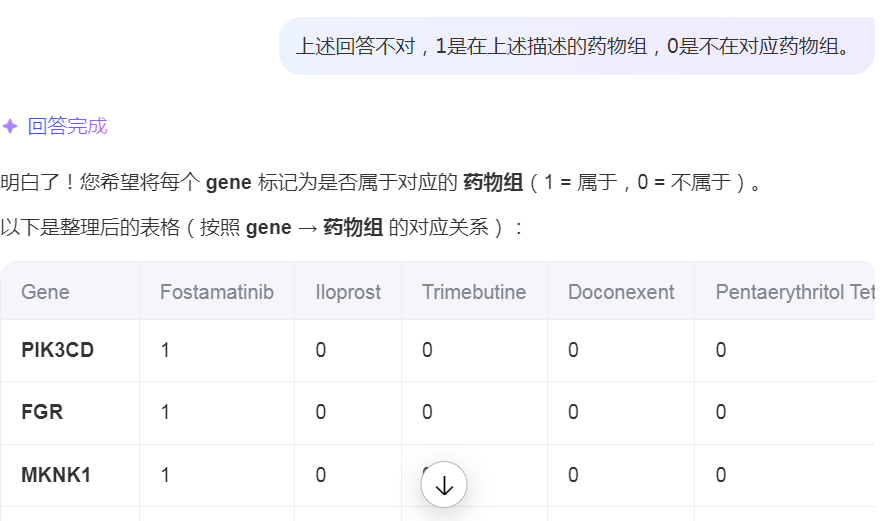
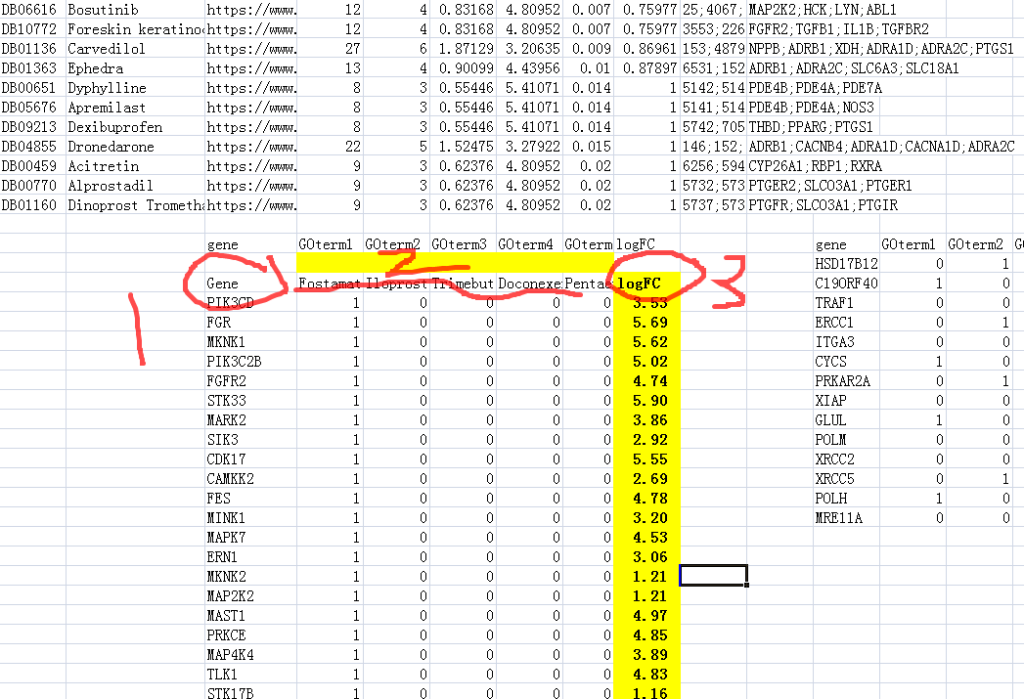
步骤12:然后按照示例2的格式进行1,2所示的粘贴。3所表示的基因logFC也可以通过RNA-seq差异基因表格中找到对应的数值,通过Vlookup函数进行查找。并复制到这个表格中。
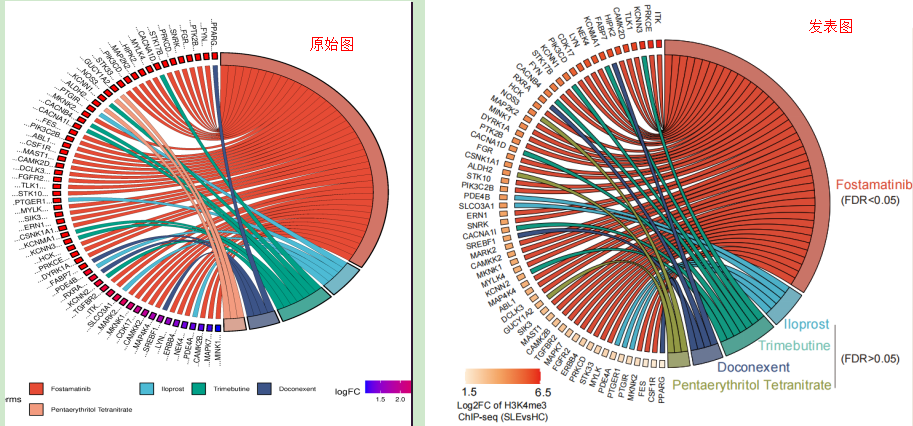
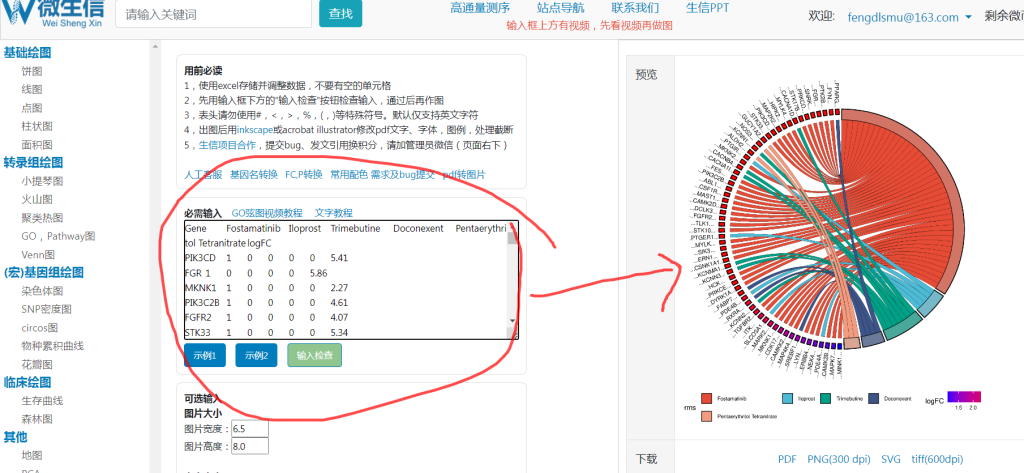
步骤13:将整理好的数据输入到作图页面输入框中,直接点击提交,如图所示药物富集分析的弦图,圆形左侧是基因名称以及logFC热图。右侧是药物所代表的颜色。中间连线是基因与药物的关联性。图片底部是药物名称与颜色对应关系 以及logFC的颜色条。选择PDF或者SVG进行保存。
图片展示: 左图是原始图,右图是本文作者文章发表图,进行了适当颜色与文字布局调整。